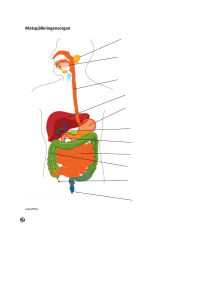
Syfte?
Inblick i vanlig genotypbestämningsmetod
Allozymer har ofta använts som genetisk
markör vid populationsgenetiska studier
Eko-genetisk
långtidsstudie
Studera genetisk variation inom och mellan
populationer – används inom bevarandebiologin
Naturliga och
introducerade
populationer av
öring
Länsstyrelser får ofta ta del av den här typen av
genetiska data. Man bör veta hur dessa är
framtagna och själv ha sett hur data ska tolkas
Tre vattensystem
Vävnadsprover för
genetisk analys
Naturliga populationer i
Trollsvattnen
Två sympatriskt existerande
kryptiska populationer
Otoliter för åldersbestämning
•
Undersöka morfologiska
skillnader
•
Populationernas utbredning i
vattensystemet
•
Statistiska styrka att upptäcka
kryptisk populationsstruktur
• lika frekvens
• stabil sympatri i 20 år
• inga uppenbara fenotypiska
skillnader
1
Bävervattsprojektet
Utplantering 1979
1000 indivder per
stam
Diagnostiskt
locus AGP-2
Identifiering av hybrider
Lillrörtjärnen (66)
Rena stammar och F1 hybrider
identifieras i locus AGP-2
E4 (83)
Tjärn 2 Östra (57)
Stora Bävervattnet 1025)
E3 (54)
Lilla Bävervattnet (760)
E2 (45)
E1 (55)
Djuptjärnen (57)
Stam A
Storvuxen
Kallsjön
Stam B
Småvuxen
Fälpfjälltjärnarna
Haravattnet (588)
Senare generationer svårare att identifiera.
Haraån (42)
Lillrörtjärnen (66)
NEWHYBRIDS
Klassificerar individer till sex grupper.
E4 (83)
Tjärn 2 Östra (57)
Stora Bävervattnet 1025)
F1
E3 (54)
Lilla Bävervattnet (760)
E2 (45)
E1 (55)
Djuptjärnen (57)
Stam A
Stam B
F2h
F1hxA
F1h
F2
Haravattnet (588)
Haraån (42)
Sammanfattade resultat
• Stam A och B har etablerat sig på olika lokaler i
sjösystemet i olika utsträckning.
• Hybrider går inte att identifiera och klassificera i
flera generationer med nuvarande markörer och
testade program
NewHybrids
Structure
GeneClass
• Stammarna kan ha hållits reprodultivt åtskilda i viss
utsträckning.
Proteinelektrofores (allozymer)
Olika “varianter” av samma enzym (protein) - uttrycks på basis av gener
i cellkärnans DNA
Olika alleler ger upphov till olika allozymer, vilka vanligtvis har olika
elektrisk laddning och därför kan särskiljas genom elektrofores
Bandmönster i en gel (med spänning) kan tolkas genetiskt:
antal alleler i detta locus (oftast två)
• homozygot – heterozygot
•
Vi studerar alltså inte DNA direkt...
2
Vad händer på lab?
4. Körning av gelelekrtofores
1. Förberedelse av vävnadsprov
2. Gjutning av geler
5. Slicing (uppskärning) av gelen
3. Applicering av enzymextrakt på gel
6. Infärgning och avläsning
ENZYMELEKTROFORES
– ”kom i håg-lista”
Gen = specifik del av en DNA-molekyl
Locus = fysiska positionen av genen på DNA-strängen, tex AGP-locus
Allel = varianter av en gen
Protein
Enzym = protein som katalyserar reaktioner, tex AGP
Aminosyrakedjor = ”bygger upp” enzym
Allozym = en viss slags enzym, representerar olika alleler av samma gen
Är det någon genetisk skillnad
mellan öringar från de tre olika lokalerna?
MONOMERT ENZYM – består av 1 aminosyrasekvens
(t.ex. PEPLT)
DIMERT ENZYM – består av 2 aminosyrasekvenser
(t.ex. AGP)
- Den ena homozygoten har bara den ena varianten av enzymet i cellkärnan
- Den andra homozygoten har bara den andra varianten av enzymet i cellkärnan
- Heterozygoten har båda varianterna av enzym i cellkärnan får därför två band
Två genprodukter måste sättas ihop för att bilda ett fungerande enzym.
Om enzymet består av två likadana genprodukter homodimer
Om enzymet består av två olika genprodukter heterodimer
Ex. med 2 alleler
- Den ena homozygoten bildar bara ”dubletter” av den ena varianten
- Den andra homozygoten bildar bara ”dubletter” av den andra varianten
- Heterozygoten har båda varianterna i cellkärnan och bildar därför alla slags ”dubletter”
Hos en heterozygot finns homo- respektive heterodimererna i mängdförhållandet 1:2:1
A1A1
A1A2
A2A2
Ex. med 2 alleler (kan också finnas med fler alleler)
Ex. med 3 alleler
A1A1
A1A2
A2A2
A1A3
A2A3
A3A3
A1A1
A1A2
A2A2
3
kodande
TETRAMERT ENZYM – består av 4 aminosyrasekvenser
(LDH-1)
icke-kodande
Hos en heterozygot finns de olika enzymvarianterna i mängdförhållandet 1:4:6:4:1
Exoner
Ex. med 2 alleler (kan också finnas med fler alleler)
A1A1
A1A2
Introner
Regulatoriska
regioner
rDNA
histoner
A2A2
Kärn-DNA
Mini- och
mikrosatellit DNA
icke-repetetivt
(ex. ...GCATACTAGGA...)
repetetivt
(ex. ATT ATT ATT ATT ATT...)
Annat repetitivt DNA
mtDNA
AGG - mikrosatellit
Separation och visualisering (sekvensator)
F-statistik kom-ihåg lista
Mikrosatelliter - tolkning
Observerad heterozygositet
Förväntad heterozygositet enligt Hardy
Weinberg
FST Genetisk differentiering mellan
populationer, 0=ingen differentiering,
1=fixering för olika alleler.
FIT Avvikelser från Hardy-Weinberg i
totalpopulationen
FIS Avvikelser från Hardy-Weinberg i
subpopulationerna
Ho
He
Infärgning av primers - visualisering
LOCUS 1
Ind 1
Heterozygot
93/99
Ind 2
Homozygot
103/103
Ind 3
Heterozygot
99/103
4
Lokal
Genepop on the Web
Locus
Individ
Polymorfigrad
Heterozygositet
Skillnader mellan
lokaler (FST)
Hardy-Weinberg
Populationsgenetiska
analyser
Ladda upp filer med
genotypdata
Allozymer
Microsatelliter
Resultat:
Scrolla ner!
Klicka i önskade alternativ
Svar som HTML
Ladda upp fil med genotyper
Den
intressanta
informationen
finns oftast
längst ner i
filen
5