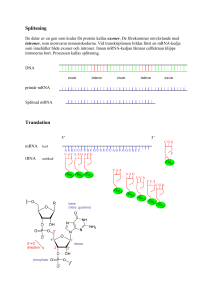
Bruksanvisning för CRC RAScan™ Combination Kit
advertisement

1 Tillverkare Bruksanvisning för CRC RAScan™ Combination Kit A SURVEYOR® Scan KRAS och NRAS Kit Exons 2, 3 & 4 CE IVD med DHPLC-system Läs dessa anvisningar noga innan du använder produkten. Spara denna bruksanvisning för framtida bruk. Denna sida har avsiktligt lämnats tom Innehållsförteckning 1 Tillverkare ..................................................................................................................... 3 2 CRC RAScan™ Combination Kit ................................................................................. 3 2.1 Avsedd användning ............................................................................................................. 3 2.2 Indikationer ......................................................................................................................... 3 3 Principer för CRC RAScan KRAS och NRAS mutationsdetektionsanalys .............. 4 3.1 KRAS och NRAS .................................................................................................................... 4 3.2 Analys av patientprover med SURVEYOR Scan Kit ............................................................... 4 3.3 SURVEYOR Nuclease ............................................................................................................ 5 4 Härledning av kittets kontrollsubstanser ................................................................... 6 5 Komponenter ................................................................................................................ 7 5.1 Låda med SURVEYOR Scan KRAS Kit Exons 2, 3 & 4 (h 710107) ........................................ 7 5.2 Låda med SURVEYOR Scan NRAS Kit Exons 2, 3 & 4 (h 710400) ........................................ 8 5.3 Antal prover som kan testas med ett kit .............................................................................. 8 5.4 DNA‐sekvensering ............................................................................................................... 9 6 Ytterligare nödvändig utrustning och reagenser ....................................................... 9 7 Reagensberedning ....................................................................................................... 9 8 Förvaring och hållbarhet ........................................................................................... 10 9 Varningar och försiktighetsåtgärder......................................................................... 10 10. Förberedande provinsamling, hantering och förvaring ....................................... 10 11 Analysprocess .......................................................................................................... 11 11.1 Somatisk mutationsdetektion med SURVEYOR Scan‐kit ‐ En översikt .............................. 11 12 Stegvisa anvisningar ............................................................................................... 12 12.1 INLEDANDE installation/kalibrering av DHPLC ............................................................. 12 12.2 Att tänka på före KRAS‐ och NRAS‐provanalys ................................................................. 12 12.3 Att tänka på avseende templat ........................................................................................ 12 12.4 Att tänka på avseende arbetsflödet ................................................................................. 12 12.5 Amplifieringsprotokoll ..................................................................................................... 15 12.6. Termocykelprogram för amplifieringsprotokoll .............................................................. 17 12.7 Kvalitetskontroll av PCR‐produkter .................................................................................. 17 12.8 SURVEYOR Nuclease‐nedbrytning ................................................................................... 18 13 Kontrollparametrar ................................................................................................... 20 13.1 Kvalitetskontroll av CRC RAScan Combination Kit ............................................................ 20 13.2 Användning av kontrollplasmid‐DNA ............................................................................... 20 14 Tolkning av resultat ................................................................................................. 21 14.1 Analys av KRAS och NRAS Exons 2, 3 och 4 med SURVEYOR Nuclease ............................. 21 14.2 Granskning av SURVEYOR Scan‐resultat .......................................................................... 21 14.3 Exempel på resultat ......................................................................................................... 22 15 Prestationsegenskaper ............................................................................................ 29 15.1 LOD för mutationer med SURVEYOR Scan‐kit .................................................................. 29 15.2 Sekvensbekräftelse .......................................................................................................... 29 15.3 Analysprocessens begränsningar ..................................................................................... 30 Bilaga A.......................................................................................................................... 31 A.1 Översikt av platta för SURVEYOR Scan kit .......................................................................... 31 A.2 Kontrollernas DNA‐stämplar ............................................................................................. 31 A.3 Systemkrav för denaturering med HPLC (DHPLC) .............................................................. 32 A.4 Laboratorieinstallation för PCR‐analyser ........................................................................... 33 A.5 Litteraturlista .................................................................................................................... 35 Bilaga B.......................................................................................................................... 36 Felsökningsguide ..................................................................................................................... 36 Beställningsinformation ............................................................................................... 43 Kontaktuppgifter ........................................................................................................... 43 Översättning Ansvarsfriskrivning ................................................................................ 43 Varumärken & upphovsrätt .......................................................................................... 44 1 Tillverkare 1 Tillverkare M Tillverkare EGrepresentant P Transgenomic, Inc. 12325 Emmet Street, Omaha, NE 68164, USA Tel +1-402-452-5400 Transgenomic Limited 40 Watt Road, Hillington Park, Glasgow G52 4RY, UK Tel +44-141-892-8800 2 CRC RAScan™ Combination Kit 2.1 Avsedd användning Endast för professionellt bruk. Transgenomics CRC RAScan Combination Kit är en in vitro diagnostisk analys som upptäcker somatiska mutationer i Exon 2, 3 och 4 i KRAS- och NRASgenerna. Dessa mutationer påvisas av SURVEYOR Nuclease-klyvningstoppar och inkluderar de mutationer som har känd potential för klinisk signifikans. Detta kit är utformat för användning i ett kliniskt diagnostiskt laboratorium av utbildad personal som testar DNA som utvunnits ur formalinfixerad paraffininbäddad vävnad. Detta kit har referensen katalognummer 710077. Denna bruksanvisning kan hämtas på http://world.transgenomic.com/files/literature/482427-SV.pdf. 2.2 Indikationer Kliniker kan använda CRC RAScan Combination Kit med DHPLC-system för att underlätta fastställande om patienters kolorektala cancer kanske inte kommer att svara på behandling med EGFR-hämmare (epidermal growth factor receptor) såsom panitumumab. CRC RAScan Combination Kit får inte användas för diagnos av kolorektal cancer eller någon annan form av cancer. CRC RAScan Combination Kit är en analys som detekterar förekomsten av potentiella somatiska mutationer i Exons 2, 3 och 4 hos KRAS-genen, men som inte bekräftar mutationens sekvensidentitet. För att bestämma exakt vilken mutation som detekteras krävs ytterligare analys, t.ex. DNA-sekvensering. Analysresultaten med detta kit kommer visserligen att visa patientens mutationsstatus, men andra kliniska faktorer bör beaktas. Resultat som erhålls mned CRC RAScan Combination Kitbör inte vara den enda metod som används för att besluta om behandling av patienter med kolorektal cancer. Det är viktigt att notera att användning av DHPLC för att identifiera prover som är positiva för en KRAS- eller NRAS-mutation med detta kit endast bör utgöra en vägledning och att alla mutationer måste bekräftas genom ytterligare analys, t.ex. DNA-sekvensering. Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 3 3 Principer för CRC RAScan KRAS och NRAS mutationsdetektionsanalys 3 Principer för CRC RAScan KRAS och NRAS mutationsdetektionsanalys 3.1 KRAS och NRAS Riktade terapier för receptorn EGFR (epidermal growth factor receptor) har visat sig vara effektiva mot kolorektalcancer. Forskning har visat att cirka 40 % av alla kolorektala tumörer bär på somatiska KRAS-genmutationer och kliniska studier visade att mutationer i KRAS Exon 2 (kodon 12 och 13) förutspår bristande svar på EGFR-hämmande behandling. Nyligen genomförda undersökande studier har visat att patientpopulationen kan sållas ytterligare eftersom inte heller patienter vars mCRC-tumörer hade ytterligare en mutation i KRAS Exons 3 och 4 eller NRAS Exons 2, 3 och 4 tenderade att svara på EGFR-hämmande behandlning1-7. Detta kit är utformat för användning vid diagnostisk analys av somatiska mutationer i KRAS och NRAS Exons 2, 3 och 4. CRC RAScan Combination Kit är en analys för detektion av alla sekvens- samt infogade/saknade avvikelser i Exons 2, 3 och 4 hos KRAS- och NRAS-generna. Mutationer i KRAS och NRASkodon 12, 13, 59, 61, 117 och 146 har förknippats med bristande effekt hos panitumumab. Positive Controls tillhandahålls i detta kit för mutationer i kodon 12, 61, 117 och 146. Detta kit använder Transgenomics patentskyddade SURVEYOR Nuclease-teknik och ger, i samverkan med DHPLC System teknik, en enkel och känslig detektion av potentiella mutationer. Det kan detektera en blandning av 2-5 % muterat DNA i en bakgrund av ej muterat DNA. Kontrollstudier har påvisat extremt hög överensstämmelse i väldefinierade prover med kolorektalcancer. Användning av CRC RAScan Combination Kit kommer att minska användarens sekvenseringbörda och även hjälpa till vid sekvenseringsbestämning då automatisk sekvenseringsprogramvara inte klarar att klargöra förekomst av låg mutationsnivå. På grund av att denna analys har en hög känslighet i förhållande till Sanger-sekvensering, rekommenderas att laboratoriets installation är optimerad för att undvika korskontamination av prover eller kontroller. Ett exempel på ett idealiskt laboratoriearrangemang finns i Bilaga A.4 Laboratorieinstallation för PCR-analyser. 3.2 Analys av patientprover med SURVEYOR Scan Kit CRC RAScan Combination Kit får endast användas i samband med det arbetsflöde som visas nedan. Arbetsflöde Patientprov Test KRAS och NRAS Exons 2, 3 & 4 Rapportera resultat Figur 1 CRC RAScan Combination Kit arbetsflöde för screening av KRAS och NRAS För förslag på hur plattor med 96 brunnar kan arrangeras för analys av alla KRAS och NRAS Exons 2-4, se Bilaga A.1 Översikt av platta för CRC RAScan Combination Kit. Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 4 3 Principer för CRC RAScan Scan KRAS och NRAS mutationsdetektionsanalys 3.3 SURVEYOR Nuclease Transgenomics SURVEYOR Nuclease är en avvikelsespecifik (mismatch-specific) växtbaserad DNA-endonukleas som kan söka efter kända och okända mutationer och polymorfismer i heteroduplex DNA8. Enzymet klyver DNA med hög precision på ställen med avvikelser i basen och andra förändringar. Denna DNA-endonukleas klipper av båda strängarna på en DNA-heteroduplex på 3’-sidan av det avvikande stället. Tillfogade/saknade och bassubstituerande avvikelser identifieras men klyvningens effektivitet varierar beroende på sekvensen på avvikelsen. Figur 2. Verkningssätt för SURVEYOR Nuclease. Endonukleasen känner igen en avvikelse och klyvs vid 3’-sidan av det avvikande stället. Detta klyver den dubbla DNA-strängen och lämnar kvar ett 3’-överhäng av ett enda baspar. SURVEYOR Nuclease har använts i många olika sammanhang för att korrekt detektera en rad mutationer och polymorfismer i gener. I synnerhet har SURVEYOR Nuclease använts för att bekräfta förekomsten av kända mutationer i ett antal gener som associeras med njurcancer, lungcancer, cancer i huvud och nacke, leukemi, endometriecancer och för att förutsäga resultatet av strålbehandling9,10,11. CRC RAScan Combination Kit har utformats för att klyva avvikelser i KRAS och NRAS Exons 2, 3 och 4 för efterföljande analys av DHPLC. Obs! Om ett prov är 100% muterat DNA, kan inga heterduplex bildas och provet kommer att visas som "Wild-Type". Biopsiprover från tumörer kommer dock att innehålla Wild-Typeceller på grund av tumörheterogenitet och/eller kontamination av normala vävnader; notera att prover med 5% och 95% muterat DNA kommer att ha identiska kromatogram. Obs! Endast den DNA Polymerase som tillhandahålls med detta kit får användas för analysens PCR-komponent. Obs! Följ specifika anvisningar i handboken till ditt DHPLC-system. För att framgångsrikt använda detta kit rekommenderar vi att du läser igenom den här handboken och noggrant följer alla instruktioner och riktlinjer. Nya användare bör utföra de kontrollexperiment som beskrivs i avsnittet Användning av kontroll-DNA-plasmider. Om du har ytterligare frågor eller behöver hjälp kan du kontakta (888) 233-9283 (endast Nordamerika), +1 (402) 452-5400 eller +44 (0) 141 892 8800 (Europa) och begära "KRAS- och NRAS-support". Alternativt kan du mejla oss på: [email protected] Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 5 4 Härledning av kittets kontrollsubstanser 4 Härledning av kittets kontrollsubstanser De kontrollsubstanser som tillhandahålls i detta kit är plasmida kloner av KRAS och NRAS Exon 2, 3- och 4-sekvenser. Alla kloner har sekvenserats för att verifiera sekvensens överensstämmelse genom jämförelse med NCBI Reference Seuquences: NG_007524.1 (KRAS) och NG_007572 (NRAS). Kontrollerna har en genetisk "stämpel". Se Bilaga A.2 Kontrollernas DNA-stämplar; dessa är variationer av KRAS eller NRAS Wild-Type-sekvenser i en region som inte förväntas ha mutationer och som kan användas för att felsöka provkontamination av Positive Controls. Se Bilaga B - Felsökningsguide, problem 8, för ett exempel på en SURVEYOR Scan-kurva av sådan kontamination. "KRAS Control Wild-Type" konstruerades genom syntes och kloning av KRAS Exons 2, 3 och 4 med användning av NCBI Reference Sequence NG_007524.1. "KRAS Positive Control Exon 2" konstruerades genom syntes av KRAS Exon 2 med användning av ovanstående referenssekvens, men innehållande G12D-mutationen. DNAsekvensering bekräftade att den enda förändringen i sekvensen är vid kodon 12 med en GGT>GAT-förändring. Denna klon blandas därefter med KRAS Control Wild-Type-DNA för att skapa en heterozygot blandning. "KRAS Positive Control Exon 3" konstruerades genom syntes av KRAS Exon 3 med användning av ovanstående referens, men innehållande Q61H-mutationen. DNA-sekvensering bekräftade att den enda förändringen i sekvensen är vid kodon 61 med en Q61H, CAA>CACförändring. Denna klon blandas därefter med KRAS Control Wild-Type-DNA för att skapa en heterozygot blandning. "KRAS Positive Control Exon 4A" konstruerades genom syntes av KRAS Exon 4 med användning av ovanstående referens, men innehållande K117N-mutationen. DNA-sekvensering bekräftade att den enda förändringen i sekvensen är vid kodon 117 med en K117N, AAA>AATförändring. Denna klon blandas därefter med KRAS Control Wild-Type-DNA för att skapa en heterozygot blandning. "KRAS Positive Control Exon 4B" konstruerades genom syntes av KRAS Exon 4 med användning av ovanstående referens, men innehållande A146T-mutationen. DNA-sekvensering bekräftade att den enda förändringen i sekvensen är vid kodon 146 med en A146T, GCA>ACAförändring. Denna klon blandas därefter med KRAS Control Wild-Type-DNA för att skapa en heterozygot blandning. "NRAS Control Wild-Type" konstruerades genom syntes och kloning av NRAS Exons 2, 3 och 4 med användning av NCBI Reference Sequence NG_007572. "NRAS Positive Control Exon 2" konstruerades genom syntes av NRAS Exon 2 med användning av ovanstående referens, men innehållande G12D-mutationen. DNA-sekvensering bekräftade att den enda förändringen i sekvensen är vid kodon 12 med en GGT>GAT-förändring. Denna klon blandas sedan med NRAS Control Wild-Type-DNA för att skapa en heterozygot blandning. "NRAS Positive Control Exon 3" konstruerades genom syntes av NRAS Exon 3 med användning av ovanstående referens, men innehållande Q61K-mutationen. DNA-sekvensering bekräftade att den enda förändringen i sekvensen är vid kodon 61 med en Q61K, CAA>AAAförändring. Denna klon blandas sedan med NRAS Control Wild-Type-DNA för att skapa en heterozygot blandning. "NRAS Positive Control Exon 4" konstruerades genom syntes av NRAS Exon 4 med användning av ovanstående referens, men innehållande A146T-mutationen. DNA-sekvensering bekräftade att den enda förändringen i sekvensen är vid kodon 146 med en A146T, GCC>ACCförändring. Denna klon blandas sedan med NRAS Control Wild-Type-DNA för att skapa en heterozygot blandning. Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 6 5 komponenter 5 Komponenter CRC RAScan Combination Kit tillhandahålls i ett ytterhölje som två separata kartonger: (a) en låda med reagenser för analys av KRAS Exons 2, 3 och 4 och (b) en låda med reagenser för analys av NRAS Exons 2, 3 och 4. Tillsammans innehåller lådorna tillräckligt med reagenser frö att utföra det antal analyser som anges i avsnitt 5.3. 5.1 Låda med SURVEYOR Scan KRAS Kit Exons 2, 3 & 4 (h 710107) Lådan med komponenter för analys av KRAS Exons 2, 3 och 4 har två innerkartonger: (1) a SURVEYOR Digestion Components Box med 10 reagensrör och (2) en yttre PCR Components and Controls Tube-Holder som innehåller 20 reagensrör. Katalognummer Komponent Färg på rörets lock Volym för 130 reaktioner Lila Svart Rosa Brunt Rött 3 x 105 µL 2 x 105 µL 2 x 105 µL 2 x 105 µL 250 µL Genomskinligt Genomskinligt Genomskinligt Blått Blått Blått Blått Blått Blått Blått Blått Gult Grönt Grönt Grönt Grönt Orange Orange 2 x 100 µL 1 mL 2 x 500 µL 125 µL 125 µL 90 µL 90 µL 90 µL 90 µL 90 µL 90 µL 120 µL 40 µL 40 µL 40 µL 40 µL 125 µL 125 µL Låda med SURVEYOR nedbrytningskomponenter 710160 710161 708049 708027 708030 SURVEYOR Nuclease W SURVEYOR Enhancer W2 SURVEYOR Enhancer Cofactor 0,15 M MgCl2 Solution Stop Solution Låda med PCR-komponenter och kontroller 703310 703315 703065 710155F 710155R 710157F 710157R 710154F 710154R 710156F 710156R 710131 710135 710136 710137 710138 710153F 710153R DNA Polymerase 10x DNA Polymerase Reaction Buffer dNTPs (10 mM) KRAS Primer: Exon 2 Forward (10 µM) KRAS Primer: Exon 2 Reverse (10 µM) KRAS Primer: Exon 3 Forward (10 µM) KRAS Primer: Exon 3 Reverse (10 µM) KRAS Primer: Exon 4A Forward (10 µM) KRAS Primer: Exon 4A Reverse (10 µM) KRAS Primer: Exon 4B Forward (10 µM) KRAS Primer: Exon 4B Reverse (10 µM) KRAS Control Wild-Type KRAS Positive Control Exon 2 KRAS Positive Control Exon 3 KRAS Positive Control Exon 4A KRAS Positive Control Exon 4B Universal Sequencing Primer 1 (10 µM) Universal Sequencing Primer 2 (10 µM) Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 7 5 komponenter 5.2 Låda med SURVEYOR Scan NRAS Kit Exons 2, 3 & 4 (h 710400) Lådan med komponenter för analys av NRAS Exons 2, 3 och 4 har två innerkartonger: (1) en SURVEYOR låda med nedbrytningskomponenter med 10 reagensrör och (2) en yttre PCRkomponenter och kontrollrörshållare som innehåller 14 reagensrör. Katalognummer Komponent Färg på rörets lock Volym för 100 reaktioner Lila Svart Rosa Brunt Rött Orange Orange 2 x 105 µL 105 µL 105 µL 105 µL 250 µL 2 x 125 µL 2 x 125 µL Rött Rött Genomskinligt Genomskinligt Blått Blått Blått Blått Blått Blått Gult Grönt Grönt Grönt 100 µL 20 µL 1 mL 500 µL 90 µL 90 µL 90 µL 90 µL 90 µL 90 µL 120 µL 40 µL 40 µL 40 µL Låda med SURVEYOR nedbrytningskomponenter 710160 710161 708049 708027 708030 710153F 710153R SURVEYOR Nuclease W SURVEYOR Enhancer W2 SURVEYOR Enhancer Cofactor 0,15 M MgCl2 Solution SURVEYOR Stop Solution Universal Sequencing Primer 1 (10 µM) Universal Sequencing Primer 2 (10 µM) Låda med PCR-komponenter och kontroller 703310 703312 703315 703065 710452F 710452R 710453F 710453R 710454F 710454R 710441 710442 710443 710444 DNA Polymerase (250 U) DNA Polymerase (50 U) DNA Polymerase 10X PCR Buffer dNTPs (10 mM) NRAS Primer Exon 2 Forward (10 µM) NRAS Primer Exon 2 Reverse (10 µM) NRAS Primer Exon 3 Forward (10 µM) NRAS Primer Exon 3 Reverse (10 µM) NRAS Primer Exon 4 Forward (10 µM) NRAS Primer Exon 4 Reverse (10 µM) NRAS Control Wild-Type Mix NRAS Positive Control Exon 2 NRAS Positive Control Exon 3 NRAS Positive Control Exon 4 5.3 Antal prover som kan testas med ett kit CRC RAScan Combination Kit är utformad för att räcka till analys av 230 reaktioner. Det totala antal prover som kan testas med kittet beror på den genomsnittliga satsstorleken som testas vid ett tillfälle eftersom en uppsättning kontrollreaktioner (Wild-Type Controls, Positive Controls och kontroller utan templat) måste inkluderas i varje reaktionsplatta. Tabellen nedan anger vilket antal prover som kan analyseras med ett KRAS- och NRAS-kit beroende på storleken på en genomsnittlig sats. I tabellen beaktas (a) de 21 kontrollerna (7x Wild-Type, 7x Positive Controls, 7x kontroller utan templat) som krävs för varje körning och (b) gränsen på 230 reaktioner per kit. Obs! Om flera plattor körs, måste en uppsättning med de 21 kontroller som anges ovan köras på varje platta. Därför kräver två plattor sammanlagt 42 kontrollreaktioner, 3 plattor kräver 63 kontrollreaktioner, o.s.v. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 8 5 komponenter Med en större provsatsstorlek ökar antalet prover som kan testas med ett kit, vilket minskar den genomsnittliga reagenskostnaden per prov. Nedanstående tabell ger en vägledning till hur många prover som kan testas med kiterna. Totalt antal provsatskörningar per kit Totalt antal prover som testas per kit Provsatsstorlek Antal kontrollreaktioner + antal provamplikoner Antal reaktioner som krävs per körning 1 21 + 7 28 8 8 2 21 + 14 35 6 12 3 21 + 21 42 5 15 4 21 + 28 49 4 16 5 21 + 35 56 4 20 5.4 DNA-sekvensering Sekvenseringsprimers (PN 710153F och 710153R) tillhandahålls för användning vid DNAsekvensering av alla testade prover. De PCR-amplikoner som skapas före SURVEYOR Nucleasenedbrytning bör användas för sekvensering. 6 Ytterligare nödvändig utrustning och reagenser Ytterligare komponenter och utrustning som behövs för att använda CRC RAScan Combination Kit med DHPLC innefattar det följande: DHPLC-system, kolumn, buffertar, DNA-storleksmarkör - se Bilaga A.3.1 DHPLC-specifikationer för SURVEYOR Scan-användningar beträffande egenskaper för ett lämpligt DHPLC-system för användning med detta kit Vatten för molekylärbiologi 0,2 mL-PCR-rör, remsor eller platta med 96 brunnar Mikropipetter Pipettspetsar Isbad Vortexblandare Mikrocentrifug Termocykler Agarosgeler och utrustning för agarosgelelektrofores 10 % blekmedel eller liknande rengöringsmedel 7 Reagensberedning Alla reagenser som ingår i detta kit är användningsklara. En del komponenter behöver tinas upp, och/eller blandas i en vortex-blandare eller mikrocentrifug före användningen. Se mer information under Analysproceduren, nedan. Reagenser behöver kombineras för att producera Master Mixblandningar och reaktionsblandningar. En fullständig beskrivning ges under Analysprocedur nedan. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 9 8 Förvaring och hållbarhet 8 Förvaring och hållbarhet Detta kit skall förvaras i mellan–18 ºC och -25 °C i en frys med konstant temperatur tills dess det används. Observera utgångsdatumet på varje kit du erhåller. Använd inte kittet efter passerat utgångsdatum. SURVEYOR Nuclease-blandningen som tillreds i Steg 7 av SURVEYOR Nuclease-nedbrytning ska användas omedelbart eftersom SURVEYOR Nuclease W avaktiveras med tiden när det förekommer tillsammans med övriga komponenter i SURVEYOR Nuclease-reaktionsblandning. 9 Varningar och försiktighetsåtgärder Ingen av reagenserna i detta kit utgör en hälsorisk i de kvantiteter som förekommer. Transgenomics dokumentnr. MSD-710107 kan hämtas från http://world.transgenomic.com/files/literature/710077-SV.pdf Det finns inga ämnen i detta kit med animaliskt eller humant ursprung som innebär en infektionsrisk. Detta kit skall bara användas av personer som har utbildats i korrekta laboratorietekniker. Använd alltid lämplig laboratorierock, engångshandskar och ögonskydd vid arbete med komponenterna i detta kit. Efter användningen ska kittets komponenter kasseras som sjukvårdsavfall och enligt lokala lagar och föreskrifter. Alikvoter av reagens som pipetterats från rören i detta kit är endast avsedda för engångsbruk. Komponenterna i detta kit har validerats som fortfarande stabila efter 25 upptiningscykler. Använd inte detta kit om antalet upptiningscykler har överskridits. 10. Förberedande provinsamling, hantering och förvaring CRC RAScan Combination Kit har validerats för användning med DNA som utvunnits från formalinfixerade paraffininbäddade (FFPE) kolorektala cancertumörprover. För optimal DNAextraktion ska vävnaden fixeras i formalin i 14-24 timmar innan den bäddas in i paraffin. Tumörbiopsier är en heterogen blandning av tumörceller och icke-tumörceller. Vidare är själva tumörcellen en heterogen blandning av tumörceller med mutationer och tumörceller utan mutationer. Eftersom dessa somatiska mutationer kanske inte är jämnt fördelade i tumören, kan den analysresultaten från olika sektioner i samma tumör skilja sig åt. För att öka möjligheten att upptäcka en mutation, bör DNA från vävnadens tumörregion isoleras genom att endast tumörområdet skrpas från objektglaset med en ny, steril skalpell för varje nytt objektglass. För framgångsrik användning av detta kit bör den extraherade DNA:n uppfylla kriterierna i Att tänka på avseende templat. OBS! Extraherade DNA-prover som inte används för omedelbar analys ska förvaras vid -20 ºC till -80 ºC. Bruksanvisning för CRC RAScan Kit med DHPLC-system Page 10 11 Analysprocess 11 Analysprocess 11.1 Somatisk mutationsdetektion med SURVEYOR Scan-kit - En översikt Detektion och bekräftelse av mutationer med SURVEYOR Nuclease omfattar tre steg: Steg 1 - Förbered PCR-amplikoner från muterat (test) och normalt (referens) DNA, och fortsätt efter den sista PCR-amplifieringscykeln med att smälta alla dubbla strängar och därefter långsamt låta dem svalna för optimal uppkomst av hetero- och homoduplexer (heteroduplexer uppstår när en sträng i en Wild-Type-sekvens härdas med en sträng av en muterad sekvens). Steg 2 – Behandla en del av den härdade heteroduplex/homoduplex -blandningen med SURVEYOR Nuclease. SURVEYOR Nuclease skär av båda strängarna av heteroduplex DNA så att DNA-fragment bildas. Control Wild-Type-DNA utgör, när den behandlas på liknande sätt, en bakgrundskontroll. Steg 3 - Analysera DNA-fragmenten på ett DHPLC-system. Formeringen av nya kluvna produkter, till följd av förekomsten av en eller flera avvikelser, påvisas genom förekomsten av ytterligare kromatografiska toppar. Migrationstiderna för de kluvna produkterna anger storleken på fragmenten och därmed avvikelsens eller avvikelsernas placering. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 11 12 Stegvisa anvisningar 12 Stegvisa anvisningar 12.1 INLEDANDE installation/kalibrering av DHPLC När SURVEYOR Nuclease-plattan ställs in för analys av DHPLC, se Bilaga A.3 Systemkrav för denaturering med HPLC (DHPLC). 12.2 Att tänka på före KRAS- och NRAS-provanalys Innan prover körs på ett DHPLC-system måste en lämplig DNA-storleksmarkör köras för att tillförsäkra att systemet fungerar korrekt. Laboratoriepersonal som använder instrumentet bör kontrollera DNA-storleksmarkörens kvalitet innan analysen startas. 12.3 Att tänka på avseende templat 1. För FFPE isolerad DNA-templat, använd normala laboratorierutiner för att bedöma kvalitet och kvantitet på extraherad DNA för att se till att det finns tillräckligt templat för PCR. 2. 260/280 absorptionsförhållandet skall vara över1,80 3. För att underlätta PCR-inställningen bör den effektiva koncentrationen av templat vara 12,5 ng/µL. Späd ut DNA-förlagorna med graderat molekylärbiologiskt vatten om så behövs. 12.4 Att tänka på avseende arbetsflödet Kittet är avsett att möjliggöra analys av 230 reaktioner. Mindre provsatser kan köras, men kitkontroller och en "kontroll utan templat" måste inkluderas med varje provsats. Det finns tillräckligt med kontrollsubstans i kittet för 230 reaktioner av alla provomgångar av alla olika storlekskombinationer. I allmänhet bör bearbetning av alla prov utföras från början till slut enligt beskrivningen i den här bruksanvisningen. Om bearbetningen av ett prov stoppas innan alla steg är klara, ska DNA förvaras i -20°C vid angivna punkter. Man bör dock undvika att utsätta ett fruset prov för upprepad upptining och förvaring (-18 till -25 °C) av PCR-amplifierad DNA eller SURVEYOR Nuclease nedbrytningsprodukter under längre perioder (>1 vecka). Bruksanvisning för CRC RAScan Kit med DHPLC-system Page 12 12 Stegvisa anvisningar Analys av prover bör följa nedan illustrerade arbetsflöde: Skrapa vävnad DNAisolering och PCR 1 2 Gelelektrofores 3 Ange Robust/Svag PCR 4 5 SURVEYOR Scan Misslyckades SURVEYOR Nuclease och DHPLC Analysis SURVEYOR Scan Positiv SURVEYOR Scan Negativ 7 6 NVD Sekvensbekräftelse 8 Sekvenskvalitet Misslyckades Sekvenskvalit et godkänd 9 Mutationer i KRAS eller NRAS kodoner G12, G13, A59, Q61, K117 eller A146 NVD Figur 3: Arbetsflöde för analys med CRC RAScan Combination Kit Bruksanvisning för CRC RAScan Kit med DHPLC-system Page 13 12 Stegvisa anvisningar Anmärkningar till figur 3 1. Isolera DNA från FFPE med vedertagen laboratorieteknik. 2. Utför PCR och kontrollera DNA-kvaliteten med gelelektrofores. 3. Registrera om PCR-bandet är robust (≥20 ng/µL) eller svagt (<20 ng/µL). Om det finns flera PCR-band ska ny genomisk DNA beredas från FFPE. 4. Gå vidare till SURVEYOR Nuclease-behandling och DHPLC systemanalys med prover som angetts som antingen robust PCR eller svag PCR efter PCR-amplifiering. Observera ett det vid en svag PCR-bestämning kan finnas otillräcklig DNA för att få meningsfulla resultat genom DHPLC, men det kan finnas tillräckligt med DNA för sekvensering. 5. Se Bilaga B – Felsökningsguide för exempel på misslyckade SURVEYOR Scanresultat. Alla prover med ett misslyckat SURVEYOR Scan-resultat bör köras om antingen genom att: a. upprepa PCR om det finns tillräckligt med genomisk DNA kvar b. extrahera DNA från FFPE på nytt; detta bör vara sekundärt val eftersom det ofta inte är önskvärt att skära ytterligare ett FFPE-block. c. om en annan sektion eller block används måste hela analysen göras om eftersom avvikelser i nedbrytningen kan bero på tumörheterogenicitet. 6. Om det inte finns några synliga klyvningstoppar ska ett negativt SURVEYOR Scanresultat registreras, d.v.s. NVD = No Variant Detected (ingen avvikelse detekterad). 7. Om ett prov visar SURVEYOR Nuclease klyvningsprodukter (matchar inte relevant WildType-Control) ska dessa skickas till sekvenseringsbekräftelse. 8. Om sekvensbekräftelseanalysen inte godkänns, ska man antingen: a. upprepa PCR om det finns tillräckligt med genomiskt DNA kvar b. extrahera DNA från FFPE på nytt; detta bör vara sekundärt val eftersom det ofta inte är önskvärt att skära ytterligare ett FFPE-block. 9. Om sekvensbekräftelseanalysen är godkänd bör resultaten visa antingen: a. Wild-Type-sekvens, d.v.s. ingen avvikelse detekterad (NVD -No Variant Detected); eller b. Avvikelse detekterad. Om sekvensbekräftelsen visar någon basförändring som leder till aminosyraförändring vid: i. kodon 12 eller 13 ii. kodon 59 eller 61 iii. kodon 117; eller iv. kodon 146 bedöm som positiv för KRAS- eller NRAS-mutation. Obs! Det är möjligt att ha en positiv SURVEYOR Scan-bestämning som också ger resultatet "Ingen avvikelse detekterad" vid ett sekvensbekräftelsetest. Detektionsnivån (LOD) för SURVEYOR Nuclease på ett DHPLC-system är så låg som 2% mutation i 98% Wild-Type-DNA för vissa mutationer, medan LOD för sekvensering är cirka 10-25% mutation i 90-75% Wild-TypeDNA. OBS! om ett prov är 100% muterat DNA, kan inga heteroduplex bildas och provet kommer att visas som "Wild-Type". Tumörbiopsiprover kommer emellertid att innehålla Wild-Type-celler på grund av tumörheterogenitet och/eller kontaminerande normal vävnad. Obs! Prover med 5% och 95% muterat DNA kommer att ha identitiska kromatogram. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 14 12 Stegvisa anvisningar Obs! Positiva SURVEYOR Scan-resultat kan bero på andra basförändringar än de som har befunnits vara KRAS- eller NRAS-aktiverande. Dessa mutationer är sällsynta och måste bekräftas av en annan metod, t.ex. DAN-sekvensering, för att fastställa om det positiva SURVEYOR Scanresultatet är en KRAS-eller NRAS-aktiverande mutation eller ej. OBS! Den formalinfixationsprocess som används vid beredning av FFPE-tumörbiopsiprover kan orsaka deaminering av cytosin. Denna deaminering omvandlar cytocin till uracil. Polymerasen kommer att "läsa" detta uracil som tymin och införliva en adenin i de kopierade strängarna. Detta kommer sedan att se ut som en mutation där den normala G nu har bytts ut mot A som orsakar en GC- till AT-mutation, vilket är en artefaktmutation på grund av fixationsprocessen och inte en äkta somatisk mutation. Detta förekommer sällan, men om de kopieras tidigt under PCR-cykeln kommer de att "se ut som" mutationer. De upprepas inte vid omanalys. Exempel på potentiella cytosindeamineringsmutationer som skulle kunna beaktas vid beslut av patientvård är för KRAS: - Kodon 12: AGT (G12S) och GAT (G12D) - Kodon 13: AGC (G13S), GAC (G13D) och GGT (G13G, en tyst mutation) - Kodon 146: GCA>ACA (A146T) Exempel på potentiella cytosindeamineringsmutationer som skulle kunna beaktas vid beslut av patientvård är för NRAS: - Kodon 12: GGT> AGT (G12S) eller GAT (G12D) - Kodon 13: GGT> AGT (G13S), GAT (G13D) eller GGC (G13G, en tyst mutation) - Kodon 146: GCC>ACC (A146T) Därför rekommenderas att sådana mutationer bekräftas med duplikatanalys av samma genomiska DNA eller att alla prover omfattas av duplikatanalys från analysens början. 12.5 Amplifieringsprotokoll 1. Transgenomics förblandade dNTP-lösningar (PN 703065 och 705020) tillhandahålls med en koncentrationsgrad av 10 mM total deoxynukleotid (2,5 mM av var och en av de fyra deoxynukleotiderna). 2. KRAS och NRAS Forward and Reverse PCR primers (KRAS PN 710155F/R, 710157F/R, 710154F/R och 710156F/R; NRAS PN 710452F/R, 710453F/R och 710454F/R) tillhandahålls vid 10 µM. 3. Ta ut 10 µM-primrar, 10 mM dNTP:er och DNA Polymerase 10X PCR Buffer (PN 703315) ur frysen och tina på is. 4. Vortexblanda alla komponenter efter upptining (~10 sekunder) så att de blandas noga, centrifugera en kort stund (~10 sekunder) för att tillförsäkra att ingen vätska finns kvar på rörens lock och lägg på is. 5. Förbered Master Mix-blandningarna på is. 6. Använd nedanstående tabell som en vägledning för beredning av en Master Mix för var och en av KRAS Exon 2-, KRAS Exon 3-, KRAS Exon 4A-, KRAS Exon 4B-, NRAS Exon 2-, NRAS Exon 3- och NRAS Exon 4-reaktionerna: Antal reaktioner (7 prover + 3 kontroller): Volymberäkning: Vattenvolym (µL) DNA Polymerase 10X PCR Buffer (µL) dNTP:er (µL) Forward Primer (µL) Reverse Primer (µL) DNA Polymerase (µL) Total volym Master Mix Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system 10 330** 50 40 25 25 10 48,0 Sid. 15 12 Stegvisa anvisningar **OBS! Användaren bör sträva efter att ha minst 25 ng DNA per 50 µL PCR-reaktion. När de extraheras är DNA-koncentrationerna mindre än 12,5 ng/µL, öka volymen extraherat DNA proportionerligt för att tillförsäkra 25 ng per reaktion. Minska vidare volymen vatten i Master Mix-blandningen med samma mängd för att få 50 µL per reaktion. Alla prover som prepareras med dessa Master Mix-blandningar kommer att behöva extraherad DNA som har spätts ut till ungefär samma lägsta koncentrationsgrad. Vi rekommenderar inte att koncentrationer av extraherad DNA på <5 ng/µL används. 7. Beräkna nödvändiga volymer för alla angivna Master Mix-blandningar med användning av ovanstående tabell. Observera att: (a) För Master Mix 1, KRAS Exon 2, krävs tre ytterligare reaktioner per provplatta: KRAS Control Wild-Type, KRAS Positive Control Exon 2 samt KRAS Exon 2 kontroll utan templat (NTC1). (b) För Master Mix 2, KRAS Exon 3, krävs tre ytterligare reaktioner per provplatta: KRAS Control Wild-Type, KRAS Positive Control Exon 3 samt KRAS Exon 3 kontroll utan templat (NTC2). (c) För Master Mix 3, KRAS Exon 4A, krävs tre ytterligare reaktioner per provplatta: KRAS Control Wild-Type, KRAS Positive Control Exon 4A samt KRAS Exon 4A kontroll utan templat (NTC3). (d) För Master Mix 4, KRAS Exon 4B, krävs tre ytterligare reaktioner per provplatta: KRAS Control Wild-Type, KRAS Positive Control Exon 4B samt KRAS Exon 4B kontroll utan templat (NTC4). (e) För Master Mix 5, NRAS Exon 2 kommer ytterligare tre reaktioner att behövas per provplatta för NRAS Control Wild-Type, NRAS Positive Control Exon 2 och NRAS Exon 2 kontroll utan templat (NTC5). (f) För Master Mix 6, NRAS Exon 3 kommer ytterligare tre reaktioner att behövas per provplatta för NRAS Control Wild-Type, NRAS Positive Control Exon 3 och NRAS Exon 3 kontroll utan templat (NTC6). (g) För Master Mix 7, NRAS Exon 4 kommer ytterligare tre reaktioner att behövas per provplatta för NRAS Control Wild-Type, NRAS Positive Control Exon 4 och NRAS Exon 4 kontroll utan templat (NTC7). OBS! Beakta att en Master Mix-volym som är något större är denna beräkning krävs för att ge utrymme för visst svinn vid pipettering. 8. Märk 0,2 mL PCR-rören eller brunnarna i en 96-hålsplatta med vederbörlig provinformation. 9. Märk 2,0 mL centrifugeringsrör för preparering av Master Mix-blandning. 10. Tillsätt nödvändig volym vatten för molekylärbiologi till 2,0 mL centrifugeringsrör som är märkta Master Mix. 11. Tillsätt nödvändig mängd DNA Polymerase 10X PCR Buffer till Master Mix-rören. 12. Tillsätt nödvändig mängd 10 mM dNTP:er till Master Mix-rören. 13. Tillsätt nödvändig mängd KRAS eller NRAS Forward Primer till respektive Master Mix-rör. 14. Tillsätt nödvändig mängd KRAS eller NRAS Reverse Primer till respektive Master Mix-rör. 15. Ta ut DNA Polymerase (PN 703310) ur frysen. 16. Centrifugera DNA Polymerase i ~10 sekunder. 17. Vortexblanda DNA Polymerase i ~10 sekunder. 18. Tillsätt nödvändig mängd DNA Polymerase till Master Mix-rören. 19. Förslut Master Mix-rören. 20. Vortexblanda Master Mix-rören i ~30 sekunder och centrifugera under en kort stund i ~10 sekunder före användning. 21. Förvara på is tills det används. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 16 12 Stegvisa anvisningar 22. Pipettera 48,0 µL (se anmärkning ovan i steg 6) av Master Mix-blandningen i lämpliga brunnar och byt pipettspets om du använder en pipett med en kanal. Se till att det inte spiller eller stänker mellan hålen om en repeterpipett används. Förvara plattan på is. 23. Tillsätt 2,0 µL (se anmärkning ovan i steg 6) av varje provtemplat-DNA eller vatten (NTC no template control) till lämpliga brunnar. Använd separata pipettspetsar för varje prov och undvik korskontamineriong av proverna genom stänk. Förslut alla brunnar med med provDNA och NTC med remsor med 8 proppar (om du använder en 96-hålsplatta) eller sätt på proppar på 0,2 mL PCR-rören. Se till att propparna sluter till ordentligt. 24. Endast därefter ska kittets kontrolltemplat-DNA (PNs 710131, 710135, 710136, 710137, 710138, 710441, 710442, 710443 & 710444) öppnas ett i taget. Pipettera 2,0 µL av var och en av kontrolltemplaten sist för att minska risken för att något prov-DNA kontamineras. När pipetteringen är klar, slut till alla brunnar med remsor med 8 proppar (om du använder en 96-hålsplatta) eller sätt på proppar på 0,2 mL PCR-rören. Se till att propparna sluter till ordentligt. OBS! Det är god laboratoriepraxis att placera kontroller utan templat (NTC) i brunnar som inte ligger intill Positive Controls eller prover. OBS! För förslag på hur plattor med 96 brunnar kan arrangeras för analys av alla KRAS och NRAS Exons 2-4, se Bilaga A.1 Översikt av platta för SURVEYOR Scan-kit. 25. Vortexblanda (~1/2 hastighet) i 10 sekunder. 26. Centrifugera i 1-2 minuter för att tillförsäkra att alla lösningar samlas i botten av brunnarna eller rören. Kontrollera att lösningarna är på botten av brunnen eller röret. Upprepa centrifugeringen i annat fall. 12.6. Termocykelprogram för amplifieringsprotokoll 1. Använd följande termocykelprotokoll för PCR-amplifiering och heteroduplexformering: 15 cykler 30 cykler 1 cykel Inledande denaturering 95 C Touchdown-amplifiering 95 C 62 C, -0,5 ºC/cykel 72 C Amplifiering 95 C 55 C 72 C Slutgiltig förlängning 72 C Heteroduplex formering 95 ºC ≤12 C 5 minuter 30 sekunder 30 sekunder 25 sekunder 30 sekunder 30 sekunder 25 sekunder 2 minuter 2 minuter Paus 12.7 Kvalitetskontroll av PCR-produkter 1. Kvaliteten och kvantiteten på amplikoner bör kontrolleras med gelelektrofores (eller motsvarande) innan du fortsätter med SURVEYOR Nuclease-nedbrytning. 2. Analysera en alikvot av PCR-produkt tillsammans med flera olika mängder av en 100-bp DNA-stege. 3. Använd stegen till att beräkna koncentrationen av amplifierad DNA. 4. Bara ett enda band >20 ng/µL som motsvarar huvud-PCR-produkten ska synas. 5. Om det finns flera band, se till att kvaliteten på tillsatt DNA-templat var tillräckligt god (se Bilaga B – Felsökningsguide). Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 17 12 Stegvisa anvisningar 6. Om ingen produkt kan iakttas, se till att kvaliteten på tillförd DNA-templat var tillräckligt god (se Bilaga B – Felsökningsguide). Om kvaliteten motsvarar specifikationerna, ska du öka volymen på templatet till 4,0 µL per 50 µL reaktion (minska vattenmängden per reaktion till 31,0 µL). 7. Inga PCR-produkter ska synas i kontrollprovet utan templat. Om DNA-produkt kan iakttas är kontrollen troligen kontaminerad, se Bilaga B – Felsökningsguide. 8. Ange PCR som robust eller svag PCR. a. Robust PCR ska ha ett enda band med minst 20 ng/µL. b. Svag PCR ska ha ett enda band med mindre än 20 ng/µL. c. Fortsätt med SURVEYOR Nuclease-nedbrytning vid såväl robusta som svaga PCR-bedömningar. TIPS: I det här stadiet kan PCR-produkter förvaras vid -20 ºC eller kallare i upp till en vecka. 12.8 SURVEYOR Nuclease-nedbrytning 1. När prov-PCR har bedömts vara av tillräckligt hög kvalitet och kvantitet, ska nedbrytning med SURVEYOR Nuclease utföras enligt beskrivningen nedan. En provkoncentration om minst 40 ng/uL krävs för optimal nedbrytning med SURVEYOR Nuclease 2. Tina rören med 0,15 M MgCl2 Solution och SURVEYOR Enhancer Cofactor på is. 3. Tillsätt 10,0 µL av varje PCR-amplifierat prov till ett nytt 0,2 mL PCR-rör eller brunn på en platta med 96 brunnar. 4. Bered en ny blandning 0,15 M MgCl2 Solution, SURVEYOR Enhancer Cofactor, SURVEYOR Enhancer W2 och SURVEYOR Nuclease W (SURVEYOR Nucleaseblandning). Använd följande tabell som en vägledning för beredning av Master Mix för SURVEYOR Nucleasenedbrytning för analys av flera prover. Nedanstående exempel har volymer för en Master Mix för 10 prover. Tänk på att en Master Mix-volym som är något större än denna beräkning krävs för att ge utrymme för svinn vid pipettering. Antal SURVEYOR Nuclease-nedbrytningsreaktioner: Volymberäkning: 0,15 M MgCl2 Solution (µL) SURVEYOR Enhancer Cofactor (µL) SURVEYOR Enhancer W2 (µL) SURVEYOR Nuclease W (µL) Total volym Master Mix: Tillsätt 5 µL SURVEYOR Nuclease Master Mix till varje PCR-amplifierat prov (µL) Total volym SURVEYOR Nucleasenedbrytningsreaktion: 10 10,0 10,0 10,0 20,0 50,0 10,0 15,0 a. Centrifugera alla reagenser före användning. b. Vortexblanda varje reagens försiktigt före pipettering; centrifugera under en kort stund i ~10 sekunder efter varje vortexmoment. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 18 12 Stegvisa anvisningar c. För varje nedbrytning, tillför följande komponenter till ett 0,2 mL PCR (eller större) mikrocentrifugeringsrör. 1,0 µL 0,15 M MgCl2 Solution (PN 708027) 1,0 µL SURVEYOR Enhancer Cofactor (PN 708049) 1,0 µL SURVEYOR Enhancer W2 (PN 710161) 2,0 µL SURVEYOR Nuclease W (PN 710160) Eller tillsätt 5 µL Master Mix som beretts enligt ovanstående tabell. 5. Vortexblanda försiktigt Master Mix för SURVEYOR Nuclease-nedbrytning i 10 sekunder på låg hastighet. 6. Centrifugera Master Mix för SURVEYOR Nuclease-nedbrytning i 10 sekunder på låg hastighet. 7. Placera Master Mix för SURVEYOR Nuclease-nedbrytning på is tills den är redo att användas. Obs! Master Mix för SURVEYOR Nuclease-nedbrytning som beretts i steg 7 ska användas omedelbart eftersom SURVEYOR Nuclease W blir inaktivt över tid vid förekomst av de övriga komponenterna i Master Mix för SURVEYOR Nucleasenedbrytning. 8. Pipettera 5,0 µL-alikvot av Master Mix för SURVEYOR Nuclease-nedbrytning i varje rör eller brunn med 10,0 µL alikvot av amplifierad PCR-produkt (se steg 3 ovan). 9. När pipetteringen är klar, centrifugera 0,2 mL PCR-rör eller plattan med 96 brunnar i 10 sekunder. 10. Vortexblanda försiktigt 0,2 mL PCR-rör eller plattan med 96 brunnar i 10 sekunder. 11. Centrifugera i 10 sekunder med låg hastighet (detta moment är extra viktigt om nedbrytningen sker i ett instrument som saknar uppvärmt lock). 12. Inkubera vid 42 °C i 30 minuter. 13. Tillsätt 1,0 µL SURVEYOR Stop Solution (PN 708030) i varje rör eller brunn och vortexblanda försiktigt (total SURVEYOR Nuclease-reaktionsvolym är 16,0 µL). TIPS: SURVEYOR nedbrytningsprodukter kan lagras vid ≤ -20 ºC i upp till en vecka. 14. Ladda provnedbrytningarna i ett DHPLC-system. Obs! För föreslagna gradientinställningar för DHPLC-systemet för analys av SURVEYOR Nuclease-nedbrytningar, besök http://world.transgenomic.com/diagnostic-tools/geneticanalysis-kits/crc-rascan-kits-eu/dhplcsystemsettings. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 19 13 Kontrollparametrar 13 Kontrollparametrar 13.1 Kvalitetskontroll av CRC RAScan Combination Kit Kontroll-DNA-plasmider ingår i detta kit för att tillhandahålla kvalitetskontroller vid specifika steg i analysprocessen. För amplifieringsprotokollet, erbjuder dessa kontroller ett sätt att säkerställa att Master Mix-blandningarna är korrekt preparerade och att amplifieringen fungerar korrekt. Kontrollsubstanser utan templat (där vatten tillförs istället för DNA-templat) krävs också för att kontrollera förekomsten av eventuell kontamination av kitkomponenter från ett ovidkommande DNA-templat. I nedbrytningsstadiet av SURVEYOR Nuclease, tillhandahåller amplikonerna från dessa kontrollDNA-plasmider en effektiv kontroll av klyvningsreaktionsförhållanden (beredning av Master Mix för SURVEYOR Nuclease-nedbrytning och inkubationsförhållanden) har varit tillfredsställande. I analysstadiet ger DHPLC System kromatogram av SURVEYOR Nuclease-nedbrutna kontrollamplikoner en vägledning huruvida klyvningsprodukttopparna motsvarande den mutationen i KRAS och NRAS Exons 2, 3 och 4 kommer att elueras även vid låga nivåer (se figurer 4-10). Klyvningsprodukttoppar som motsvarar andra mutationer i KRAS eller NRAS Exons 2, 3 och 4 kan elueras i något avvikande positioner. Om PCR-amplikonerna inte motsvarar resultaten som beskrivs i Kvalitetskontroll av PCRprodukter, se Bilaga B - Felsökningsguide eller kontakta Transgenomics tekniska support innan du går vidare med ytterligare provanalyssteg. 13.2 Användning av kontrollplasmid-DNA Detta kit är utrustat med nio kontroll-DNA: KRAS Control Wild-Type; PN 710131 KRAS Positive Control Exon 2; PN 710135 KRAS Positive Control Exon 3; PN 710136 KRAS Positive Control Exon 4A; PN 710137 KRAS Positive Control Exon 4B; PN 710138 NRAS Control Wild-Type; PN 710441 NRAS Positive Control Exon 2; PN 710442 NRAS Positive Control Exon 3; PN 710443 NRAS Positive Control Exon 4; PN 710444 Dessa kontroll-DNA är plasmider med tillägg. Den Positive Controls innehåller två plasmider: en blandning av KRAS eller NRAS Control Wild-Type och en mutationsklon som skiljer sig från WildType vid ett enstaka baspar. Kontrollerna tillhandahålls i separata flaskor, var och en med en koncentration på 105 kopior/µL. KRAS och NRAS Exons 2, 3 och 4 Forward och Reverse PCR-primrar som krävs för PCRamplifiering tillhandahålls separat i kitten. Följ instruktionerna som står i Amplifieringsprotokollet, SURVEYOR Nuclease-nedbrytning och Analys av KRAS och NRAS Exons 2, 3 och 4 med SURVEYOR Nuclease för användning av dessa kontroller. VI REKOMMENDERAR STARKT ATT NYA ANVÄNDARE FÖRST UTFÖR EXPERIMENTEN MED ENBART KONTROLLSUBSTANSERNA INNAN TESTNING GÖRS PÅ GENOMISKA PROVER Bruksanvisning för CRC RAScan Kit med DHPLC-system Page 20 14 Tolkning av resultat 14 Tolkning av resultat 14.1 Analys av KRAS och NRAS Exons 2, 3 och 4 med SURVEYOR Nuclease För jämförelse och kontroll ska man ALLTID utföra SURVEYOR Nuclease-nedbrytning på var och en av kontrollerna (Wild-Type och Positive Controls), en kontroll utan templat (NTC), samt provDNA som ska köras med samma provplatta i samma DHPLC-system. I nedbrytningsstadiet av SURVEYOR Nuclease, tillhandahåller amplikonerna från dessa kontrollplasmid-DNA en effektiv kontroll att förhållandena för klyvningsreaktion (beredning av SURVEYOR Nuclease-blandning och inkuberingsförhållanden) var tillfredsställande. I analysstadiet ger DHPLC-systemets kromatogram av SURVEYOR Nuclease-nedbrutna kontrollamplikoner en vägledning om var DNA-fragmenten efter klyvningen vid dessa specifika mutationers avvikelseställe kommer att elueras, även vid låga nivåer (se figurerna 4-10). Om antingen PCR-amplikonerna eller SURVEYOR Nuclease-klyvningsfragment som härletts av kontroll-DNA inte motsvarar beskrivna resultat, se Bilaga B -Felsökningsguide eller kontakta Transgenomics tekniska support innan nästa steg i provanalysen tas. Nedanstående exempel är endast i illustrationssyfte och ska INTE användas för att fastställa identiteten hos någon viss mutation. Bekräftelse av identiteten hos en mutation krävs för att oåterkalleligt fastställa förekomsten av en specifik basförändring i Exons 2, 3 eller 4 i KRAS- eller NRAS-generna. SURVEYOR Nuclease klyver vid alla avvikelser som uppstår till följd av heteroduplex formering mellan Wild-Type och muterat DNA, inte bara mutationer i Exons 2, 3 eller 4. SURVEYOR Nuclease bekräftar att en avvikelse förekommer. Mutationens specifika basförändringsidentitet krävs för bestämning av KRAS- och NRAS-aktiverande status och den måste bekräftas med en annan metod, t.ex. sekvensering. 14.2 Granskning av SURVEYOR Scan-resultat Inspektera kromatogrammen och jämför de för Wild-Type och Positive Controls med provets. Fastställ om provets kromatogram liknar Wild-Type-mönstret eller ej. Om det är annorlunda bör provet betraktas som "SURVEYOR Scan-positivt" och skickas till DNA-sekvensanalys. Exempel på sådana prov finns i figurer 4-10 och Bilaga B – Felsökningsguide, problem 7 och 8. Ett prov med SURVEYOR Nuclease-mönster som skiljer sig från mönstret för Wild-Type bör skickas för sekvensbekräftelse även om det inte är identiskt med den Positive Controls. Ett exempel på ett sådant prov finns i Bilaga B – Felsökningsguide, problem 7. Zooma vid behov in på området av intresse och lägg provets kromatogram ovanpå Wild-Type Control för den amplikonen. Notera skillnader mellan Wild-Type Control och det analyserade provet. Viktigt! Efter en grundlig genomgång av varje prov bör datagranskaren undersöka om intilliggande prover i analysplattan har identiska positiva SURVEYOR Scan-resultat. Om identiska positiva resultat förekommer kan de bero på korskontamination av prov eller kontroll och analysen måste upprepas. Bruksanvisning för CRC RAScan Kit med DHPLC-system Page 21 14 Tolkning av resultat 14.3 Exempel på resultat Exempel på resultat som erhållits med CRC RAScan Combination Kit med ett DHPLC-system visas i figurerna 4-10 nedan. I dessa exempel har den metod som beskrivs i Stegvisa anvisningar med användning av WAVE® 4500HT-HS System med såväl UV- som fluorescensdetektorer, följts exakt. För föreslagna gradientinställningar för DHPLC-system för analys av SURVEYOR Nuclease-nedbrytningar, besök http://world.transgenomic.com/diagnostictools/genetic-analysis-kits/crc-rascan-kits-eu/dhplcsystemsettings. Figur 4A DHPLC-genomflöde G12D ger 74- och 116-bpfragment KRAS Control Wild-Type KRAS Control Exon 2 OkluvenProv 1, KRAS Exon 2 200-bp Prov 2, KRAS Exon 2 amplikonProv 3, KRAS Exon 2 DNA-storleksmarkör Okluven 190-bp amplikon DHPLCavrinning Figur 4B G12D ger 74- och 116-bpfragment KRAS Control Wild-Type KRAS Control Exon 2 OkluvenProv 1, KRAS Exon 2 200-bp Prov 2, KRAS Exon 2 amplikonProv 3, KRAS Exon 2 DNA-storleksmarkör Okluven 190-bp amplikon Figurerna 4A och 4B: WAVE DHPLC-analys med UV-detektion (figur 4A) och fluorescensdetektion (figur 4B) av SURVEYOR Nuclease-nedbrytning av KRAS Positive Control Exon 2 och KRAS Control Wild-Type samt CRC DNA som isolerats från FFPEsektioner. Vid visuell granskning av kromatogrammen ska de nedbrutna proverna jämföras med Wild-Type Control (brun linje). KRAS Positive Control Exon 2 (blå linje) är en blandning av WildType och muterade G12D-plasmider som ger nedbrytningstoppar på 74 och 116 bp; denna kontroll visar att nedbrytningsmomentet i SURVEYOR Nuclease fungerar och visar det område i kromatografen som bör undersökas visuellt för att fastställa om ett prov ska skickas för DNAsekvensanalys. När nedbrytningsmönster jämförs för proverna 1-3 med Wild-Type ej nedbrutet kromatogram är det uppenbart att proverna 1 och 2 (röda och turkosa linjer) har sannolika mutationer och bör sekvenseras för att bekräfta förekomst eller frånvaro av en mutation i relevant kodon. Prov 3 (grön linje) har liknande kromatogram som de som observeras för Wild-Type Control och behöver inte skickas för DNA-sekvensanalys. Observera att sekvenseringsresultatet är det definitiva resultatet eftersom det kan finnas andra ej relevanta mutationer som kan ge samma fregmentstorlekar. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 22 14 Tolkning av resultat Figur 5A DHPLCgenomflöde Q61H ger 65- och 135-bpfragment KRAS Control Wild-Type KRAS Control Exon 3 OkluvenProv 1, KRAS Exon 3 200-bp Prov 2, KRAS Exon 3 amplikonProv 3, KRAS Exon 3 DNA-storleksmarkör Okluven 200-bp amplikon DHPLCavrinning Figur 5B Q61H ger 65- och 135-bpfragment KRAS Control Wild-Type KRAS Control Exon 3 OkluvenProv 1, KRAS Exon 3 200-bp Prov 2, KRAS Exon 3 amplikonProv 3, KRAS Exon 3 DNA-storleksmarkör Okluven 200-bp amplikon Figurerna 5A och 5B: WAVE DHPLC-analys med UV-detektion (figur 5A) och fluorescensdetektion (figur 5B) av SURVEYOR Nuclease-nedbrytning av KRAS Positive Control Exon 3 och KRAS Control Wild-Type samt CRC DNA som isolerats från FFPEsektioner. Vid visuell granskning av kromatogrammen ska de nedbrutna proverna jämföras med Wild-Type Control (brun linje). KRAS Positive Control Exon 3 (grön linje) är en blandning av WildType och muterade Q61H-plasmider som ger nedbrytningstoppar på 65 och 135 bp; denna kontroll visar att nedbrytningsmomentet i SURVEYOR Nuclease fungerar och visar det område i kromatografen som bör undersökas visuellt för att fastställa om ett prov ska skickas för DNAsekvensanalys. När nedbrytningsmönster jämförs för proverna 1-3 med Wild-Type ej nedbrutet kromatogram är det uppenbart att prov 2 (svart linje) har en sannolik mutation och bör sekvenseras för att bekräfta förekomst eller frånvaro av en mutation i relevant kodon. Prov 1 och 3 (blå och röda linjer) har liknande kromatogram som de som observeras för Wild-Type Control och behöver inte skickas för DNA-sekvensanalys. Observera att sekvenseringsresultatet är det definitiva resultatet eftersom det kan finnas andra ej relevanta mutationer som kan ge samma fregmentstorlekar. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 23 14 Tolkning av resultat Figur 6A DHPLCgenomflöde K117N ger 80- och 88-bpfragment DHPLCKRAS Control Wild-Type avrinning KRAS Control Exon 4A OkluvenProv 1, KRAS Exon 4A 200-bp Prov 2, KRAS Exon 4A amplikonDNA-storleksmarkör Okluven 168-bp amplikon DHPLCavrinning Figur 6B K117N ger 80- och 88-bpfragment DHPLCKRAS Control Wild-Type avrinning KRAS Control Exon 4A OkluvenProv 1, KRAS Exon 4A 200-bp Prov 2, KRAS Exon 4A amplikonDNA-storleksmarkör Okluven 168-bp amplikon Figurerna 6A och 6B: WAVE DHPLC-analys med UV-detektion (figur 6A) och fluorescensdetektion (figur 6B) av SURVEYOR Nuclease-nedbrytning av KRAS Positive Control Exon 4A och KRAS Control Wild-Type och CRC DNA som isolerats från FFPEsektioner. Vid visuell granskning av kromatogrammen ska de nedbrutna proverna jämföras med Wild-Type Control (grön linje). KRAS Positive Control Exon 4A (blå linje) är en blandning av WildType och muterade K117N-plasmider som ger nedbrytningstoppar på 80 och 88 bp; denna kontroll visar att nedbrytningsmomentet i SURVEYOR Nuclease fungerar och visar det område i kromatografen som bör undersökas visuellt för att fastställa om ett prov ska skickas för DNAsekvensanalys. När nedbrytningsmönster jämförs för proverna 1 och 2 med Wild-Type ej nedbrutet kromatogram är det uppenbart att prov 1 (röd linje) har en sannolik mutation och bör sekvenseras för att bekräfta förekomst eller frånvaro av en mutation i relevant kodon. Prov 2 (svart linje) har liknande kromatogram som de som observeras för Wild-Type Control och behöver inte skickas för DNA-sekvensanalys. Observera att sekvenseringsresultatet är det definitiva resultatet eftersom det kan finnas andra ej relevanta mutationer som kan ge samma fregmentstorlekar. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 24 14 Tolkning av resultat Figur 7A DHPLCgenomflöde Okluven 194-bp amplikon DHPLCKRAS Control Wild-Type avrinning KRAS Control Exon 4B OkluvenProv 1, KRAS Exon 4B 200-bp Prov 2, KRAS Exon 4B amplikonProv 3, KRAS Exon 4B DNA-storleksmarkör DHPLCavrinning A146T ger 78- och 116-bpfragment Figur 7B A146T ger 78- och 116-bpfragment DHPLCKRAS Control Wild-Type avrinning KRAS Control Exon 4B OkluvenProv 1, KRAS Exon 4B 200-bp Prov 2, KRAS Exon 4B amplikonProv 3, KRAS Exon 4B DNA-storleksmarkör Okluven 194-bp amplikon Figurerna 7A och 7B: WAVE DHPLC-analys med UV-detektion (figur 7A) och fluorescensdetektion (figur 7B) av SURVEYOR Nuclease-nedbrytning av KRAS Positive Control Exon 4B och KRAS Control Wild-Type och CRC DNA som isolerats från FFPEsektioner. Vid visuell granskning av kromatogrammen ska de nedbrutna proverna jämföras med Wild-Type Control (brun linje). KRAS Positive Control Exon 4B (mörkgrön linje) är en blandning av Wild-Type och muterade A146T-plasmider som ger nedbrytningstoppar på 78 och 116 bp; denna kontroll visar att nedbrytningsmomentet i SURVEYOR Nuclease fungerar och visar det område i kromatografen som bör undersökas visuellt för att fastställa om ett prov ska skickas för DNAsekvensanalys. När nedbrytningsmönster jämförs för proverna 1 — 3 med Wild-Type ej nedbrutet kromatogram är det uppenbart att prov 3 (röd linje) har en sannolik mutation och bör sekvenseras för att bekräfta förekomst eller frånvaro av en mutation i relevant kodon. Prov 2 och 3 (svarta och ljusgröna linjer) har liknande kromatogram som de som observeras för Wild-Type Control och behöver inte skickas för DNA-sekvensanalys. Observera att sekvenseringsresultatet är det definitiva resultatet eftersom det kan finnas andra ej relevanta mutationer som kan ge samma fregmentstorlekar. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 25 14 Tolkning av resultat Figur 8A NRAS Control Wild-Type NRAS Control Exon 2 OkluvenProv 1, NRAS Exon 2 200-bp Prov 2, NRAS Exon 2 amplikonDNA-storleksmarkör DHPLCgenomflöde G12D ger 88- och 148-bpfragment Okluven 236-bp amplikon DHPLCavrinning Figur 8B G12D ger 88- och 148-bpfragment NRAS Control Wild-Type NRAS Control Exon 2 OkluvenProv 1, NRAS Exon 2 200-bp Prov 2, NRAS Exon 2 amplikonDNA-storleksmarkör Okluven 236-bp amplikon Figurerna 8A och 8B: WAVE DHPLC-analys med UV-detektion (figur 8A) och fluorescensdetektion (figur 8B) av SURVEYOR Nuclease-nedbrytning av NRAS Positive Control Exon 3 och Wild-Type-Controls samt CRC DNA som isolerats från FFPE-sektioner. Vid visuell granskning av kromatogrammen ska de nedbrutna proverna jämföras med NRAS Control Wild-Type (blå linje). NRAS Positive Control Exon 2 (grön linje) är en blandning av WildType och muterade G12D-plasmider som ger nedbrytningstoppar på 88 och 148 bp; denna kontroll visar att nedbrytningsmomentet i SURVEYOR Nuclease fungerar och visar det område i kromatografen som bör undersökas visuellt för att fastställa om ett prov ska skickas för DNAsekvensanalys. När nedbrytningsmönster jämförs för proverna 1 och 2 med Wild-Type nedbrutet kromatogram är det uppenbart att prov 1 (röd linje) har en sannolik mutation och bör sekvenseras för att bekräfta förekomst eller frånvaro av en mutation i relevant kodon. Prov 2 (svart linje) har liknande kromatogram som de som observeras för NRAS Control Wild-Type och behöver inte skickas för DNA-sekvensanalys. Observera att sekvenseringsresultatet är det definitiva resultatet eftersom det kan finnas andra ej relevanta mutationer som kan ge samma fregmentstorlekar. Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 26 14 Tolkning av resultat Figur 9A NRAS Control Wild-Type NRAS Control Exon 3 OkluvenProv 1, NRAS Exon 3 200-bp Prov 2, NRAS Exon 3 amplikonDNA-storleksmarkör DHPLCgenomflöde DHPLCavrinning Q61K ger 78- och 125-bpfragment Okluven 203-bp amplikon Figur 9B NRAS Control Wild-Type NRAS Control Exon 3 OkluvenProv 1, NRAS Exon 3 200-bp Prov 2, NRAS Exon 3 amplikonDNA-storleksmarkör Q61K ger 78- och 125bpfragment Okluven 203-bp amplikon Figurerna 9A och 9B: WAVE DHPLC-analys med UV-detektion (figur 9A) och fluorescensdetektion (figur 9B) av SURVEYOR Nuclease-nedbrytning av NRAS Positive Control Exon 3 och Wild-Type-Controls samt CRC DNA som isolerats från FFPE-sektioner. Vid visuell granskning av kromatogrammen ska de nedbrutna proverna jämföras med NRAS Control Wild-Type (grön linje). NRAS Positive Control Exon 3 (blå linje) är en blandning av WildType och muterade Q61K-plasmider som ger nedbrytningstoppar på 78 och 125 bp; denna kontroll visar att nedbrytningsmomentet i SURVEYOR Nuclease fungerar och visar det område i kromatografen som bör undersökas visuellt för att fastställa om ett prov ska skickas för DNAsekvensanalys. När nedbrytningsmönster jämförs för proverna 1 och 2 med Wild-Type ej nedbrutet kromatogram är det uppenbart att prov 1 (röd linje) har en sannolik mutation och bör sekvenseras för att bekräfta förekomst eller frånvaro av en mutation i relevant kodon. Prov 2 (svart linje) har liknande kromatogram som de som observeras för NRAS Control Wild-Type och behöver inte skickas för DNA-sekvensanalys. Observera att sekvenseringsresultatet är det definitiva resultatet eftersom det kan finnas andra ej relevanta mutationer som kan ge samma fregmentstorlekar. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 27 14 Tolkning av resultat Figur 10A DHPLCgenomflöde NRAS Control Wild-Type NRAS Control Exon 4 OkluvenProv 1, NRAS Exon 4 200-bp Prov 2, NRAS Exon 4 amplikonDNA-storleksmarkör Okluven 233-bp amplikon DHPLCavrinning A146T ger 68- och 165-bpfragment Figur 10B A146T ger 68- och 165-bpfragment NRAS Control Wild-Type NRAS Control Exon 4 OkluvenProv 1, NRAS Exon 4 200-bp Prov 2, NRAS Exon 4 amplikonDNA-storleksmarkör Okluven 233-bp amplikon Figurerna 10A och 10B: WAVE DHPLC-analys med UV-detektion (figur 10A) och fluorescensdetektion (figur 10B) av SURVEYOR Nuclease-nedbrytning av NRAS Positive Control Exon 4 och Wild-Type Controls samt CRC DNA som isolerats från FFPE-sektioner. Vid visuell granskning av kromatogrammen ska de nedbrutna proverna jämföras med NRAS Control Wild-Type (grön linje). NRAS Positive Control Exon 4 (blå linje) är en blandning av WildType och muterade A146T-plasmider som ger nedbrytningstoppar på 68 och 165 bp; denna kontroll visar att nedbrytningsmomentet i SURVEYOR Nuclease fungerar och visar det område i kromatografen som bör undersökas visuellt för att fastställa om ett prov ska skickas för DNAsekvensanalys. Vid jämförelse mellan nedbrytningsmönster för prover 1 och 2 med Wild-Type nedbrutna kromatogram, är det tydligt att kromatogrammen för proverna 1 och 2 (röda och svarta linjer) liknar det som observerats för NRAS Control Wild-Type och de behöver inte skickas till DNA-sekvensanalys. Observera att sekvenseringsresultatet är det definitiva resultatet för ett positivt SURVEYOR Scan-resultat, eftersom det kan finnas andra ej relevanta mutationer som kan ge samma fregmentstorlekar. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 28 15 Prestationsegenskaper 15 Prestationsegenskaper 15.1 LOD för mutationer med SURVEYOR Scan-kit Validering av SURVEYOR Scan-kitten för DHPLC-plattformar med användning av plasmidkloner av alla indicerade mutationer har visat att SURVEYOR Nuclease-toppar kan detekteras i en blandning av 2-5% mutation i en Wild-Type-bakgrund. Automatiserad sekvenseringsbestämning av både framåt och bakåt-strängar misslyckas ofta med att detektera mutationer i blandningar med mindre än 10% Wild-Type-DNA. Tillsammans med resultaten från SURVEYOR Nuclease är det möjligt att med högre säkerhet tolka kromatogram om 5-10% för mutant sekvensering. Om analysen visar ett positivt SURVEYOR Scan-resultat, men utan detekterbar KRAS- eller NRAS-mutation med DNA-sekvensering, rekommenderar vi att resultatet "Ingen avvikelse detekterad" registreras. Ytterligare detaljer finns i avsnittet Att tänka på avseende arbetsflödet. 15.2 Sekvensbekräftelse Fortsätt med sekvensbekräftelse för alla positiva SURVEYOR Scan-resultat för att fastställa sekvensidentiteten för KRAS eller NRAS Exons 2, 3 eller 4-mutationer. Figur 11 är ett exempel på vikten av sekvensbekräftelse för att fastställa exakt mutation. Fortsätt inte med sekvensbekräftelse om SURVEYOR Scan-resultatet var negativt. Provet kan rapporteras som Wild-Type eller ingen avvikelse detekterad. Universal Sequencing Primers som inkluderas i kittet är avsedda för sekvensbekräftelse. Framåtprimer är PN 710153F (Universal Sequencing Primer 1) och bakåtprimer är PN 710153R (Universal Sequencing Primer 2). Prov 1: G13C Prov 2: G13D Figur 11 Sekvensanalys av prover med samma KRAS Exon 2 SURVEYOR Nuclease nedbrytningsmönster. Proverna 1 och 2 ovan visade samma DHPLC-toppmönstwer efter SURVEYOR Nuclease-nedbrytning och analyserades vidare med DNA-sekvensering. Sekvenseringen visade det är två olika mutationer; G13C och G13D. Dessa prover illustrerar (a) förmågan hos SURVEYOR Nuclease att detektera potentiella mutationer men (b) inte fastställa exakt placering eller specifik basförändring hos mutation(erna) i provet. Obs! Det är viktigt att kontrollera att ingen "stämpel"-sekvens förekommer i varje sekvenserings kromatogram för att bekräfta att mutationen inte beror på att provet kontaminerats av en av kittets kontroller. Obs! Om provet är Wild-Type vid relevanta kodon av intresse och den "stämplade" sekvensen förekommer, är provet kontaminerat och analysen måste göras om. Förekomst av "stämpeln" anger att provet kan vara kontaminerat med en av kittets Wild-Type-Controls. Denna kontamination kan maskera eventuell lågnivåmutation i provet. Se A.2 Kontrollernas DNAstämplar för information om kontrollernas stämpelsekvenser. Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 29 15 Prestationsegenskaper 15.3 Analysprocessens begränsningar Kontaminerande ämnen som överförs genom extraktion av formalinfixerade paraffininbäddade prover kan inverka negativt på PCR-amplifieringen och nedbrytningsprocessen med SURVEYOR Nuclease. Rutinerna för kvalitetskontroll som beskrivs under Kvalitetskontroll av PCR-produkter säkerställer att extraherad DNA är lämplig för användning i detta kit. Detta kit har validerats för analys av formalinfixerade paraffininbäddade kolorektala cancertumörprover. Det har inte validerats för diagnostisk användning med andra cancertyper eller med färska eller frysta biopsiprover. Läs i Bilaga B - Felsökningsguide, nedan, för felsökning av onormala resultat och information om faktorer som kan påverka analysen. Försiktighet måste iakttas för att undvika överföring och korskontamination med detta kit. På grund av analysmetodens extrema känslighet, måste försiktighetsåtgärder vidtas vid följande punkter: Se till att alla prover hanteras på ett sådant sätt att korskontamination mellan prover inte kan förekomma Arbeta i en PCR-arbetsstation eller annat lämpligt utrymme där arbetsområdet kan utsättas för UV-ljus innan PCR-amplifieringsreaktionerna ställs in. Använd en separat PCR-arbetsstation eller ett separat rum för att öppna proverna efter PCR-amplifiering för kvalitetskontroll med gelelektrofores. Förberedelse för SURVEYOR Nuclease-nedbrytning bör göras i ett separat rum eller på en annan PCR-arbetsstation än den arbetsstation där inledande PCR ställdes in. Se till att kittets plasmidkontroller hanteras separat från testproverna under alla stadier i analysen. o Kontrollera att alla lösningar, kontroll utan templat, samt prov-DNA-brunnar är förslutna innan rören med kontrollplasmid-DNA öppnas. o KONTROLLER SKA HANTERAS SIST. Tillsätt kontrollplasmid-DNA till relevanta rör först EFTER DET ATT ALLA kontroller utan templat och provbrunnar har förslutits. o När kontroll-DNA-rören har förslutits ska ALLA rör och lock torkas av med ett medel som utplånar DNA (t.ex. 10% blekmedel) innan de överförs till ett annat område. Se till att pipettering av prover i plattor med 96 brunnar inte möjliggör provkontamination av intilliggande brunnar på grund av stänk vid blandningen eller på grund av att pipettspetsarna inte bytts ut. Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 30 Bilaga A Bilaga A A.1 Översikt av platta för SURVEYOR Scan kit Den översikt av plattan som föreslås nedan är för SURVEYOR Scan-analys för alla sju KRAS och NRAS Exons samtidigt i 10 prover. Förklaring: Ex = Exon; WT = Wild-Type; Mut = Mutation; NTC = No Template Control (kontroll utan templat) A.2 Kontrollernas DNA-stämplar Sekvenserna med en "stämpel" anges nedan. Förklaring: De vanligaste mutationsområdena markeras med lila. Sekvenser med VERSALER är kodande baser; ej kodande baser anges med gemener. Kontrollernas genetiska "stämpel" är markerad i gult; denna avvikelse från KRAS eller NRAS Wild-Type-sekvensen, i en region som inte förväntas ha mutationer, kan användas för att felsöka provkontamination av Positive Controls. Se Bilaga B Felsökningsguide, problem 8, för ett exempel på en SURVEYOR Scan-kurva av sådan kontamination. KRAS Exon 2-"stämpel" Amplikonstorlek: 190 bp. För att skapa KRAS Exon 2-kontrollplasmidens genetiska stämpel har TAT ändrats till ATA. Kodon 12 och 13 har också markerats nedan. GCTGGTGGCGTAGGCAAGAGTGCCTTGACGATACAGCTAATTCAGAATCATTTTGTGGACGAAATAGAT KRAS Exon 3-"stämpel" Amplikonstorlek: 200 bp. För att skapa KRAS Exon 3-kontrollplasmidens genetiska stämpel har ACC ändrats till TGG. Kodon 59 och 61 har också markerats nedan. GAATGGTGTCTCTTGGATATTCTCGACACAGCAGGTCAAGAG KRAS Exon 4A-"stämpel" Amplikonstorlek: 168 bp. För att skapa KRAS Exon 4A-kontrollplasmidens genetiska stämpel har AGA ändrats till TCT. Kodon 117 har också markerats nedan. AATAAATGTGATTTGCCTTCTAGAACAGTTCTCAC Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 31 Bilaga A KRAS Exon 4B-"stämpel" Amplikonstorlek: 194 bp. För att skapa KRAS Exon 4B-kontrollplasmidens genetiska stämpel har agt ändrats till tca. Kodon 146 har också markerats nedan. TCAGCAAAGACAAGACAGgtatcaaac NRAS Exon 2-"stämpel" Amplikonstorlek: 236 bp. För att skapa NRAS Exon 2-kontrollplasmidens genetiska stämpel har aca ändrats till tgt. Kodon 12 och 13 har också markerats nedan. ccatgtggttcttgctggtgtgaaATGACTGAGTACAAACTGGTGGTGGTTGGAGCAGGTGGTGTT NRAS Exon 3-"stämpel" Amplikonstorlek: 203 bp. För att skapa NRAS Exon 3-kontrollplasmidens genetiska stämpel har GAC ändrats till CTG. Kodon 59 och 61 har också markerats nedan. ACAGCTGGACAAGAAGAGTACAGTGCCATGAGACTGCAA NRAS Exon 4-"stämpel" Amplikonstorlek: 233 bp. För att skapa NRAS Exon 4-kontrollplasmidens genetiska stämpel har CAA ändrats till GTT. Kodon 117 och 146 har också markerats nedan. AACAAGTGTGATTTGCCAACAAGGACAGTTGATACAAAAGTTGCCCACGAACTGGCCAAGAGTTACGGG ATTCCATTCATTGAAACCTCAGCCAAG A.3 Systemkrav för denaturering med HPLC (DHPLC) A.3.1 DHPLC-specifikationer för SURVEYOR SCAN-användningar Följande specifikationer är minimikrav för DHPLC-systemets specifikationer för att utföra analyser med SURVEYOR Scan KRAS- och NRAS-kittet. Tillförselsystem för högpresterande gradientlösning Binär gradient kapacitet; högtryck eller lågtryck Flödeshastighetskontroll, minsta område: 0,7 – 1,6 mL/min Flödeshastighet: ± 2% (H2O vid 20 ºC) Lösningsavgasare Autoprovtagare Temperaturkontroll för kylning av plattor, inställningsbar: 4 till 14 ºC Injektionsvolym: 5–50 µL Separationskassett Utformad för separation av dsDNA-fragmentstorlek, fragmentstorlekar 50 till 250 bp Ugn med temperaturkontroll med hög precision Temperaturområde: 40 till 70 ºC Temperaturprecision: ± 0,2 ºC Temperatur-reproducerbarhet: ± 0,2 ºC Linjäritet över hela temperaturområdet: ± 2,0 ºC Detektionsalternativ 1 UV-/VIS-detektor Område för våglängd: 190-700 nm UV-källa: Deuteriumlampa Detektionsalternativ 2 Fluorescensdetektor Område för våglängd: excitation: 200-85 nm; emission: 250-900 nm Fluorescenskälla: 150 W xenonlampa Pump för fluorescerande färg Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 32 Bilaga A Fast flödeshastighet vid 0,1 mL/min – 20%/+50% Lågt pulsationsflöde A.3.2 Systemdrift För inställning och drift av DHPLC-systemet, läs och följ tillverkarens rekommendationer för analys av dsDNA-fragment i storleksområdet 50 bp till 250 bp, med inriktning på storleksurskiljning om 10 bp eller bättre. Detta är storleksområdet för fragment efter SURVEYOR Nuclease-nedbrytning med användning av detta kit. För föreslagna gradientinställningar för DHPLC-system för analys av SURVEYOR Nucleasenedbrytningar, besök http://world.transgenomic.com/diagnostic-tools/genetic-analysis-kits/crcrascan-kits-eu/dhplcsystemsettings. A.3.3 Underhåll av DHPLC-kassetter Följ tillverkarens anvisningar beträffande underhåll av DHPLC-kassetter. Obs! Analys av SURVEYOR Nuclease-reaktioner kräver mer frekventa och striktare rengöringsprotokoll än de som används för vanlig PCR-analys. Läs i riktlinjerna för ditt DHPLCsystem för mer information. A.4 Laboratorieinstallation för PCR-analyser För tillförlitliga och kontaminationsfria PCR-resultat ska god laboratoriesed iakttas när PCRlaboratoriet utformas. När laboratoriet planeras ska behovet av avstånd mellan förberedelse för PCR-amplifiering och aktiviteter efter amplifiering beaktas. Det är viktigt att separera (i) DNA-isolering: (ii) PCRamplifiering; och (iii) aktiviteter efter PCR, t.ex. att öppna amplifierade prover vid förberedelse för att köra geler och förbereda andra analyser, t.ex. SURVEYOR Nuclease-nedbrytningar och DNAsekvensering. Det är också viktigt att laboratorieutrustning och material tilldelas varje område och att de endast används i det specifika området. Genom att torka av ytorna med 10 % (v/v) blekmedel, som blandas varje vecka, kommer underlättas eliminering av DNA-fragment ≤ 500 bp som templat för PCR. Det kan vidare vara gynnsamt att behandla plastmaterial och lösningar med UV-ljus med kort våglängd i 7-10 minuter med användning av en UV-korslänkare. Obs! Enzymer och DNA som ska amplifieras får inte UV-behandlas. Genom att använda lämplig skyddsutrustning, byta handskar ofta samt torka av handskbeklädda händer med 10 % (v/v) blekmedel före inträde i en arbetsstation kommer att var till stor hjälp för att reducera risken för kontamination. Det bör minst finnas ett arrangemang som liknar det tvårumsarrangemang som visas i nedanstående diagram som är specifikt utformat för PCR och analys med SURVEYOR Nuclease. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 33 Bilaga A Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 34 Bilaga A A.5 Litteraturlista 1. Amgen News Release (2013) New analyses identify predictive biomarkers for Vectibix (panitumumab) in patients with metastatic colorectal cancer. http://wwwext.amgen.com/media/media_pr_detail.jsp?-year=2013&releaseID=1820728 ® 2. Peeters M et al (2013) Massively parallel tumor multigene sequencing to evaluate response to panitumumab in a randomized phase III study of metastatic colorectal cancer. Clin Cancer Res. 19 1902-12. 3. De Roock W et al (2010) Association of KRAS p.G13D mutation with outcome in patients with chemotherapy-refractory metastatic colorectal cancer treated with cetuximab. JAMA 304 1812-20. 4. Peeters M et al (2013) Mutant KRAS codon 12 and 13 alleles in patients with metastatic colorectal cancer: assessment as prognostic and predictive biomarkers of response to panitumumab. J Clin Oncol. 31 759-65. 5. Andre T et al (2013) Panitumumab combined with irinotecan for patients with KRAS wildtype metastatic colorectal cancer refractory to standard chemotherapy: a GERCOR efficacy, tolerance, and translational molecular study. Ann Oncol. 24 412-9. 6. Loupakis F et al (2009) KRAS codon 61, 146 and BRAF mutations predict resistance to cetuximab plus irinotecan in KRAS codon 12 and 13 wild-type metastatic colorectal cancer. Br J Cancer 101 715-21. 7. De Roock W et al (2010) Effects of KRAS, BRAF, NRAS, and PIK3CA mutations on the efficacy of cetuximab plus chemotherapy in chemotherapy-refractory metastatic colorectal cancer: a retrospective consortium analysis. Lancet Oncol. 11 753-62. 8. Qiu P et al (2004) Mutation detection using Surveyor™ nuclease. Biotechniques 36 702707 9. Kuang Y et al (2009) Noninvasive detection of EGFR T790M in gefitinib or erlotinib resistant non-small cell lung cancer. Clin. Cancer Res. 15 2630-6 10. Mitani N et al (2007) Surveyor nuclease-based detection of p53 gene mutations in haematological malignancy. Ann. Clin. Biochem. 44 557-9 11. Engelman JA et al (2006) Allelic dilution obscures detection of a biologically significant resistance mutation in EGFR-amplified lung cancer. J. Clin. Invest. 116 2695-2706 Se även http://world.transgenomic.com/files/literature/482146.pdf SURVEYOR Nuclease och dess tillämpningar. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system för referenser om Sid. 35 Bilaga B Bilaga B Felsökningsguide Effektiv användning av SURVEYOR Scan kit för DHPLC är beroende av att ett antal olika steg genomförs framgångsrikt. Ett av de mest avgörande stegen är PCR-amplifiering som måste resultera i produktion av specifika, jämnstora DNA i tillräcklig kvantitet för att kunna detekteras efter hybridisering och klyvning. Detta beror i sin tur på kvaliteten på det ursprungliga provet. Att utföra analysen med DNA som inte motsvarar kvalitetsvillkoren avseende kvalitet och kvantitet rekommenderas inte. Obs! Om det är första gången du använder SURVEYOR Nuclease ska du utföra experimenten i avsnittet Använda kontrollplasmid-DNA efter det att du har läst och förstått avsnittet Stegvisa anvisningar. Se till att du har resultaten från kontrollplasmid-DNA innan du kontaktar Transgenomics tekniska support. Denna felsökningsguide omfattar en rad problem som du kan komma att stöta på medan du använder SURVEYOR Scan-kitter för DHPLC samt förslag på hur du kan lösa dem. De exempel som visas kommer från både KRAS och NRAS Exons. Läs i instrumenthandboken för DHPLC-systemet för detaljerad information om användning och underhåll av instrumentet. Handboken innehåller specifika processer för att kontrollera och underhålla kolumnprestation och inkluderar felsökningsinformation. Problem 1 – Låg PCR-avkastning eller avsaknad av PCR-produkt vid analys på agarosgel LÅG PCRAVKASTNING God Dålig PCRPCRavkastningavkastning MÖJLIG ORSAK LÖSNING Dålig kvalitet på DNAtemplat Gör om reningen av DNA; gå igenom den använda reningsmetoden Om FFPEextraherat material är alltför fragmenterat kan alternativa FFPE-block behövas. För mycket RNA i provet som orsakar att DNAkoncentrationen beräknas för högt Upprepa RNase-behandlingen av DNAprovet och upprepa DNA-reningen. Termocyklern är inte kalibrerad Kalibrera termocyklern. Otillräckligt med templat Öka mängden templat. RNAband Obs! Högkvalitativ DNA från FFPE ska användas. DNA bör ha en koncentration på 25 ng/µL enligt bestämning genom absorption av 260 nm, ha en absorptionsgrad vid 260/280 nm som överstiger 1,8 samt vara högre än 90% DNA (d.v.s. fritt från den mesta tRNA- och rRNAkontaminationen enligt visuell bedömning på agarosgel). Förvara DNA-prover vid en temperatur mellan -20 °C och -80 °C. Analys av DNA-templat som extraherats från paraffininbäddad vävnad kräver iakttagande av ett flertal försiktighetsåtgärder. Extraherat DNA kan behandlas med uracil DNA-glykosylas för att förebygga amplifiering av DNA-fragment som innehåller deaminerade C-rester. En hög procentandel av det A260-absorberande material som extraheras från paraffininbäddad vävnad kommer ofta inte att amplifieras väl under PCR. Genom att använda en större mängd DNA till starten kan man ofta producera en tillräcklig mängd med amplifieringsprodukt. En annan sak att tänka på är att välja FFPE-tumörsektioner med en hög andel tumörceller. Mikrodissektion kan också användas, men detta är tidskrävande och torde inte rekommenderas vid generell laboratorietestning. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 36 Bilaga B Problem 2 – Flera PCR-produkter vid analys på agarosgel FLERA PCR-PRODUKTER God PCRavkastning MÖJLIG ORSAK LÖSNING För låg härdningstemperatur Kontrollera termocyklerns kalibrering. Flera PCRband Obs! PCR ska producera en tillräckligt stor avkastning (>20 ng/µL) av EN ENSTAKA amplifierad typ av korrekt storlek. DNA Polymerase och DNA Polymerase 10X PCR Buffer som tillhandahålls i detta kit måste användas för PCR. Amplikoner från kontroller ska brytas ned med SURVEYOR Nuclease och köras för att utesluta falska bakgrundsstörningar genom visuell jämförelse av kromatogramprofiler (se Exempel på resultat, figurer 4-10). Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 37 Bilaga B Problem 3 – Inga klyvningsprodukter observeras vid analysen efter behandling av heteroduplex med SURVEYOR Nuclease INGA KLYVNINGSPRODUKTER Position för saknade heteroduplex MÖJLIG ORSAK LÖSNING Andelen avvikande mål är för liten Kontrollera analysprocessen genom att använda kontrollsubstanserna. Ineffektiv formering av heteroduplex Följ korrekt PCR- och hybridiseringsprocess. Använd nyss hybridiserad DNA vid SURVEYOR Nucleasenedbrytning. Utför nya PCRamplifiering med genom att (1) använda samma kvantitet DNAtemplat; (2) öka mängden start-DNA; eller (3) isolera färsk DNA från samma eller en annan FFPEsektion. Ej beskuren homoduplextopp Overksam SURVEYOR Nuclease Utför Positive Controlreaktion för att kontrollera enzymfunktionen. Använd endast färska SURVEYOR Nuclease Master Mixblandningar. Obs! SURVEYOR Nuclease klyver huvudsakligen avvikelser i heteroduplex. Andelen heteroduplex i förhållande till homoduplex i det hybridiserade provet avgör storleken på SURVEYOR Nuclease-nedbrytningssignal. Om KRAS- eller NRAS-mutationen förekommer i väldigt låga koncentrationer i det genomiska DNA-provet, kan signalen vara för svag för att ge ett positivt resultat. Det är viktigt att se till att hybridiseringssteget ingår i termocykelprogrammet (se Amplifieringsprotokoll) för att maximera effektiviteten i heteroduplexformationen. Heteroduplex är väldigt ineffektivt formade under vanliga PCR-reaktioner. Obs! Förhållandet mellan signal och bakgrundsstörningar är normalt tillräckligt högt för att detektera mutationer som förekommer i en liten andel av de totala DNA-templaten. Det är möjligt att detektera 1 till 2% av muterat DNA. Figurer 4-10 visar nedbrytningsprodukter som genereras med homoduplex och heteroduplex KRAS och NRAS Positive Control-DNA (inkluderas i detta kit) samt FFPE-prover på ett WAVE DHPLC 4500-system. Klyvningsprodukterna syns tydligt som två nya toppar eluerande med de förväntade storlekar som kan beräknas i förhållande till DNAstorleksmarkören. Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 38 Bilaga B Varning! Kommersiellt tillgängliga PCR-buffertar varierar kraftigt när det gäller innehåll och sammansättningen anges ofta inte av leverantörerna. Ett flertal buffertar är INTE kompatibla med SURVEYOR Nuclease på grund av pH-värdet eller förekomsten av tillsatser, ytaktiva ämnen eller andra skyddade ingredienser. ANVÄND INTE någon annan polymeras eller 10X polymerasbuffert än de som tillhandahålls i detta kit. Problem 4 – Höga bakgrundsstörningar efter behandling med SURVEYOR Nuclease HÖGA BAKGRUNDSSTÖRNINGAR MÖJLIG ORSAK LÖSNING Undermåligt hybridiseringssteg Gör så här: 1. Se till att DNAkoncentrationen är i en nivå >25 ng/µL. 2. Upprepa steget med heteroduplex formering och var noga med att följa rekommenderad process; se 12.7.1. DHPLCgenomflöde Prov med mycket störningar i bakgrunden DHPLCtvätt Mängden DNA är för låg Kontrollera avkastning och kvalitet på DNA-templat Obestämda PCRprodukter Kontrollera avkastning och kvalitet på DNA-templat SURVEYOR Enhancer W2 och/eller SURVEYOR Enhancer Cofactor har förlorat verkan Kontrollera kittets utgångsdatum OBS! Det kan hända att SURVEYOR Nuclease rispar dubbelsträngad DNA vid slumpvist matchade ställen. Detta orsakar störningar efter nedbrytningen. Denna aktivitet dämpas av SURVEYOR Enhancer W2 och dess Cofaktor utan att ha andra negativa effekter på klyvningen av avvikelser. SURVEYOR Enhancer W2 och Cofaktor ingår i detta kit och skall alltid användas. Då denna typ av rispning fortfarande förekommer, genereras mindre toppar på DHPLC-systemet. Nedbrytningen av Wild Type Control kommer att visa dessa mindre toppar. Denna jämförelse används för att söka efter skillnader i elektroferogrammönsten mellan Wild-Type Control och provet och används vid jämförelse av provets mönster med Wild-Type Control. Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 39 Bilaga B Problem 5 – Toppar i negativa kontroller med SURVEYOR Nucleasenedbrytning NEDBRYTNINGSTOPPAR I NEGATIVA KONTROLLER Nedbrytningstoppar med svag signal i Wild-Typekontroll DHPLCgenomflöde Ej beskuren homoduplextopp MÖJLIG ORSAK LÖSNING Kittets innehåll har kontaminerats med KRAS- elller NRASamplikoner eller plasmidkontroller Kassera alla kitkomponenter och använd ett nytt kit. DHPLCtvätt Kontakta Transgenomic tekniska support för att diskutera eventuella anledningar och källor till kontaminationen Problem 6 – Misslyckade SURVEYOR Nuclease-resultat SURVEYOR NUCLEASE-NEDBRYTNING AV POSITIVE CONTROLS HAR MISSLYCKATS Inga tydliga nedbrytnings toppar Ej beskuren homoduplextopp MÖJLIG ORSAK LÖSNING SURVEYOR Nuclease tillsattes inte Bryt ned ett nytt prov SURVEYOR Nucleasenedbrytning skedde vid fel temperatur Bryt ned ett nytt prov Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 40 Bilaga B Problem 7 – Oväntade toppar i SURVEYOR Nuclease-nedbrytningskurva SURVEYOR NUCLEASE-NEDBRYTNING AV POSITIVE CONTROLS HAR MISSLYCKATS MÖJLIG ORSAK Mutation på ovanlig plats Oväntad nedbrytningstopp DHPLCgenomflöde Förväntade nedbrytningstoppar Ej beskuren homoduplextopp LÖSNING Sekvensera provet för att bekräfta mutationen DHPLCtvätt Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 41 Bilaga B Problem 8 – Proverna kontamineras av Positive Controls NEDBRYTNINGSMÖNSTER SOM ÖVERENSSTÄMMER MED FÖREKOMST AV BLANDADE POSITIVE CONTROLS STÄMPLADE REGION OCH WILD-TYPE HETERODUPLEX MÖJLIG ORSAK Dålig pipetteringsteknik Prov 1 Prov 2 Prov 3 LÖSNING Var försiktig vid pipettering för att undvika korskontamination. Kontrollera kontrollstämplarnas sekvensregioner för kontamination av Positive Control. Sekvenering visar att NRAS Exon 2 stämpelregioner kontaminerar ett prov. Kodon 12 och 13 region av NRAS Exon 2 Plasmidkontrollens "Stämpel"-region av NRAS Exon 2 Provet är Wild-Type Stämpelsekvensen finns Kontaminerad Dålig pipetteringsteknik Var försiktig vid pipettering för att undvika korskontamination. Kontrollera kontrollstämplarnas sekvensregioner för kontamination av Positive Control. Provet är muterat Ingen stämpelsekvens Giltigt prov Provet är Wild-Type Ingen stämpelsekvens finns Giltigt prov Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 42 Beställningsinformation Beställningsinformation Produktnummer Produktnamn Storlek 710104 SURVEYOR Scan KRAS Kit Exon 2 CE IVD 100 reaktioner 710106 SURVEYOR Scan KRAS Kit Exons 3 & 4 CE IVD 100 reaktioner 710400 SURVEYOR Scan NRAS Kit Exons 2, 3 & 4 CE IVD 100 reaktioner 710077 CRC RAScan Combination Kit 230 reaktioner Kontaktuppgifter Huvudkontor Europa Transgenomic, Inc. 12325 Emmet Street Omaha Nebraska 68164 United States of America Transgenomic Limited 40 Watt Road Hillington Park, Hillington Glasgow G52 4RY United Kingdom Tel: (888) 233-9283* eller +1 (402) 452-5400 Fax: +1 (402) 452-5401 E-post: [email protected] Tel: +44 141 892 8800 Fax: +44 141 883 5967 E-post: [email protected] *gäller bara Nordamerika www.Transgenomic.com Översättning Ansvarsfriskrivning Transgenomic, Inc. har gjort allt för att säkerställa riktigheten i denna översättning. Om du har frågor om någon information i denna översatta version av handboken, se den engelska versionen Instructions for Use for the CRC RAScan™ Combination Kit for DHPLC Systems – 482427(EN) http://world.Transgenomic.com/files/literature/482427(EN).pdf och / eller kontakt Transgenomic vid [email protected]. Bruksanvisning för CRC RAScan Combination Kit med DHPLC-system Sid. 43 Varumärken & upphovsrätt Varumärken & upphovsrätt SURVEYOR Nuclease: Denna produkt tillverkas med en exklusiv licens under amerikanska patent 6 391 557; 5 869 245; deras utländska motsvarigheter och andra sökta patent. Användning av SURVEYOR Nuclease kräver licens från Transgenomic. Utbildnings-, ideella och vinstdrivande organisationer har begränsad rätt att använda SURVEYOR Nuclease för forskning genom inköp av denna produkt. Återförsäljning eller andra tillämpningar är strängt förbjudna. Var vänlig kontakta Transgenomic för mer information. DNA Polymerase: Användning av denna produkt omfattas av ett eller flera av följande patent i US och motsvarande patent utanför USA:, 5,789,224, 5,618,711, 6,127,155 samt patentanspråk utanför USA motsvarande amerikanskt patent nummer 4,889,818. Köp av denna produkt inkluderar en begränsad, ej överlåtbar immunitet mot intrångsanspråk enligt ovanstående patent vid användning av endast denna mängd produkt för köparens egna interna forskning. Ingen rättighet under något annat patent (t.ex. de patenterade 5' nukleasprocessanspråken i de amerikanska patenten 5 210 015 och 5 487 972), ingen rätt att utföra någon patenterad metod, och ingen rätt att utföra kommersiella tjänster av något slag, inklusive bland annat rapportering av resultaten av köparens aktiviteter mot avgift eller andra kommersiella överenskommelser, förmedlas vare sig uttryckligen, underförstått eller på grund av tidigare dispens. Denna produkt är endast avsedd för forskning. Diagnostisk användning under Roches patent kräver en separat licens från Roche. Ytterligare information om köp av licenser kan erhållas genom att kontakta Director of Licensing, Applied Biosystems, 850 Lincoln Centre Drive, Foster City, California 94404, USA. Transgenomic, SURVEYOR och WAVE är registrerade varumärken och Navigator, CRC RAScan, spirallogotypen samt Advancing Personalized Medicine är varumärken som tillhör Transgenomic, Inc. Alla övriga varumärken tillhör sina respektive innehavare. © 2013 Transgenomic, Inc. Med ensamrätt. Tryckt i USA. Dokumentnr. 482427(SV) rev-00 Bruksanvisning för CRC RAScan Kit med DHPLC-system Sid. 44