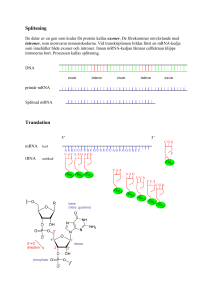
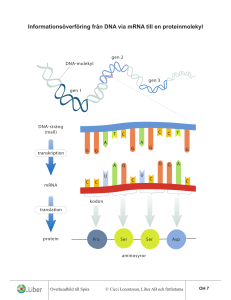
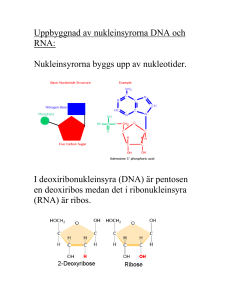
Human Molekylärgenetik
advertisement

2008-12-08 Human Molekylärgenetik Del 2 – Identifiering av riskgener för g g komplexa sjukdomar ANDERS MÄLARSTIG, molekylärgenetiker på enheten för aterosklerosforskning, Centrum för Molekylärmedicin, Karolinska Institutet, Solna [email protected] Monogen sjukdom Nära 100 % penetrans Låg incidens Miljöfaktorer mindre viktiga Exempel: huntingtons l h sjukdom, cystisk fibros, hyperkolesterolemi Fenotypen beror helt på vilken gen och i vilken position i genen variationen finns Kopplingsstudie Komplex sjukdom Låg penetrans Hög incidiens Miljöfaktorer lika viktiga som genetiska Exempel: hjärtinfarkt, stroke, benskörhet, reumatoid artrit, grön starr, Chrons sjukdom, manodepressivitet Patienter med sjukdomen är en heterogen grupp med h d olika bakomliggande orsaker till sjukdom Associationsstudie 1 2008-12-08 Vetenskaplig frågeställning / hypotes Design av experimentell eller klinisk studie Bioinformatik Genotypning replikering Statistisk analys Funktionella studier Tillämpning Identifiering av riskgen Från känd biologi Kandidatgen Genome‐wide association Genotypning av ett stort antal SNPs spridda över genomet Genotypning av ett mindre antal SNPs i d t l SNP Statistisk analys Statistisk analys Funktionella studier och bekräftande kliniska studier 2 2008-12-08 Kandidatgen • Utgångspunkt i kända biologiska sjukdomsmekanismer • Prövar en hypotes, men den är begränsad av befintlig Prövar en hypotes men den är begränsad av befintlig biologisk kunskap om sjukdomen • Enkel epidemiologisk statistik kan användas • Många SNPs inom en liten region kan bestämmas för att maximera genetisk information Associationsstudie helgenoms SNP • Genome‐wide association prövar hypoteser som ännu inte formulerats • Stora fall‐kontrollstudier Stora fall‐kontrollstudier • Chip tekniker som Affymetrix och Illumina • Helgenoms‐chip för mellan 100k SNPs och 1M SNPs • Särskilda statistiska tekniker som beaktar att ett stort antal statistiska test görs 3 2008-12-08 Helgenomsstudier har identifierat flera sjukdomslokus senaste 2 åren bl.a. • • • • • • TCF7L2 för typ‐2 diabetes Kromosom 9 p21 för hjärtinfarkt och typ‐2 Kromosom 9 p21 för hjärtinfarkt och typ 2 diabetes diabetes Kromosom 6q23 och IRF5 genen för reumatoid artrit 10q26 i den okända genen LOC387715 för grön starr FTO genen för ökat body mass index m.m. Venös trombos (blodpropp) • Allvarligt tillstånd där blodflödet i en ven hindras, vid lungemboli livshotande • Hög ålder, cancer, p‐piller och kirurgiska ingrepp är Hög ålder cancer p‐piller och kirurgiska ingrepp är vanliga riskfaktorer • Incidens cirka 175 fall / 100 000 inv. och år • Genetiska riskfaktorer i factor 5, protrombin, protein C och S generna, vilka ingår i reglering av koagulation – SNPs i 5 andra gener har visat association till sjukdomen SNPs i 5 andra gener har visat association till sjukdomen • Ärftlighet över 50 % 4 2008-12-08 Urval av fall och kontroller Rekrytering av patientgrupp via klinik Rekrytering av matchade friska individer via populationsregister individer via populationsregister Venöst blodprov för att ta DNA och blodplasma Exkludering av patienter som inte uppfyller fördefinierade kriterier, Längd, vikt, ålder, livsstilsvanor dokumenteras Databas Bestämning av plasmaprotein i blodprover Upprättande av biobank Framrening av DNA från blod Genotypning av 300k SNPs med chip teknik Teknologier för SNP genotypning • • • • • Pålitliga Snabba K t d ff kti Kostnadseffektiva Robotiserade –mindre manuellt arbete Flexibilitet vid design av metod 5 2008-12-08 ATGCC Princip för genom‐wide associattion TCGGG AAATG ATGCT TCATAT ATGCT 300 000 5000 TCATAT 10 GCGCT 1 6 2008-12-08 Illumina 7 2008-12-08 8 2008-12-08 CHR SNP POSITION GENE LOC MAF HWE_P A 1 A 2 CHISQ P_CMH OR_CMH X rs2563751 90976 PCDH11X 5UTR 46.7% 0.79 T C 59.75 1.1E‐14 2.4 X rs34259897 92210 PCDH11X flanking_3UTR 20.3% 0.24 A G 49.19 2.3E‐12 0.5 X rs2573828 91516 ‐ ‐ 48.8% 0.92 T C 41.55 1.2E‐10 0.5 5 rs34868670 40273 PTGER4 flanking_5UTR 36.4% 0.97 C T 37.09 1.1E‐09 1.5 1 rs12743401 200743 PPP1R12B intron 36.7% 0.75 C T 37.01 1.2E‐09 1.5 1 rs3817222 200731 PPP1R12B coding 35.9% 0.96 T C 36.98 1.2E‐09 1.5 1 rs3881953 200794 PPP1R12B coding 36.3% 0.93 A G 36.66 1.4E‐09 1.5 1 rs12734338 200736 PPP1R12B intron 36.8% 0.19 C T 36.59 1.5E‐09 1.5 1 rs3354 94123 F3 3' UTR 28.0% 0.94 C T 35.24 1.3E‐09 1.6 13 rs2451078 18996 TPTE2 intron 48.5% 0.79 C G 30.85 2.8E‐08 1.4 X rs2524583 91047 ‐ ‐ 34.7% 1.00 G T 29.68 5.1E‐08 0.5 9 rs7866590 132700 ABL1 X rs4341301 91587 ‐ 12 rs3217907 4277 X 6292 rs6529942 2.2% 0.98 T C 25.77 3.8E‐07 3.2 ‐ intron 42.2% 0.79 T C 23.12 1.5E‐06 1.7 CCND2 intron 33.9% 0.95 A C 21.25 4.0E‐06 0.7 NLGN4X flanking_5UTR 36.8% 0.18 T C 21 4.6E‐06 0.6 Hardy‐Weinberg equilibrium This model relies on the following assumptions: a. b. c. d. e. f. g. Infinite population size. Discrete generations. Random mating. No selection. No migration. No mutation. Equal initial genotype frequencies in the two sexes. 9 2008-12-08 Hardy‐Weinberg equilibrium If we define the frequencies of the alleles as: • p = P(A) = u + v/2 • q = P(a) = v/2 + w then, the genotype frequencies are: • P(A/A) = p2 • P(A/a) = 2pq • P(a/a) = q2 Second generation respects the same distribution: P(AA)= (p2 + ½2 pq)2 = [p(p+q)]2 = p2 P(Aa)= 2(p2 + ½2pq) (½2pq +q2) =2p(p+q)q(p+q)= 2pq P(aa) = (½2pq + q2)2 = [q(p + q)]2 = q2 THE HARDY‐WEINBERG LAW • p + q = 1 • p2 + 2pq + q2 = 1 • p = frequency of the dominant allele in the population q = frequency of the recessive allele in the population • p2 = percentage of homozygous dominant individuals q2 = percentage of homozygous recessive individuals 2pq = percentage of heterozygous individuals 10 2008-12-08 Statistisk metodik 1, Hardy‐Weinberg Equilibrium (kvalitetskontroll av genotypning) Hardy‐Weinberg observed Controls AA Aa aa 146 129 28 AA Aa aa 161 155 49 SUM Patients SUM expected chi‐square p‐value (1‐tailed) 146,238 0,0004 128,523 0,0018 28,238 0,0020 0,004171869 0,9485 check result 155,842 165 316 165,316 43,842 0,1707 0,6438 0 6438 0,6069 1,421413249 0,233171 check result p-värde >0.05 ger att fördelningen av genotyper av vår SNP är inom Hardy-Weinberg equilibrium Statistisk metodik 2 Allele Frequency rs3354 Kontroller Fall CC CT TT 182 CC CT TT 161 count CT total frequency C 129 28 185 678 72,7% 253 730 65,3% 155 49 11 2008-12-08 Statistisk metodik 3 –homogenitetstest med chi‐två fördelningen Kontroller Observed 185 493 678 C T Total Kolumn ChiTvåvärde Exp 210 91 210,91 467,09 Fall Observed 253 477 730 Exp Total rad 227 09 227,09 438 502,91 970 1408 3,18 1,44 2,96 1,34 8 91 8,91 ChiTvåvärde Kritiskt värde p‐value (1‐tailed) 3,841 0,0028 check result P-värde <0.05, alltså är fördelningen av allelerna är signifikant skilda i fall och kontroller. Högre frekvens av T allel i patientgruppen ger att: T allelen ökar risken för venös blodpropp. Vilka andra SNPs finns i tissue factor genen? Primary Transcript rs592435 Exon 4 rs610277 Exon 5 rs3917642 rs3917641 rs958587 Exon 3 rs762484 rs3917643 Exon 1 Exon 2 rs1034363 Exon 6 rs1144300 rs3354 -2695 rs644530 -9914 rs698942 F3 Gene 12 2008-12-08 Samvariation mellan SNPs (www.hapmap.org) R2-måttet D’-måttet bäst 1 sämst 0 NCBI dbSNP refSNP ID: rs3354 human (Homo Organism: sapiens) Molecule Type: Genomic Created/Updated in 36/127 build: Allele SNP: Variation Class: single nucleotide polymorphism p y p Alleles: C/T Ancestral Allele: C Map to Genome 36.2 Build: GeneView via analysis of contig annotation: F3 coagulation factor III (thromboplastin, tissue factor) Click to see [all] [cSNP] [has frequency] [double hit] [haplotype tagged] variations associated with this gene. …….GTTAAAAATT AAAACTTGGA ATTGGTTGTA GTACCATT C/T GTTACATTTC AAAGTGACTA ATGCTGATGT CAAAACCAGA……. 13 2008-12-08 Allel och genotypfrekvenser Individual Chrom. ss# Population G Group Sample Cnt. C/C C/T T/T HWP C T 184AF 0.710 0.290 120IG 0.500 0.483 0.017 0.050 0.742 0.258 HapMap‐HCB Asian 90IG 0.267 0.578 0.156 0.254 0.556 0.444 HapMap‐JPT HapMap JPT 88IG 0 295 0.545 0.295 0 545 0.159 0 159 0.479 0 479 0.568 0 568 0.432 0 432 120IG 0.633 0.283 0.083 0.150 0.775 0.225 74IG 0.554 0.446 ss1882765 CEPH Source HapMap‐CEU European HapMap‐YRI Asian Sub‐Saharan African CHMJ Asian Prediktion av vilka SNPs som kan påverka funktion • PolyPhen ‐ Predikterar hur en aminosyraförändring påverkar proteinets hydrofobicitet, isolelektriska punkt och struktur • Ensembl – Sammanfogar information från ett hundratal databaser för regioner i genomet • Fylogenetisk footprinting (ECR‐browser) –undersöker om SNPn ligger i en region som är evolutionärt konserverad genom att jämföra ortologa sekvenser i andra arter • Transfac –undersöker vilka transkriptionsfaktorer som p potentiellt kan binda i regionen där SNPn är positionerad • ESEfinder –söker potentiella splice sites 14 2008-12-08 Splice‐sites The first 400 basepairs in the tissue factor promoter and potential transcription factor binding sites 15 2008-12-08 Studiedesign för uppföljande studier • • • • Fall‐kontroll (enklast) Populationsbaserad prospektiv studie Populationsbaserad prospektiv studie Prospektiv patientstudie Friska frivilliga för en kvantitativ fenotyp som är relaterad till sjukdomen exempelvis koncentration av tissue factor protein i blodet Urval av genotypningsmetoder Gamla metoder RFLP Allelspecifik amplifiering Sangersekvensering Moderna metoder för uppföljande studier Realtids PCR (Taqman och Lightcycler) ASO (allele specifik oligonukleotid) ASO (allele‐specifik oligonukleotid) hybridisering OLA (oligonukleotid ligering) Single‐base extension techniques (Illumina+Affymetrix) Pyrosekvensering Massive parallell sequencing (Solexa Massive parallell sequencing (Solexa, 454‐sequencing) 16 2008-12-08 RFLP Elektroforesbaserad detektion 17 2008-12-08 Allelspecifik amplifiering Taqman‐probe 18 2008-12-08 Realtids PCR Alleldiskriminering med realtids‐PCR CC CT TT 19 2008-12-08 Molekylärgenetisk funktionalitet • Protein – mängd – aktivitet • aminosyrasekvens och struktur – post‐translationella förändringar (tex fosforylering) – interaktion med andra protein – timing Exempel: Variationer IRF‐5 genen 20 2008-12-08 Detektion av mRNA förändringar • Studier i rätt typ av cell under förhållanden som efterliknar sjukdomen • Long‐range PCR och elektrofores kan detektera förekomsten av flera transkript (splice‐varianter) • Realtids‐PCR kan detektera hur mycket mRNA som finns i ett prov • Odling av celler, stopp av nysyntes av med Actinomycin‐D, detektion med realtids PCR (halveringstid för mRNA) • Jämföra exonsekvens i DNA med mRNA sekvens för allelisk Jämföra exonsekvens i DNA med mRNA sekvens för allelisk obalans • Kloning av luciferas‐reporter gene i en gens promotor kan visa skillnader i transkriptionseffektivitet för ena eller andra allelen 21 2008-12-08 Allelspecifikt mRNA uttryck G/C C/A Regulatory region Coding region Obalans av alleluttryck i mRNA G C mRNA C A mRNA Kaijzel et al Genes Immunity 2001;2:135-144 Kureeman et al Hum Mol Genetics 2004;13:1755-1762 Tillämpningar genetik • Identifiering av biologiska signalvägar och mekanismer • Identifiering av helt nya läkemedel Identifiering av helt nya läkemedel • Farmakogenetik (effektivitet och biverkningar) • Tidiga preventiva åtgärder för bärare av riskvarianter • Genterapi 22 2008-12-08 VISION OM GENETISKA TEST Sjukdomsrisk Träningsråd K t åd Kostråd Läkemedel GENETISKT TEST FÖR • Att tidigt kunna förutsäga sjukdomsrisk • Kost och livsstil • Lägga upp träning • Effektivitet och biverkningsrisk av läkemedel INVÄNDNINGAR • Hjälp att ta emot informationen behövs! Eff k i ä fö hi d • Effektiva sätt att förhindra att sjukdomen bryter ut måste hittas! • Kliniska prövningar om kostrekommendationerna på genetisk basis är effektivt behövs. • Mer kunskap behövs. • Kan användas begränsat idag för tex VKORC1 genen. Många studier görs i skrivande stund. 23