fulltext - DiVA portal
advertisement

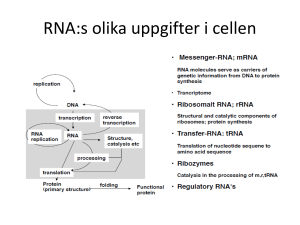
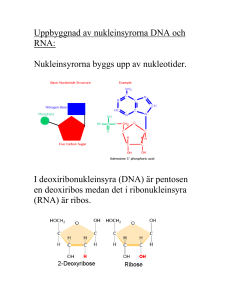
Institutionen för fysik, kemi och biologi Examensarbete 16 hp Histonmodifieringar och alternativ splicing Jenny Berggren LiTH-IFM- Ex--11/2498--SE Handledare: Johan Edqvist, Linköpings universitet Examinator: Anders Hargeby, Linköpings universitet Institutionen för fysik, kemi och biologi Linköpings universitet 581 83 Linköping Avdelning, Institution Division, Department Datum Date Biologi, IFM Biology, IFM 2011-06-01 Rapporttyp Report category Språk Language x Svenska/Swedish Engelska/English ________________ Licentiatavhandling x Examensarbete C-uppsats D-uppsats Övrig rapport ISBN __________________________________________________ ISRN __________________________________________________ Serietitel och serienummer ISSN LITH-IFM-A-EX--—11/2498—SE _______________ Handledare URL för elektronisk version Supervisor: Johan Edqvist Ort Location: Linköping Sammanfattning Abstract: Alternative splicing of pre-mRNA generates protein diversity. Histone modifications are connected to the regulation of alternative splicing through adaptor systems that transfers the epigenetic information directly to the splicing factors. The cis- acting RNA elements on the exons and introns together with the trans- regulating splicing factors are therefore directly affected of specific histone modifications. An integrated model over several DNA process mechanisms is suggested. This complex model explains the interactions of the different parts and how they affect each other. Chromatin remodelers are required to obtain euchromatin. Nucleosome positioning at exon rich regions with a specific modification pattern point out where the exons are, and this enable the RNA polymerase II to find and bind to the DNA. It’s CTD domain recruits both splicing- and modifications factors. The transcription rate is also affected of the nucleosome positioning and that in turn affects the recruitment of the components of the spliceosomen, other trans- acting regulators and even the formation of the secondary structure of the pre-mRNA transcript. Chromatin- adaptor complex reads specific histone modifications and transfers this information to the splicing apparatus. All this creates the possibility to regulate important cell- and tissue specific alternative splicing patterns. The integrated model makes the complex processes more clearer when all these integrates with each other and the cis- acting regulating elements on the pre-mRNA transcript. Titel Title: Histonmodifieringar och alternativ splicing Histone modifications and alternative splicing Författare Author: Jenny Berggren Nyckelord Keyword: Alternative splicing, Histone modifications, exon, intron, pre-mRNA splicing, splicing regulation, chromatin- adaptor system, integrated model 2 Innehållsförteckning Abstrakt……………………………………………………………………………………..…4 Introduktion………………………………………………………………………………….5-6 Alternativ splicing…………………………………………………………………………..….6 Intronernas uppbyggnad med igenkänningssites……………………………….....................6-7 Typer av alternativ splicing……………………………………………………………….…7-8 Spliceosomens uppbyggnad………………………………………………………………...….8 Splicingmekanismen med de olika komplexen……………………………………………...8-9 Reglering av alternativ splicing………………………………………………………….…9-12 Histonmodifieringar…………………………………………………………………….…12-13 Histonmodifieringar och splicing…………………………………………………………13-14 Systemet med kromatin- splicing adaptor……………………………………………...…14-16 Diskussion…………………………………………………………………………………16-17 Referenser…………………………………………………………………………………18-22 3 Abstrakt Alternativ splicing av pre-mRNA ger upphov till proteindiversitet. Histonmodifieringar kopplas till den alternativa splicingens reglering genom adaptorsystem som överför den epigenetiska informationen direkt till splicingfaktorerna. De cis- agerande RNA- elementen på exoner och introner med tillhörande trans- reglerande splicingfaktorer påverkas därför direkt av specifika histonmodifieringar. En sammankopplande integrerad modell över en rad DNA- baserade processer föreslås. Denna komplexa modell ger en bild av interaktioner och påverkan mellan dessa delar. Kromatin remodellering krävs för bildandet av eukromatin. Nukleosomers placering vid exonrika regioner med specifika modifieringsmönster pekar ut exonerna samt möjliggör inbindning av RNA polymeras II som med sin CTD domän rekryterar både splicing- och modifieringsfaktorer. Transkriptionshastigheten påverkas av nukleosomplaceringen vilket i sin tur påverkar rekrytering av spliceosomens komponenter, andra trans- agerande regulatorer och även pre-mRNA sekvensens sekundärstruktur. Kromatin- adaptorkomplex läser av specifika histonmodifieringar och överför informationen till splicingapparaten. Detta skapar möjlighet till den viktiga cell- och vävnadsspecifika alternativa splicingens reglering. I den integrerade modellen blir komplexiteten tydligare där alla dessa processer interagerar med varandra och de cis- regulatoriska sekvenserna på premRNA transkriptet. 4 Introduktion Det genetiska materialet hos eukaryota celler är linjärt dubbelsträngat DNA. Det packas i formen av kromosomer inne i cellkärnan. Kromosomerna består av kromatinfibrer med nukleosomstrukturer. Dessa fungerar som spolar där ~147 baspar långt DNA lindas runt. Nukleosomerna är uppbyggda av histoner (Kouzarides, 2007). Genom att variera packningsgraden mellan DNA:t och histonerna styrs genuttrycket. Modifiering av histonerna tillsammans med association av ytterligare proteiner leder till strukturförändringar. Både transkription och replikation kräver lösare packning. Tätpackat heterokromatin är vanligen helt inaktivt och transkriberas inte. Lösare packat eukromatin är aktivt då transkriptionsapparaten RNA polymeras II (RNA pol II) kan binda in och transkriptionen av gener initieras (Krebs et al, 2010). En gen är en transkriptionsenhet som består av en mängd olika segment. Exoner är de segment som kodar för protein medan introner är segment som inte kodar för protein. Exonlängden hos däggdjur är generellt mellan 50-250 baspar långa (Wang och Burge, 2008), men är i medel detsamma som antal baspar virade runt nukleosomen (Schwartz et al, 2009). Inom en gen ligger inte exonerna på en rad efter varandra utan är uppdelade med introner mellan sig. Ett visst evolutionärt tryck tros verka för att bibehålla exonlängden på ~140 baspar (Wang och Burge, 2008) till skillnad mot introner som kan vara flera tusen baspar långa. Då nukleosomer verkar vara placerade speciellt vid exonrika DNA- sekvenser kan det tyda på att dess position fungerar som en mekanism för att peka ut var exonerna är men även för att skydda dessa (Schwartz et al, 2009). När en gen transkriberas bildas precursor- mRNA (premRNA). Det är en omogen mRNA- sekvens där även de ickekodande intronernas DNA information skrivits av till RNA. I detta pre-mRNA är > 90 % transkript från introndelarna (Wang och Burge, 2008). För att pre-mRNA:t ska bli ett funktionellt proteinkodande mRNA krävs modifieringar. Intronerna måste tas bort från pre-mRNA transkriptet genom splicing och ändarna trimmas (Krebs et al, 2010). På transkriptets 5′ ände adderas 7-metylguanosin av enzymet guanin-7metyltransferas (MT) så att en 5′ cap bildas. Vanligtvis sker detta tidigt under transkriptionen. En 5′ cap förhindrar att transkriptet bryts ner och är viktig för att RNA pol II ska kunna fortsätta transkriberingen av hela genen. På transkriptets 3′ ände adderas en poly-A svans med upp till 200 adenin- nukleotider. Denna polyadenylation stabiliserar molekylen och krävs för transport genom kärnmembranet. Den fungerar också som igenkänning vid inbindning till ribosomen (Tamarin, 2004). Olika typer av splicing finns. Gemensamt för alla är att introndelarna tas bort och att exondelarna fogas ihop. Utan splicing, capping och polyadenylation skulle aldrig pre-mRNA transkriptet kunna översättas till ett protein av ribosomen. Dess funktion är därför livsnödvändig. Det mänskliga genomet har ~25 000 gener. Om alla dessa uttrycks borde proteinantalet vara detsamma, men så är inte fallet då det mänskliga proteomet består av mer än ~60 000 olika proteiner (Krebs et al, 2010). Proteomets stora diversitet i förhållande till genomet är bland annat ett resultat av alternativ splicing. Studier visar att 92-94 % av de mänskliga generna uppskattas genomgå denna typ av splicing (Pan et al, 2008; Wang et al, 2008). Vid alternativ splicing verkar ett protein- RNA komplex, spliceosomen. Detta komplex kan splica pre-mRNA transkriptet olika och resultatet ger flera olika mRNA- sekvenser från samma gen. Vid translationen fås då protein isoformer. En gen kan alltså ge upphov till flera distinkt olika proteiner, med olika strukturella och funktionella egenskaper (Krebs et al, 2010). 5 Viss pre-mRNA splicing verkar ske cotranskriptionellt (Pandya-Jones et al, 2009). Kromatinstrukturen verkar således inte bara reglera genuttrycket genom packningsgrad utan även påverka hur transkriptet splicas. Därför föreslås att de olika reaktionerna interagerar och påverkar varandra. Proteindiversiteten till följd av alternativ splicing bidrar till ökat antal genprodukter livsviktiga för organismen. Processen har därför stor betydelse för genuttrycket hos en organism. När mekanismen inte fungerar kan resultatet leda till en rad sjukdomar, så som cancer (Berasain et al, 2010 ; Cooper et al, 2009). Mutationer kan ge upphov till felaktig splicing och leda till sjukdomar som Timothys syndrom (Tang et al, 2011). Genom olika sorters reglering styrs den alternativa splicingen och på det viset kan vävnadsoch cellspecifika proteiner fås. Regleringen av alternativ splicing och dess förhållande till histonmodifieringar är ett pågående forskningsområde, och studier pekar på att epigenetiska histonmodifieringar påverkar (Luco et al, 2011). Dessa modifieringar verkar inte bara ha en roll i regleringen av alternativ splicing. De verkar också reglera det vävnadsspecifika genuttrycket. De gener som kodar för komponenter inblandade i cellers medicinupptag (DPGs- drug processing genes) tros regleras bland annat på detta sätt (Klaassen och Lu, 2011). Forskningen är betydelsefull och kan leda till framställning av nya mediciner riktade mot felaktig reglering som leder till sjukdom (Luco et al, 2011). En ökad förståelse av informationen bakom specifika histonmodifieringar är därför av betydelse (Hon et al, 2009). Examensarbetets syfte är att ge en övergripande bild av alternativ splicing och hur histonmodifieringar kan kopplas till dess reglering. Informationen har hämtats från vetenskapliga artiklar och ämnesspecifik litteratur. Syftet uppnås genom grundlig genomgång av alternativ splicing, mekanismer och komponenter som deltar. Fokus ligger på regleringen. Därefter förklaras olika typer av histonmodifieringar och dess funktion. Slutligen knyts kopplingen mellan alternativ splicing och histonmodifieringar ihop. Alternativ splicing Alternativ splicing har stor betydelse för människans utveckling och för fullt fungerande cellprocesser. Det är den största orsaken till ökad proteindiversitet, men det finns andra bidragande orsaker som exempelvis alternativa start sites för transkription, alternativ polyadenylation, och posttranskriptionella modifieringar som fosforylering. Även dessa leder till formandet av proteinisoformer (Nilsen och Graveley, 2010). Vid konstitutiv splicing inkluderas alltid alla exonerna till den mogna mRNA- sekvensen, och i samma ordning som på pre-mRNA sekvensen. Vid alternativ splicing däremot reagerar spliceosomen på regulatoriska signaler vilket leder till att exoner både kan inkluderas och exkluderas (Wahl et al, 2009). De regulatoriska signalerna till de alternativt splicade exonerna kan vara proteiner som bundit till speciella konserverade sekvenser på pre-mRNA transkriptet. Dessa sekvenser finns både på introner och på exoner. De förstärker eller hämmar val av splice sites (Krebs et al, 2010). För att alternativ splicing alls ska vara möjligt krävs reglering samt flexibilitet av spliceosomen att reagera på dessa. Intronernas uppbyggnad med igenkännings sites För att intronerna ska kunna splicas bort från pre-mRNA transkriptet måste det finnas mekanismer för att hitta dem. Inom intronerna finns en mängd olika igenkänningssekvenser. De är cis- agerande och spelar både en igenkännings- och reglerande roll. Även exoner har cis- agerande regulatoriska sekvenser. Figur 1 visar en generell uppbyggnad av ett pre-mRNA transkript. 6 Figur 1. Intronens igenkänningssites. Bindningsställen mellan exonerna och intronen med 5′ splice site, branchpoint och 3′ splice site som följer GU-AG regeln (Freeman et al, 2005). Mellan branchpoint och 3′ splice site finns även en polypyrimidinsekvens (Krebs et al, 2010) Varje intron binder till två olika exoner. Dessa bindningar är detsamma som splice sites då det är dessa som ska brytas så att intronen kan lösgöras från pre-mRNA:t. Därefter kan exonerna fogas ihop. Dessa bindningar är konserverade konsensussekvenser och följer en GU-AG regel. Detta för att de flesta intronernas 5′ splice site har dinukleotiden GU och dess 3′ splice site har dinukleotiden AG. Ytterligare konserverade sekvenser inom introner förekommer och en sådan viktig igenkänningssekvens är branchpointsekvensen (BPS). Den ligger 18 till 40 nukleotider uppströms om 3′ splice site. Mellan branchpoint sekvensen och 3′ splice site finns en polypyrimidinsekvens (PPT, polypyrimidine tract) som också spelar en viktig roll. Hela splicingprocessen är beroende av igenkänning av de två splice ställena. De kan ligga långt ifrån varandra då många introner är väldigt långa. Inbindning av trans-agerande regulatorer till dessa cis- agerande sekvenser fungerar som en viktig mekanism i den proteinmedierade alternativa splicingens reglering (Krebs et al, 2010). Typer av alternativ splicing För att uppnå den enorma mängden proteinisoformer från de ~25000 generna används olika varianter av alternativ splicing. En enda gen kan ge upphov till allt emellan två och flera tusen proteinisoformer. Figur 2 visar fyra typer av mekanismer vid alternativ splicing. Figur 2. Bilden visar fyra olika typer av alternativ splicing med pre-mRNA sekvensen i mitten och de olika mRNAsekvenserna till följd av splicingmekanismerna på var sida om denna. Exonerna är de olikfärgade fyrkanterna och intronerna de blå strecken emellan dem. (a) visar alternativt val av 5′ splice site på mittersta pre-mRNA sekvensen och de två möjliga mRNA- sekvenserna till följd av de två olika valen, (b) visar samma som (a) fast med alternativt val av 3′ splice site, (c) visar casette- exon inkludering(mRNA till höger) och casette- exon exkludering(mRNA till vänster, (d) visar intron retention där den rosa fyrkanten representerar en intron som tas med till det högra mRNA:t om pre-mRNA sekvensen (Nilsen och Graveley., 2010). I den första och andra typen (figur 2, a och b) påverkar alternativt val av 5′ splice site eller 3′ splice site om exonet inkluderas eller exkluderas i mRNA sekvensen. Den tredje typen (figur 2, c) visar casette- exon inkludering på den högra mRNA sekvensen och casette exon exkludering på den vänstra mRNA sekvensen. I den fjärde typen (figur 2, d) visas intron retention där intronet inte splicas ut utan behålls i mRNA:t till höger (Nilsen och Graveley, 2010). Ett pre-mRNA kan dessutom genomgå splicing flera gånger och vilken typ av splicing som väljs beror på regleringen. Bland annat påverkar interaktionerna med de trans-agerande splicingfaktorerna tillsammans med spliceosomens förmåga att reagera på dessa. Dessa 7 grundprocesser ligger bakom möjliggörandet av den stora diversitet med flera isoformer av ett protein från samma pre-mRNA (Krebs et al., 2010). Spliceosomens uppbyggnad Den trans- agerande splicingfaktorn spliceosomen, är som tidigare nämnts ett RNAproteinkomplex. Den är uppbyggd av fem stycken small nuclear ribonucleoproteiner (snRNPs), proteiner och splicingfaktorer (Krebs et al, 2010). Totalt samverkar ~ 145 proteiner vilket gör spliceosomen till ett av det mest komplexa cellmaskinerierna (Zhou et al, 2002). De fem olika snRNPs betecknas U1, U2, U4, U5 och U6. Var och en av dessa består av en eller flera proteiner, däribland Sm- proteiner tillsammans med en liten RNA molekyl (Wahl et al, 2009). RNA molekylerna är mellan 100-215 baser långa och har sekvenser som är komplementära till antingen exonerna, intronerna eller till de andra snRNPs RNA (Krebs et al, 2010). Splicingmekanismen med de olika komplexen Under splicingprocessen interagerar spliceosomens olika komponenter med varandra, andra trans- agerande regulatorer och de cis- agerande RNA sekvenserna på pre-mRNA:t. En rad komplex bildas under processen och är nödvändiga för att de biokemiska reaktionerna ska ske. Dessa består av att fosfodiesterbindningen vid 5′ splice site bryts genom att en adenosin i branchpointsekvensen attackerar fosfodiesterbindningen med sin hydroxylgrupp. Då bildas på exon 1 en fri exonände medan intronen och exon 2 bildar en lariat struktur då den nu fria intronänden binds till branchpoint. Hydroxyländan på exon 1 gör därefter en nukleofil attack på 3′ splice site så även denna fosfodiesterbidning bryts. Därefter ligeras exonerna ihop och intronen lösgörs. Hela processen medieras av spliceosomen (Wahl et al, 2009) och figur 3 visar en förenklad bild av hur spliceosomen sätts ihop och verkar. Figur 3. Uppbyggnaden av spliceosomen med U1, U2, U4, U5 och U6 snRNPs och förenklad bild över vilka olika komplex som bildas samt vad de olika snRNPs gör. (Max Planck Institute forBiophysical Chemistry, 2008) I figur 3 saknas ett tidigt komplex, E komplexet. Formandet av det komplexet kräver att de två splice ställena känns igen. Igenkänning kan ske på två sätt, antingen via intron- eller exondefinition. När det handlar om gener med små introner (~100-300 nukleotider långa) gäller vanligen introndefinition. Då känner U1 snRNP igen 5′ splice site och binder in dit. Ett branchpointbindande protein (BBP, kallas även SF1, Splicing Factor 1) binder in till branchpointsekvensen. Heterodimeren U2AF auxiliary faktor (U2AF65/35) binder till polypyrimidinsekvensen mellan branchpoint och AG dinukleotiden. Då hittar U2 snRNP rätt inbindningsställe och binder till pre-mRNA sekvensen. E komplexet övergår då till A komplexet vilket visas som det första komplexet i figur 3. Fram till E komplexet är reaktionerna oberoende av ATP. Men för att A komplexet ska bildas krävs ATP (Chen et al, 2009). De två splice sites känns då igen samtidigt och interaktionerna sker över intronerna. Hos exoner med längre och fler introner mellan sig sker igenkänningen istället med exondefinition. Detta för att korrekt igenkänning av splice sites ska vara möjlig. De långa intronerna kan nämligen ha en mängd sekvenser inom sig snarlika de korrekta splice ställena. 8 Exonerna är däremot normalt små i jämförelse med de flesta introner. Vid exondefinition binds U2AF heterodimeren till 3′ splice site och U1 snRNP till 5′ splice site nedströms det exonet. Inbindningen stabiliseras av SR proteiner som binder till mellan 3′ splice site och 5′ splice site inom exonet. Därefter sker en skiftning av komplexen så komplexet vid 5′ splice site länkas till nästa 3′ splice site nedströms intronet. Och komplexet vid 3′ splice site länkas till nästa 5′ splice site uppströms det intronet. Med SR proteiners hjälp länkas då de korrekta splice ställena till varandra (Krebs et al, 2010). Oavsett om A komplexet formats efter intron- eller exondefinition har de olika komponenterna en viss funktion. När U1 snRNP binder in till intronens 5′ ände märks detta splice site ut så att hela splicingprocessen kan startas. Denna reaktion stabiliseras av splicingfaktorer, bland annat de tidigare nämnda U2AF, BBP och SR proteiner. Dessa proteiner behövs för att öka stabiliteten av basparningen emellan U1 snRNP och 5′ splice site (Wahl et al, 2009). SR proteinerna formar en brygga mellan de RNA- bindande komplexen på splice ställena. Tillsammans bildar de E-komplexet. Först när alla dessa bundit in kan U2 snRNP hitta och binda till branchpoint på intronen. Bildandet av komplex A kräver även det proteiner för ökad stabilitet. Detta står SF3a, SF3b (Splicing Factor 3a och 3b), SC35 och U2AF för. Därefter formas ett komplex av U4, U5 och U6 snRNPs (U4.6/U5 tri-snRNPs). Även detta binder till pre-mRNA:t och då bildas B1 komplexet (se figur 3). U4 är bunden till U6 vilket inaktiverar U6, men vid inbindningen frisläpps U4 och lösgörs från tri-snRNP så U6 aktiveras och binder till intronens branchpoint. Därefter tränger U6 undan U1 som också frisläpps. B1 komplexet övergår då till B2 komplexet och den katalytiska reaktionen startar. U6 har även en komplementär region till U2 så de binds ihop och lariat form bildas när 5′ splice site klyvs enligt figur 3. Detta kallas för komplex C1. U5 har komplementära regioner till båda exonerna på var sida om intronen. När 3′ splice site klyvs och exonerna ligeras ihop övergår C1-komplexet till C2-komplexet. Hela splicingprocessen fullbordas då lariaten lösgörs. Denna samverkan av spliceosomens olika komponenter tillsammans med de cis- agerande sekvenserna och de trans- agerande splicingfaktorerna leder till möjliggörandet av de biokemiska splicingreaktionerna under den alternativa splicingen (Krebs et al, 2010). Reglering av alternativ splicing En rad olika regleringsmekanismer i den alternativa splicingen är kända. De alternativt splicade exonerna är ofta associerade med svaga splice sites (Black, 2003). De har däremot de konserverade cis- agerande sekvenserna omkring sig och dessa fungerar som splicingregulatoriska element. CTD domänen (carboxy terminal domain) hos RNA pol II har också en roll i regleringen, men även transkriptionshastigheten. De regulatoriska RNAelementen på pre-mRNA:t kan hämma eller förstärka val av splice site genom inbindning av de trans-agerande regulatorerna. Det finns fyra olika typer av dessa splicingregulatoriska element, och de är lokaliserade som tidigare nämnts både på introner och exoner. Dessa är exonic splicing enhancers (ESEs), exonic splicing silencers (ESSs), intronic splicing enhancers (ISEs) och intronic splicing silencers (ISSs) (se figur 4) (Krebs et al, 2010). 9 Figur 4. De fyra olika cis- regulatoriska splicing elementen; ISEs, ISSs, ESEs och ESSs. Det mittersta exonet kan antingen inkluderas eller exkluderas i mRNA sekvensen beroende på de trans-agerande splicingfaktorerna (Wang och Burge, 2011) ESEs och ESSs är lokaliserade på exoner (se figur 4). ESEs förstärker splice site val genom inbindning av positiva splicingregulatorer, där de vanligaste är SR proteiner. Inbindningen sker genom SR proteinets N- terminal som har en RNA- bindande domän. SR proteiner interagerar även med branchpoint genom sin arginin- och serinrika RS domän C- terminalt (Graveley, 2000). När den interagerar med branchpoint förstärks splice site valet genom att underlätta formandet av pre- spliceosomen, och detta kräver en fullt fungerande branchpointsekvens (Shen et al, 2004). Det finns ett tiotal medlemmar i SR familjen och de mest utforskade är SF2/ASF (Splicing Factor 2/ Alternative Splicing Factor) och SC35. För att A komplexet ska bildas krävs proteinet SC35. Detta protein interagerar även med andra splicingkomponenter bundna till 5′- och 3′ splice sites under formandet av spliceosomen (Fu och Maniatis, 1992). SF2/ASF förstärker val av 5′ splice site och förändringar i dess koncentration eller aktivitet påverkar val av splice site (Ge and Manley, 1990). ESSs är också lokaliserade på exoner men hämmar istället val av närmaste splice site genom inbindning av andra splicingregulatorer. Exempel på de splicingregulatorerna är hnRNP A och B (heterogeneous nuclear RNPs, RNA-bindande protein) vilket visas i figur 4. ISEs och ISSs är de cis-agerande elementen motsvarande de på exonerna (Chen et al, 2009), (se figur 4). Splicingregulatorer till ISEs kan vara hnRNP F, hnRNP H, NOVA 1 och 2 (neurooncological ventral antigen), och FOX 1 och 2. NOVA proteinerna kan verka både förstärkande och hämmande. De förstärker exoninkludering om de binds till ISEs nedströms om exonet (Ule et al, 2006), men hämmande om de binder till ISSs uppströms exonet (Krebs et al, 2010). FOX regulatorerna tillhör FOX familjen med RNA- bindande proteiner (Yeo et al, 2009). Vid inbindning till ISEs nedströms exoner verkar de förstärka exoninkludering precis som NOVA proteinerna. Splicingregulatorn hnRNP H förstärker exoninkludering vid inbindning till ISEs men hämmar exoninkludering vid inbindning till ESSs (Mauger et al, 2008). De flesta olika splicingregulatorer används i alla celltyper. Vävnadsspecifik splicing uppnås vanligtvis inte genom vävnadsspecifika splicingregulatorer, även om det förekommer. NOVA proteinerna är neuronspecifika (Ule et al, 2006) och FOX proteinerna är specifika för neural- och muskelceller. FOX 2 är till exempel en stamcellsregulator som är viktig för att dessa celler hos embryon överlever (Yeo et al, 2009). Då genuttrycket av splicingregulatorerna är vävnadsspecifikt fås olika koncentrationer av regulatorerna i olika vävnader och de regulatorer med högst koncentration är de som binder in till de cis- regulatoriska elementen (Hanamura et al, 1998). För regleringen av alternativ splicing finns även kopplingar till transkriptionsapparaten. Denna har visat sig påverka en rad andra processer förutom det uppenbara att transkribera 10 DNA till en pre-mRNA sekvens. RNA polymerasets CTD domän modifieras genom fosforylering (se figur 5) och det finns flera olika ställen på denna domän som kan fosforyleras. Detta medieras av olika enzymer under olika stadier av transkriptionen. För att RNA pol II ska kunna bryta bindningen till transkriptionsfaktorer på promotorn så att elongeringen kan initieras krävs det att CTD domänen fosforyleras. Modifieringarna påverkar en rad andra processer förutom de nödvändiga för transkriptionen. En av fosforyleringarna aktiverar CTD att rekrytera mRNA modifieringsfaktorer (Buratowski, 2003). Pre-mRNA transkriptets 5′ cap binds på tack vare att guanin-7-metyltransferas binder till fosforylerad CTD (Krebs et al, 2010). Komplexet som står för polyadenylationen interagerar också med CTD (se figur 5). Figur 5 . Under transkriptionen interagerar RNA Pol II komplexet (grön) med det CAP-bindande komplexet (orange), splicingkomplexet (röd) och det poly-A bindande komplexet (blå) via sin fosforylerade CTD domän(Luco et al., 2011). Figuren visar även hur nukleosomerna ligger på rad efter varandra likt ett pärlband Kopplingen mellan transkription och splicing sker bland annat genom CTD domänen (Yuryev et al, 1996). Även splicingfaktorer som SCAFs proteinerna (SR liknande proteiner med RS domän) binds till den fosforylerade CTD domänen (Misteli och Spector, 1999). Inbindningen har därför en regulatorisk roll för den alternativa splicingen (Yuryev et al, 1996). Bland annat står SR proteinerna för sammankopplingen mellan transkriptions- och splicingapparaten. Interaktion sker då mellan SR proteinerna och U1snRNP (Das et al, 2007). Dessa komponenter verkar som bekant tidigt under spliceosomens formande när U1 snRNP känner igen och binder till intronens 5′ splice site för att märka ut denna. Förutom att sammankoppla transkriptions- och splicingapparaterna stabiliserar SR proteinerna även reaktionen (Wahl et al, 2009). Inbindningen av U1 snRNP till RNA Pol II via SR proteiner verkar placera U1 snRNP i korrekt position gentemot pre-mRNA transkriptets 5′ splice site och då skapas effektivt formande av spliceosomen (Das et al, 2007). Elongerings hastigheten verkar också påverka formandet av spliceosomen (Muñoz et al, 2010), men omfattningen av transkriptionhastighetens roll i den alternativa splicingens reglering är inte helt utredd. Flera studier pekar dock på att en kinetisk koppling finns (Luco et al, 2010). Bland annat påverkas alternativt val av 5′ splice site för att längden på pre-mRNA strängens RNA- loop (en sekundärstruktur) avgörs av transkriptionshastigheten (Eperon et al, 1988). Det är även så att vid långsam hastighet kan splicingfaktorerna binda till intronet uppströms exonet innan intronet nedströms ens blivit färdigsyntetiserat. Detta förstärker exoninkludering genom att spliceosomen då binder in till intronet (de la Mata et al, 2003). Däremot verkar det inte betyda att det är den intronen som splicas bort först, utan bara att det påverkar val av splice site (de la Mata et al, 2010). Vävnadsspecifik splicing tros därför även uppnås genom olika transkriptionshastigheter. De nu beskrivna mekanismerna i den alternativa splicingens reglering räcker inte till som 11 förklaringsmodell. Utöver de cis- agerande regulatoriska RNA- sekvenserna, de transagerande splicingregulatorerna, CTD domänen och transkriptionshastighet behövs ytterligare mekanismer. Den redan komplexa splicingprocessen är långt ifrån helt kartlagd. Det har nu upptäckts att även histonmodifieringar verkar reglerande (Luco et al, 2010). Histonmodifieringar Nukleosomerna är uppbyggda av histonerna H1, H2A, H2B, H3 och H4, samt negativt laddat DNA. Histonerna är en grupp arginin- och lysinrika proteiner vilket gör dem speciellt lämpade att binda det negativt laddade DNA:t (Krebs et al, 2010). Nukleosomen består av en histonkärna av H2A, H2B, H3 och H4 med uppbyggnaden som en oktamer, och det är runt denna DNA:t är lindat (Kouzarides, 2007). Nukleosomerna är placerade efter varandra likt ett pärlband med länk DNA mellan (se figur 5), och histon H1 tros vara associerat till detta länk DNA. Packningsgraden mellan DNA och histonerna påverkar genuttrycket och kovalenta modifieringar på kärnhistonernas svansar reglerar hur hårt bundet DNA:t är (Krebs et al, 2010). Dessa svansar med sina amino- och karboxyterminaler sticker ut från nukleosomkärnan, och ett flertal ställen på varje svans kan vara föremål för modifiering. Beroende på typ av modifiering uppnås olika strukturförändringar hos kromatin. Acetylering, metylering och fosforylering är exempel på övergående histonmodifieringar (Kouzarides, 2007), och dessa förändringar används för att ändra laddningar. Histonernas funktionella egenskaper kan på det viset anpassas efter behov i cellen. Att tillföra negativa laddningar för att minska DNA packningen är nödvändigt vid transkription såväl som vid replikation, och acetylering och fosforylering tillför negativa laddningar. Det som händer är att den positiva laddningen på histonens aminosyra neutraliseras och då försvagas bindningen mellan histonen och det inbundna DNA:t så transkriptionsapparaten kan binda in till DNA:t. Acetylering är därför associerat med aktiva gener. Metylering förstärker bindningen mellan histonen och DNA:t och är därför associerat med inaktiva gener. Strukturförändringar hos histonerna på grund av modifieringar kan påverka ett närliggande site som vanligtvis också är föremål för modifieringar genom kooperation så att det närliggande sitet då inhiberas mot egna modifieringar. Strukturförändringarna kan även skapa bindningsställen för icke- histon proteiner (Krebs et al, 2010). Exempel på aminosyror att modifiera på histonsvansarna är lysin, arginin, serin och treonin. Lysin kan både metyleras och acetyleras, och kan dessutom bli både mono, di och trimetylerad. Arginin kan bli mono- och dimetylerad. Serin och treonin kan bli fosforylerade. Icke- histon proteinerna har speciella proteindomäner och kända sådana är bromo- och chromodomänerna. Specifikt modifierade histonsvansar har visat sig binda till sig dessa. Bland annat känner bromodomänen igen acetylerade lysin som den binds till och icke- histon proteinet interagerar på det viset med transkriptionsapparaten, men också med vissa av de enzymer som står för acetyleringen. Chromodomänen känner igen metylerade lysin och arginin och dessa kan även skilja på graden av metylering (Krebs et al, 2010). De enzymatiska modifieringsreaktionerna utförs bland annat av ATP-beroende kromatin remodellerare. Det finns tre klasser av dessa proteinkomplex. Ett av dessa är SWI/SNF (mating-type switch/sucrose nonfermenting) där SWI2/SNF2 som tillhör SF2 familjen (splicingfaktorer) har en bromodomän (Martens och Winston, 2003). SWI/SNF komplexen av kromatin remodellerare krävs i många fall för att kromatinet ska modifieras till sin aktiva form. Annars kan inte transkriptionsfaktorer binda in till promotorn på DNA:t (Batsché et al, 2006). Denna remodellerare har även en funktion att reglera transkriptionshastigheten (Tyagi et al, 2009). Andra enzym involverade i modifieringarna är histon acetyltransferas (HATs) så som STAGA- komplexet hos däggdjur (homolog till SAGA- komplexet hos jäst, 12 Saccharomyces cerevisiae) (Martinez et al, 2001) och deacetyltransferas (HDACs) (Hnilicová et al, 2011). Proteinet GCN5 ingår i STAGA- komplexet och acetylerar H3 och H2B som gör att transkriptionsfaktorer kan binda in till DNA:t (Gunderson och Johnson, 2009). Alla de olika histonmodifieringarna har skapat ”histon-kod hypotesen” och ”kromatin nätverks signaleringsmodellen” där alla modifieringar tros ha en specifik funktion (Schreiber och Bernstein, 2002). Funktionerna kan vara att interagera med olika proteiner och på det viset reglera olika DNA-baserade processer (Kutney et al, 2004). Faktiskt rekryterar CTD domänen hos RNA pol II även histonmodifieringsfaktorer, som enzymet vilket trimetylerar lysin 36 på H3 (H3K36me3) (Li et al, 2007). Histonmodifieringar och splicing En allt tydligare bild framträder av de cellulära processernas komplexitet, där många av de olika delarna är nära associerade och har stark påverkan på varandra. Det finns ingen enkel beskrivning av histonmodifieringens roll i regleringen av alternativ splicing. Tydligt framgår dock att histonerna med DNA:t virat runt sig samverkar med RNA- splicingen och kopplas samman med bland annat RNA pol II, som med sin CTD domän rekryterar både splicingfaktorer (Scwartz et al, 2009) och faktorer till histonmodifieringar (Li et al, 2007). Vissa faktorer som är associerade till RNA pol II verkar alltså både som kromatin remodellerare och splicingregulatorer (Batsché et al, 2006). Enzymerna ansvariga för histonmodifieringarna kan i flera fall kopplas till splicingen genom deras direkta interaktion med splicingfaktorer (Luco et al, 2011). Histon metyltransferaset CARM1 interagerar med U1 snRNPs (Cheng et al., 2007). Kromatin remodellerare från SWI/SNF familjen interagerar med U5 snRNPs (Batsche et al, 2006). STAGA- komplexet med HAT aktivitet interagerar med U2snRNPs och U2snRNP binder i sin tur in till intronens branchpoint. STAGA är därför involverad i den cotranskriptionella pre-mRNA splicingen, och det kan vara så att detta är ett sätt att rekrytera splicingmaskineriet till aktivt transkriberade gener (Martinez et al, 2001). Kromatinets struktur leder då till rekryteringen och är helt oberoende av transkriptionshastigheten. Det talar för en direkt funktion i rekryteringen av splicingmaskineriet (Gunderson och Johnson, 2009). Aktivt eukromatin uppnås som beskrivits genom acetylering, ju lösare packning desto högre transkriptionshastighet. Det kan därför vara så att vävnadsspecifik splicing till viss del regleras av vävnadsspecifika skillnader i transkriptionshastighet beroende av histonmodifieringar, och till mindre del av vävnadsspecifika koncentrationer av de trans-agerande regulatorerna (Nilsen och Graveley, 2010). Nukleosomernas placering samt metylering och acetylering av histonerna spelar en viktig roll för transkriptionen (Li et al, 2007) och transkriptionen i sin tur påverkar även splicingen (Luco et al, 2010). Exoner med svaga splice sites eller exoner som flankeras av långa introner har som tidigare nämnts högre koncentration av nukleosomer (Spies et al, 2009), och alternativt splicade exoner som inkluderas är till större del utmarkerade av nukleosomer (Schwartz et al, 2009). Då nukleosomer påverkar RNA pol II genom att bland annat bromsa transkriptionshastigheten kan splicingeffektiviteten moduleras (Luco et al, 2010). RNA pol II uppvisar högre koncentration vid exoner än introner till följd av nukleosomernas placering (Schwartz et al, 2009). Den ökade koncentrationen vid alternativt splicade exoner leder till effektivare association av både histonmodifierare och splicingfaktorer. Hur histonmodifieringarna, nukleosomplaceringarna, RNA pol II:s aktivitet och splicingen kan kopplas ihop är inte fullt kartlagt. Även om vissa histonmodifieringar som trimetylerad H3 (H3K36me3) verkar ha en roll i splicingregleringen (Hon et al, 2009; Luco et al, 2010). I figur 6 visas denna trimetylering tillsammans med H3K79me2, och dessa modifieringar är vanliga 13 på nukleosomer vid exoner i aktivt transkriberade gener (Guccione et al, 2006). Figur 6. Bilden visar hur olika histonmodifieringar kan vara lokaliserade på gener. Exoner i aktivt transkriberade gener uppvisar modifieringarna H3K36me3 och H3K79me2 (Guccione et al, 2006) i jämförelse med introner och exoner i inaktiva gener som uppvisar ändra modifieringsmönster(H3K9me2 och H3K9me3) (Zhou et al, 2011). Ytterligare exempel på vanliga modifieringar som markerar ut exoner är H3K4me3 och H3K27me2 (Dhami et al, 2010; Spies et al, 2009) och just aktiva geners start sekvens, dess 5′ ände är associerad med H3K4me3 och denna trimetylering är även associerad med hög transkriptionshastighet (Bernstein et al, 2005). Modifieringarna är alltså inte slumpmässiga utan högst specifika. Det har även visat sig att DNA- metyleringsmönstret korrelerar med histonernas (Meissner et al, 2008) samt att DNA- metyleringen också uppvisar hög specificitet som tydligt markerar ut exoner och kan därför också ha en roll vid exondefinition (Chodavarapau et al, 2010). Systemet med kromatin- splicing adaptor I ett antal gener har det visat sig att histonmodifieringar direkt påverkar alternativ splicing, till exempel genom att olika modifieringar leder till olika splice site val. En adaptormodell föreslås där ett komplex känner igen modifieringar på histonerna och binder in till dessa, och att detta komplex sedan rekryterar splicingfaktorerna. Histonmodifieringarna avgör inte ensamt splicing utgången utan verkar reglerande. Ett antal av denna sorts kromatin- splicing adaptorsystem finns troligtvis hos däggdjur (Luco et al, 2010). Viss alternativ splicing är beroende av splicingfaktorn PTB, ett polypyrimidinbindande protein (polypyrimidin tract binding protein). Det verkar vara just denna typ av alternativ splicing som regleras med ett kromatin- splicing adaptorsystem. Kända alternativt splicade gener kan kopplas ihop med specifika histonmodifieringar. En sådan gen är FGFR2 (human fibroblast growth factor receptor 2) som uppvisar vävnadsspecifik splicing av exon IIIb och IIIc (Luco et al, 2010). Genen kodar för plasma membranreceptorer och splicingen resulterar i isoformer av denna i olika celltyper (Wagner och Garcia-Blanco, 2002). I prostata epitelceller (PNT2-prostate normal epithelium cells 2) inkluderas vanligtvis exon IIIb. I mesenkymala stamceller (hMSCs-human mesenchymal stem cells) däremot exkluderas IIIb medan exon IIIc vanligtvis inkluderas (Luco et al, 2010). Dess reglering är beroende av PTB där proteinet fungerar som en repressor vid inbindning till antingen ett ISSs element uppströms exonet eller till ett annat PTB bindande element nedströms, vilket leder till att exon IIIb exkluderas (Carsten et al, 2000). Med tekniken kromatin immunoprecipitation kan protein- DNA interaktioner utvärderas. Med denna teknik har man gjort jämförande studier för att se kopplingar mellan specifika histonmodifieringar vid den alternativt splicade genen i dessa prostata- och stamceller. Histonmodifieringarna mellan prostata- och stamcellerna jämfördes med varandra. Resultaten visade att histonmodifieringarna H3K36me3 och H3K4me1 var förstärkta medan H3K27me3, H3K4me3 och H3K9me1 var reducerade över FGFR2 genen i stamcellerna där exon IIIb exkluderades i jämförelse med prostatacellerna. Skillnader fanns över hela genen men dessa var de största, samt att de var placerade just vid de alternativt splicade regionerna. Skillnader mellan andra PTB- beroende alternativt splicade exoner i andra gener har utvärderats och dessa uppvisar ett liknande mönster som de mellan prostata- 14 och stamcellerna. Att dessa histonmodifieringar med samma splicingmönster återfinns hos flera olika PTB- beroende exoner pekar på en korrelation. PTB- oberoende och konstitutivt splicade exoner uppvisar inte detta mönster (Luco et al, 2010). Dessa studier banade väg för upptäckten av kromatin- splicing adaptorsystemet. Adaptorproteinet, MRG15 (MORF related gene 15) är kromatinbindande och binder specifikt till H3K36me3. Vid inbindning till den trimetylerade H3 moduleras splice site val för de PTBberoende alternativt splicade exonerna (Luco et al, 2010). Metyltransferaset SET2 är det som står för metyleringen och dess aktivitet är beroende av MRG15. De exoner med svagare bindningssites för PTB verkar generellt vara mer beroende av adaptor- PTB komplexet (Luco et al, 2010). Tillsammans med de RNA- bindande elementen förstärker då de epigenetiska modifieringarna effekten genom adaptorsystemet (Luco et al, 2011). Den alternativa splicingen av dessa exoner regleras därför genom histonmodifieringarna (Luco et al, 2010). Figur 7 visar hur adaptorsystemet fungerar. Histonmodifieringen H3K36me3 leder till inbindning av det kromatinbindande proteinet MRG15 som interagerar med splicing regulatorn PTB. Denna hamnar då i rätt position för att binda in till de cis-agerande elementen på pre-mRNA transkriptet. Komplexet överför alltså den epigenetiska informationen till premRNA modifieringsapparaten och styr på det viset splicingresultatet (Luco et al, 2010). Figur 7. Kromatin- splicing adaptorsystemet. Det kromatin bindande MRG15 (grön) associerar till specifika histonmodifieringar (H3K36me3) vilket rekryterar splicingfaktorn PTB (orange) som binder in till de regulatoriska elementen på pre-mRNA transkriptet. PTB fungerar då som en repressor och exonerna i detta system exkluderas. Histonmodifieringen H3K36me3 har i detta system en direkt roll i splice site val. (Luco et al, 2010). Ett annat kromatin- adaptor system verkar genom adaptorproteinet CHD1 (chromoATPase/helicase- DNA binding domain 1) och principen är densamma som med MRG15 (se figur 7). CHD1 är ett icke- histon protein (ingår i ett större komplex) med HAT aktivitet och en chromodomän (Pray-Grant et al, 2005). Denna domän binder in till en annan trimetylering på H3, H3K4me3. CDH1 rekryterar därefter U2snRNP genom dess association med SF3a (Sims et al, 2007). SF3a har en roll att stabilisera A komplexet vilket tidigare beskrevs (Krebs et al, 2010). Detta system har därför en roll i den tidiga fasen av spliceosomens formande (Sims et al, 2007). En annan komponent av komplexet vilket CHD1 ingår i är GCN5 (PrayGrant et al, 2005) och detta protein acetylerar H3 och H2B (Gunderson och Johnson, 2009). Acetylerad H3 och aktiviteten hos GCN5 är en viktig factor för att rekrytera U2 snRNP till branchpoint (Gunderson och Johnson, 2009). Ytterligare exempel är genom adaptorproteinet HP1 (heterochromatin protein1) som binder till H3K9me3 och associerar sedan med splicingfaktorer så som ASF/SF2 (Loomis et al, 2009). Det finns flera olika varianter av HP1 proteiner. Förutom att fungera som ett adaptorprotein verkar varianten HP1γ sakta ner elongeringshastigheten som leder till förändringar på pre-mRNA sekvensens sekundärstruktur, vilket också förstärker rekryteringen av spliceosomen (Saint-André, 2011). Som tidigare nämnts finns troligtvis ännu fler varianter av dessa kromatin- splicing adaptorsystem för andra alternativt splicade exoner. Exempel på detta är SR protein och 15 ASF/SF2 som associerar med H3 och reglering av denna inbindning styrs av histonmodifieringar, i detta fall fosforylering (Loomis et al, 2009). Vilket är ett ytterligare exempel på direkta interaktioner mellan splicingfaktorer och histoner som regleras genom histonmodifieringar (Luco et al, 2010). Dessa system verkar som beskrivet via olika varianter av komplex vilka bildar en länk mellan histonerna och splicingfaktorerna. Splicingfaktorerna interagerar med de cis- agerande RNA sekvenserna på pre-mRNA transkriptet. Alla olika komponenter inblandade i den alternativa splicingens reglering verkar samverka och kan kopplas ihop genom ett flertal mekanismer. Därför föreslås en integrerad modell över splicingen, transkriptionen och kromatinstrukturen (Luco et al, 2011). Figur 8 pekar på processernas möjliga kopplingar. Figur 8. Integrerad modell av den alternativa splicingens reglering. Kromatin remodellerare krävs för formandet av eukromatin, så att transkriptionsfaktorer ska kunna binda in. Nukleosomernas placering pekar ut exonerna och saktar ner RNA pol II. RNA sekundärstrukturen påverkas av hastigheten och modulerar val av 5′ splice site. RNA pol II CTD domän rekryterar premRNA modifieringsfaktorer och splicingfaktorer. Kromatin- adaptorkomplex binder in till specifika histonmodifieringar och kan då verka och rekrytera splicingfaktorer. Dessa interagerar med varandra och de cis- agerande sekvenserna på pre-mRNA transkriptet (Luco et al, 2011). Diskussion Examensarbetets syfte var att ge en övergripande bild av alternativ splicing och hur histonmodifieringar kan kopplas till dess reglering. De epigenetiska histonmodifieringarna verkar fungera reglerande och har en betydande roll för formandet av proteinisoformer. Det står klart att splicingen av pre-mRNA transkriptet ingår i en extremt komplex process där en mängd DNA relaterade mekanismer samverkar och påverkar varandra vilka sammanfattas i den integrerade modellen som visas i figur 8 (Luco et al, 2011). Denna modell sammankopplar alla de tidigare beskrivna processerna. För att transkriptionsapparaten alls ska kunna binda in till promotorn på DNA:t krävs kromatin remodellerare för bildandet av aktivt eukromatin (Krebs et al, 2010). Nukleosomernas placering vid exonrika regioner (Schwartz et al, 2009) med specifikt modifieringsmönster pekar ut exonerna och saktar ner RNA pol II. Minskad hastighet påverkar rekryteringen av spliceosomens komponenter och andra transagerande regulatorer samt att pre-mRNA sekvensens sekundärstruktur påverkas (Luco et al, 2011). Den fosforylerade CTD domänen på RNA pol II spelar en viktig roll för rekryteringen av splicing- och modifieringsfaktorer (Batsché et al, 2006). Kromatin- adaptorkomplex fungerar som en brygga mellan histonerna och pre-mRNA sekvensen och överför den epigenetiska informationen. De olika processerna interagerar med varandra och de cisagerande regulatoriska sekvenserna på pre-mRNA:t (Luco et al, 2011). Kromatinstrukturen påverkar således en rad processer och genom denna sammanfattande modell framgår det att dess modifieringar påverkar splicingen både direkt och indirekt. Koppling finns alltså mellan histonmodifieringar och transkriptionsapparaten samt att splicing valet verkar tas cotranskriptionellt (Saint-André et al, 2011). Histonmodifieringarna har alltså direkt påverkan på splicingen genom kromatin- splicing adaptorsystem (Loomis et al, 2009; Luco et al, 2010; Sims et al, 2007). Detta tillsammans med det faktum att de alternativt splicade exonerna är utmärkta av nukleosomer (Schwartz et al, 2009) som verkar följa ett generellt modifieringsmönster pekar på en direkt roll för epigenetiska histonmodifieringar i regleringen 16 av alternativ splicing (Luco et al, 2010). Vävnadsspecifik alternativ splicing kan uppnås genom koncentrationsskillnader av de trans- agerande regulatorerna, skillnader i transkriptionshastighet men också troligtvis som följd av histonmodifieringar (Nilsen och Graveley, 2010) som direkt påverkar genuttrycks variation av regulatorerna. Genuttrycket av DPGs uppnås bland annat genom vävnadsspecifika modifieringar och används som regleringsmekanism (Klaassen och Lu, 2011). Korrelation mellan alternativt splicade exoner och histon- metyleringsmönster är områden som behöver utredas. Alternativ splicing ökar på proteomets storlek utan ökning av genomet. På grund av processens komplexitet finns det också en uppsjö av delar i den som kan gå fel. Processen är livsviktig för fullt fungerande cellprocesser och felaktig reglering kan leda till sjukdomar (Berasain et al, 2010 ; Cooper et al, 2009; Tang et al, 2011; Wang och Burge, 2008). Mutationer på gener som undergår alternativ splicing, mutationer i gener som kodar för de trans- agerande regulatorerna, HATs eller för spliceosomens komponenter är exempel som kan vara sjukdomsframkallande då de ofta leder till felreglering. Ett flertal studier inom området har gjorts men för en klarare bild och ökad förståelse är det viktigt med fortsatt forskning inom ämnet. 17 Referenser Batsché, E., Yaniv, M. & Muchardt, C. (2006). The human SWI/SNF subunit Brm is a regulator of alternative splicing. Nat. Struct. Biol. 13, 22-29. Berasain, C., Goñi, S., Castillo, J., Latasa, M.U., Prieto, J. & Ávila, M.A. (2010). Impairment of pre-mRNA splicing in liver disease: Mechanisms and consequences. W. J. Gastro. 16, 30913102. Bernstein, B.E., Kamal, M., Lindblad- Toh, K., Bekiranov, S., Bailey, D.K., Huebert, D.J., McMahon, S., Karlsson, E.K., Kulbokas III, E.J., Gingeras, T.R., Schreiber, S.L. & Lander, E.S. (2005). Genomic Maps and Comparative Analysis of Histone Modifications in Human and Mouse. Cell. 120, 169-181. Black, D.L. (2003). Mechanisms of alternative pre-messenger RNA splicing. Ann. Rev. Biochem. 72, 291-336. Buratowski, S. (2003). The CTD code. Nat. Struct. Biol. 10, 679-680. Carstens, R.P., Wagner, E.J. & Garcia-Blanco, M.A. (2000). An intronic splicing silencer causes skipping of the IIIb exon of fibroblast growth factor receptor 2 through involvement of polypyrimidine tract bindning protein. Mol. Cell. Biol. 20, 7388-7400. Chen, M. & Manley, J.L. (2009). Mechanisms of alternative splicing regulation: insights from molecular and genomic approaches. Nature. 10, 741-754. Cheng, D., Côté, J., Shaaban, S. & Bedford, M.T. (2007). The Arginine Methyltransferase CARM1 Regulates the Coupling of Transcription and mRNA Processing. Mol. Cell. 25, 71-83. Chodavarapu, R.K., Feng, S., Bernatavichute, Y.V., Chen, P-Y., Stroud, H., Yu, Y., Hetzel, J.A., Kuo, F., Kim, J., Cokus, S.J., Casero, D., Bernal, M., Huijser, P., Clark, A.T., Krämer, U., Merchant, S.S., Zhang, X., Jacobsen, S.E. & Pellegrini, M. (2010). Relationship between nucleosome positioning and DNA methylation. Nature. 466, 388-392. Cooper, T.A., Wan, L. & Dreyfuss, G. (2009). RNA and Disease. Cell. 136, 777-793. Das, R., Yu, J., Zhang, Z., Gygi, M.P., Krainer, A.R., Gygi, S.P. & Reed, R. (2007). SR Proteins Function in Coupling RNAP II Transcription to Pre-mRNA Splicing. Mol. Cell. 26, 867-881. Dhami, P., Saffrey, P., Bruce, A.W., Dillon, S.C., Chiang, K., Bonhoure, N., Koch, C.M., Bye, J., James, K., Foad, N.S., Ellis, P., Watkins, N.A., Ouwehand, W.H., Langford, C., Andrews, R.M., Dunham, I. & Vetrie, D. (2010). Complex Exon-Intron Marking by Histone Modifications Is Not Determined Solely by Nucleosome Distribution. PLoS ONE 5, e12339. de la Mata, M., Alonso, C.R., Kadener, S., Fededa, J.P., Blaustein, M., Pelisch, F., Cramer, P. & Kornblihtt, A.R. (2003). A slow RNA polymerase II affects alternative slicing in vivo. Mol. Cell. 12, 525-532. de la Mata, M., Lafaille, C. & Kornblihtt, A.R. (2010). First come, first served revisited : factors affecting the same alternative splicing event have different effects on the relative rates 18 of intron removal. RNA. 16, 904-912. Eperon, L.P., Graham, I.R., Griffiths, A.D. & Eperon, I.C. (1988). Effects of RNA secondary structure on alternative splicing of pre-mRNA: is folding limited to a region behind the transcribing RNA polymerase?. Cell. 54, 393-401. Fu, X-D. & Maniatis, T. (1992). Isolation of a complementary DNA that encodes the mammalian splicing factorSC35. Science. 256, 535-538. Ge, H. & Manley, J.L. (1990). A protein factor, ASF, controls cell-specific alternative splicing of SV40 early pre-mRNA in vitro. Cell. 62, 25-34. Graveley, B.R. (2000). Sorting out the complexity of SR-protein functions. RNA. 6, 11971211. Guccione, E., Martinato, F., Finocchiaro, G., Luzi, L., Tizzoni, L., Dall′ Olio, V., Zardo, G., Nervi, C., Bernard, L. & Amati, B. (2006). Myc-binding-site recognition in the human genome is determined by chromatin context. Nat. Cell. Biol. 8, 764-770. Gunderson, F.Q. & Johnson, T.L. (2009). Acetylation by the transcriptional coactivator Gcn5 plays a novel role in co-transcriptional spliceosomen assembly. PLoS Genet. 5, e1000682. Hanamura, A., Cáceres, J.F., Mayeda, A., Franza JR, B.R. & Krainer, A.R. (1998). Regulated tissue-specific expression of antagonistic pre-mRNA splicing factors. RNA. 4, 430-444. Hnilicová, J., Hozeifi, S., Dušková, E., Icha, J., Tománková, T. & Staněk, D. (2011). Histone Deacetylase Activity Modulates Alternative Splicing. PLoS One. 6(2), e16727. Hon, G., Wang, W. & Ren, B. (2009). Discovery and annotation of functional chromatin signatures in the human genome. PloS. Comp. Biol. 5(11), e1000566. Klaassen, C.D. & Hong Lu, J.U.C. (2011). Epigenetic regulation of drug processing genes. Toxi. Mech. Meth. 21, 312-324. Krebs, J.E., Goldstein, E.S. & Kilpatrick, S.T. (2010). Lewin’s Genes X. Jones and Bartlett Publisher, LLC. Kolasinska-Zwierz, P., Down, T., Latorre, I., Liu, T., Liu, S. & Ahringer, J. (2009). Differential chromatin marking of introns and expressed exons by H3K36me3. Nat. Gen. 41, 376-381. Kouzarides, T. (2007). Chromatin Modifications and Their Function. Cell. 128, 693-705. Kutney, S.N., Hong, R., Macfarlan, T. & Chakravarti, D. (2004). A Signaling Role of Histone-binding Proteins and INHAT Subunits pp32 and Set/TAF-Iβ in Integrating Chromatin Hypoacetylation and Transcriptional Repression. J. Biol. Chem. 279, 30850-30855. Li, B., Carey, M. & Workman, J.L. (2007). The Role of Chromatin during Transcription. Cell. 128, 707-719. 19 Loomis, R.J., Naoe, Y., Parker, B.J., Savic, V., Bozovsky, M.R., Macfarlan, T., Manley, J.L. & Chakravarti, D. (2009). Chromatin Binding of SRp20 and ASF/SF2 and Dissociation from Mitotic Chromosomes Is Modulated by Histone H3 Serine 10 Phosphorylation. Mol. Cell. 33, 450-461. Luco, R.F., Allo, M., Schor, I.E., Kornblihtt A.R., & Misteli, T. (2011). Epigenetics in Alternative Pre-mRNA Splicing. Cell 144, 16-21. Luco, R.F., Pan, Q., Tominaga, K., Blencowe, B.J., Pereira-Smith, O. & Misteli, T. (2010). Regulation of Alternative Spilcing by Histone Modifications. Science. 327, 996-1000. Martens, J.A. & Winston, F. (2003). Recent advances in understanding chromatin remodeling by Swi/Snf complexes. Curr. Op. Gen. Dev. 13, 136-142. Martinez, E., Palhan, V.B., Tjernberg, A., Lymar, E.S., Gamper, A.M., Kundu, T.K., Chait, B.T. & Roeder, R.G. (2001). Human STAGA complex is a chromatin-acetylating transcription coactivator that interacts with pre-mRNA splicing and DNA damage-binding factors in vivo. Mol. Cell. Biol. 21, 6782-6795. Mauger, D.M., Lin, K. & Garcia-Blanco, M.A. (2008). hnRNP H and hnRNP F Complex with Fox2 To Silence Fibroblast Growth Factor Receptor 2 Exon IIIc. Mol. Cell. Biol. Vo28 No.17, 5403-5419. Meissner, A., Mikkelsen, T.S., Hongcang, G., Wernig, M., Hanna, J., Sivachenko, A., Xiaolan, Z., Bernstein, B.E., Nusbaum, C., Jaffe, D.B., Gnirke, A., Jaenisch, R. & Lander, E.S. (2008). Genome scale DNA methylation maps of pluripotent and differentiated cells. Nature. 454, 766-770. Misteli, T. & Spector, D.L. (1999). RNA polymerase II targets pre-mRNA splicing factors to transcriptions sites in vivo. Mol. Cell. 3, 697-705. Muñoz, J.M., de la Mata, M. & Kornblihtt. (2010). The carboxy terminal domain of RNA polymerase II and alternative splicing. Tr. Biochem. Sci. 35, 497-504. Nilsen, T.W. & Graveley, B.R. (2010). Expansion of the eukaryotic proteome by alternative splicing. Nature. 463, 457-463. Pan, Q., Shai, O., Lee, L.J., Frey, B.J., & Blencowe, B.J. (2008). Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing. Nat. Genet. 40, 1413-1415. Pandya-Jones, A., & Black, D.L. (2009). Co-transcriptional splicing of constitutive and alternative exons. RNA 15, 1896-1908. Pray-Grant, M.G., Daniel, J.A., Schieltz, D., Yates III, J.R. & Grant, P.A. (2005). Chd1 chromodomain links histone H3 methylation with SAGA- and SLIK- dependent acetylation. Nature. 433, 434-438. Saint-André, V., Batsché, E., Rachez, C. & Muchardt, C. (2011). Histone H3 lysine 9 trimethylation and HP1γ favor inclusion of alternative exons. Nat. Struct. Mol. Biol. 18, 337- 20 344. Schreiber, S.L. & Bernstein, B.E. (2002). Signaling Network Model of Chromatin. Cell. 111, 771-778. Schwartz, S., Meshorer, E., & Ast, G. (2009). Chromatin organization marks exon-intron structure. Nat. Struct. Mol. Biol. 16, 990-995. Shen, H., Kan, J.L.C. & Green, M.R. (2004). Arginine-Serine-Rich Domains Bound at Splicing Enhancers Contact the Branchpoint to Promote Prespliceosome Assembly. Mol. Cell. 13, 367-376. Sims III, R.J., Millhouse, S., Chen, C-F., Lewis, B.A., Erdjument- Bromage, H., Tempst, P., Manley, J.L. & Reinberg, D. (2007). Recognition of Trimethylated Histone H3 Lysine 4 Facilitates the Recruitment of Transcription Postinitiation Factors and Pre-mRNA Splicing. Mol. Cell. 28, 665-676. Spies, N., Nielsen, C.B., Padgett, R.A. & Burge, C.B. (2009). Biased chromatin signatures around polyadenylation sites and exons. Mol. Cell. 36, 245-254. Tang, Z.Z., Sharma, S., Zheng, S., Chawla, G., Nikolic, J. & Black, D.L. (2011). Regulation of the Mutually Exclusive Exons 8a and 8b in the CaV1.2 Calcium Channel Transcript by Polypyrimidine Tract-binding Protein. J. Biol. Chem. 286, 10007-10016. Tamarin, R.H. (2004). Principles of genetics. The McGraw-Hill Companies. Tyagi, A., Ryme, J., Brodin, D., Östlund-Farrants, A.K. & Visa, N. (2009). SWI/SNF Associates with Nascent Pre-mRNPs and Regulates Alternative Pre-mRNA Processing. PLoS. Genet. 5, e1000470. Ule, J., Stefani, G., Mele, A., Ruggio, M., Wang, X., Taneri, B., Gaasterland, T., Blencowes, B.J. & Darnell, R.B. (2006). An RNA map predicting NOVA- dependent splicing regulation. Nature. 444, 580-586. Wagner, E.J. & Garcia-Blanco, M.A. (2002). RNAi-Mediated PTB Depletion Leads to Enhanced Exon Definition. Mol. Cell. 10, 943-949. Wahl, M.C., Will, C.L. & Luhrmann, R. (2009). The Spliceosome: Design Principles of a Dynamic RNP Machine. Cell. 136, 701-718. Wang, Z. & Burge, C.B. (2008). Splicing regulation: From a parts list of regulatory elements to an integrated splicing code. RNA. 14, 802-813. Wang, E.T., Sandberg, R., Luo, S., Khrebtukova, I., Zhang, L., Mayrs, C., Kingsmore, S.F., Schroth, G.P. & Burge, C.B. (2008). Alternative isoform regulation in human tissue transcriptome. Nature. 456, 470-476. Yeo, G.W., Coufal, N.G., Liang, T.Y., Peng, G.E., Fu, X-D. & Gage, F.H. (2009). An RNA code for the FOX2 splicing regulator revealed by mapping RNA-protein interactions in stem cells. Nat. Struct. Mol. Biol. 16, 130-137. 21 Yuryev, A., Patrurajan, M., Litingtung, Y., Joshi, R.V., Gentile, C., Bebara, M. & Cordens, J.L. (1996). The C-terminal domain of the largest subunitof RNA polymerase II interacts with a novel set of serine/arginine-rich proteins. Proc. Natl. Acad. Sci. 93, 6975-6980. Zhou, Z., Licklider, L.J., Gygi, S.P. & Reed, R. (2002). Comprehensive proteomic analysis of the human spliceosomen. Nature. 419, 182-185. Figurer: Figur 1: Intronens uppbyggnad; Freeman, W.H. & Pierce, B. (2005). Genetics: A Conceptual Approach. W.H. Freeman and Company. 2nd ed. 381 Figur 2: Fyra typer av alternative splicing; Nilsen, T.W. & Graveley, B.R. (2010). Expansion of the eukaryotic proteome by alternative splicing. Nature. 463, 457-463. Figur 3: Uppbyggnaden av spliceosomen; Max Planck Institute for Biophysical Chemistry. (2008). [http://www.mpibpc.mpg.de/groups/pr/PR/2008/08_10_eng_RN/]. 2011-04-11 Figur 4: De fyra olika cis- regulatoriska elementen; Wang, Z. & Burge, C.B. (2008). Splicing regulation: From a parts list of regulatory elements to an integrated splicing code. RNA. 14, 802-813. Figur 5: RNA polymeras II med CTD domänen; Luco, R.F., Allo, M., Schor, I.E., Kornblihtt A.R., & Misteli, T. (2011). Epigenetics in Alternative Pre-mRNA Splicing. Cell 144, 16-21. Figur 6: Histonmodifieringsmönster på genen: Zhou, V.W., Goren, A. & Bernstein, B.E. (2011). Charting histone modifications and the functional organization of mammalian genomes. Nat. Rev. Gen. 12, 7-18. Figur 7: Kromatin- splicing adaptor systemet; Luco, R.F., Pan, Q., Tominaga, K., Blencowe, B.J., Pereira-Smith, O. & Misteli, T. (2010). Regulation of Alternative Spilcing by Histone Modifications. Science. 327, 996-1000. Figur 8: Integrerad modell av den alternativa splicingens reglering; Luco, R.F., Allo, M., Schor, I.E., Kornblihtt A.R., & Misteli, T. (2011). Epigenetics in Alternative Pre-mRNA Splicing. Cell. 144, 16-21. 22