Hur regleras tillverkningen av livets byggstenar?
advertisement

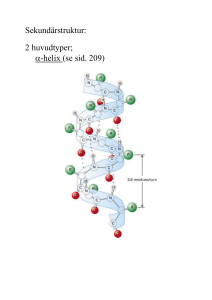
Alexander Balhuizen Hur regleras tillverkningen av livets byggstenar? DNA och dess byggstenar, deoxinukleosidtrifosfat, dNTP, bygger upp arvsmassans skelett och förmedlar den genetiska informationen för det biologiska livet. För att det skall finnas tillräckligt med byggstenar att bygga upp och reparera DNA med krävs det att det produceras nya byggstenar i en reglerad takt. Om denna reglering inte sker kan det bildas fel i informationen vilket kan ge upphov till mutationer. Byggstenarna, dNTP kan inte bildas direkt utan endast föregångaren nukleosidtrifosfat, NTP kan bildas utav de befintliga kemiska beståndsdelarna. Därför krävs det att det finns ett enzym som omvandlar NTP till dNTP. Ett sådant enzym är ribonukleotidreduktas, RNR som finns i tre olika klasser I, II och III. Klass I finns i djur och bakterier, klass II finns i syretåliga bakterier medan klass III finns i bakterier och virus som inte tål syre. Omvandlingen från NTP till dNTP sker med hjälp av en radikal, atom med en extra fri elektron, för att klippa av en syreatom från NTP. Regleringen av byggstenarna är styrd av flertalet mekanismer. Ett exempel är att det kan ske en allosterisk reglering – en reglering som sker i en annan del av enzymet när detta binder till NTP/dNTP. Beroende på vilken sort som binder in förmedlas informationen genom proteinstrukturen, och i proteinets tillverkningsdel produceras en av de fyra byggstenarna. För att kunna studera denna reglering är det viktigt att studera proteinmolekylens verkliga, tredimensionella utseende som fastställs med kristallisering och röntgenkristallografi. Syftet med mitt projekt var att studera den allosteriska regleringen dels på enzymet i sin naturliga form, dels studera, med muterat enzym, den troliga påverkan som två speciella aminosyror har på produktionsmekanismen i RNR klass III från ett virus, fag T4. Samt att kristallisera och lösa strukturen för en variant av samma enzym från bakterien Thermotoga maritima. Proteinstrukturen behövs för att man skall kunna studera regleringen och produktionsmekaniken av dNTP:s. Projektet startades med en produktion av enzymer från fag T4 med bakterien Escherichia coli. Enzymerna från fag T4 renades därefter fram och sattes upp för kristallisering med kända betingelser. Det genomfördes även efter försök att hitta nya kristallisationsbetingelser med två olika maskinella tester som innehåller de vanligaste kemikalierna för att bilda kristaller t.ex. salter, alkoholer med olika pH. Thermotoga maritima RNR klass III kördes i samma tester och lämpliga kristaller hittades. Dessa förfinades för hand och gav slutligen fina kristaller. Ju finare, större, kristaller desto bättre upplösning. Dessa kristaller hade en upplösning på 2,8 Ångström vid bestrålning med röntgenstrålar. Ju lägre Ångström desto fler detaljer kan ses. Vid 2,8 Å kan aminosyror och deras atomer skönjas. I framtiden skulle dessa kristaller kunna leda till att den tredimensionella strukturen för RNR klass III Thermotoga maritima kan lösas och att man evolutionärt kan dra viktiga slutsatser om hur övergången från RNA- till DNA-värld gick till. Handledare: Derek Logan Examensarbete 20 p i Molekylärbiologi. Vt. 2007 Institutionen för kemi, Avdelningen för molekylär biofysik, Lunds universitet Alexander Balhuizen Production, purification and crystallization of the class III ribonucleotide reductase from Thermotoga maritima and phage T4 Every biological life form is dependent upon DNA and its building blocks, the deoxyribonucleoside triposphates (dNTPs). Ribonucleotide reductase (RNR) is an essential enzyme that synthesises new building dNTP building blocks.This enzyme exists in three different classes, I, II and III. The class III RNRs, which have been studied in this project, exist in anaerobic bacteria and archaea. RNR synthesizes dNTPs from NTPs using radical chemistry. The rate and specificity of the synthesis is controlled by the cellular dNTP concentration through feedback allosteric regulatory mechanisms, which involve structural movements of the protein. There are only a few structures solved from RNR class III, all of which are of variants of the enzyme from phage T4, and the crystallization of RNR class III from Thermotoga maritima will contribute necessary information on how this enzyme has evolved. In addition it will clarify which of the functional conclusions made on the basis of the phage T4 structure are general and which are specific for T4. This project started out with the production of three variants of the protein from phage T4, using heterologus expression in E. coli. The proteins were extracted, purified with chromatography and setup to crystallize. Meanwhile the conditions for crystallization of the homologous enzyme from T. maritima were determined using large screens with robotics at MAX-lab and later optimized by hand to get good crystals for diffraction. The crystals from T. maritima were examined with X-rays at MAX-lab and the diffraction data was processed to a best resolution of 2.8 Å. In the future this will lead to the structure being solved. Advisor: Derek Logan Degree project 20 credits in Molecular biology. Spring 2007 Department of Chemistry, Division of Molecular Biophysics, Lund University