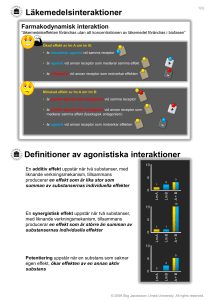
Forskning på IDA Exempel: Datasäkerhet
advertisement

Hur lagras informationen?
(hög nivå)
Hur accessar man informationen?
(användarnivå)
1
Biologiska databanker
• biologisk data i elektronisk format
• exempel: SWISS-PROT, EMBL, DDBJ,
PDB, GENBANK, KEGG, ACEDB
• används dagligen i forskningen
2
Biologiska databanker
Forskningsresultat
Modell
Databank
Databankhanteringssystem
Frågor
Svar
behandling av
frågor/uppdateringar
Access till lagrad data
Fysiska
databanken
3
1 tgctacccgc gcccgggctt ctggggtgtt ccccaaccac ggcccagccc tgccacaccc
61 cccgcccccg gcctccgcag ctcggcatgg gcgcgggggt gctcgtcctg ggcgcctccg
121 agcccggtaa cctgtcgtcg gccgcaccgc tccccgacgg cgcggccacc gcggcgcggc
181 tgctggtgcc cgcgtcgccg cccgcctcgt tgctgcctcc cgccagcgaa agccccgagc
241 cgctgtctca gcagtggaca gcgggcatgg gtctgctgat ggcgctcatc gtgctgctca
301 tcgtggcggg caatgtgctg gtgatcgtgg ccatcgccaa gacgccgcgg ctgcagacgc
361 tcaccaacct cttcatcatg tccctggcca gcgccgacct ggtcatgggg ctgctggtgg
421 tgccgttcgg ggccaccatc gtggtgtggg gccgctggga gtacggctcc ttcttctgcg
481 agctgtggac ctcagtggac gtgctgtgcg tgacggccag catcgagacc ctgtgtgtca
541 ttgccctgga ccgctacctc gccatcacct cgcccttccg ctaccagagc ctgctgacgc
601 gcgcgcgggc gcggggcctc gtgtgcaccg tgtgggccat ctcggccctg gtgtccttcc
661 tgcccatcct catgcactgg tggcgggcgg agagcgacga ggcgcgccgc tgctacaacg
721 accccaagtg ctgcgacttc gtcaccaacc gggcctacgc catcgcctcg tccgtagtct
781 ccttctacgt gcccctgtgc atcatggcct tcgtgtacct gcgggtgttc cgcgaggccc
841 agaagcaggt gaagaagatc gacagctgcg agcgccgttt cctcggcggc ccagcgcggc
901 cgccctcgcc ctcgccctcg cccgtccccg cgcccgcgcc gccgcccgga cccccgcgcc
961 ccgccgccgc cgccgccacc gccccgctgg ccaacgggcg tgcgggtaag cggcggccct
1021 cgcgcctcgt ggccctacgc gagcagaagg cgctcaagac gctgggcatc atcatgggcg
1081 tcttcacgct ctgctggctg cccttcttcc tggccaacgt ggtgaaggcc ttccaccgcg
1141 agctggtgcc cgaccgcctc ttcgtcttct tcaactggct gggctacgcc aactcggcct
1201 tcaaccccat catctactgc cgcagccccg acttccgcaa ggccttccag ggactgctct
1261 gctgcgcgcg cagggctgcc cgccggcgcc acgcgaccca cggagaccgg ccgcgcgcct
1321 cgggctgtct ggcccggccc ggacccccgc catcgcccgg ggccgcctcg gacgacgacg
1381 acgacgatgt cgtcggggcc acgccgcccg cgcgcctgct ggagccctgg gccggctgca
1441 acggcggggc ggcggcggac agcgactcga gcctggacga gccgtgccgc cccggcttcg
1501 cctcggaatc caaggtgtag ggcccggcgc ggggcgcgga ctccgggcac ggcttcccag
1561 gggaacgagg agatctgtgt ttacttaaga ccgatagcag gtgaactcga agcccacaat
1621 cctcgtctga atcatccgag gcaaagagaa aagccacgga ccgttgcaca aaaaggaaag
1681 tttgggaagg gatgggagag tggcttgctg atgttccttg ttg
4
DEFINITION
ACCESSION
SOURCE ORGANISM
REFERENCE
AUTHORS
TITLE
REFERENCE
AUTHORS
TITLE
Homo sapiens adrenergic, beta-1-, receptor
NM_000684
human
1
Frielle, Collins, Daniel, Caron, Lefkowitz,
Kobilka
Cloning of the cDNA for the human
beta 1-adrenergic receptor
2
Frielle, Kobilka, Lefkowitz, Caron
Human beta 1- and beta 2-adrenergic
receptors: structurally and functionally
related receptors derived from distinct
genes
5
Entity-relationship
protein-id
source
PROTEIN
accession
m
definition
Reference
n
title
article-id
ARTICLE
author
6
Hur lagras informationen?
(hög nivå)
Hur accessar man informationen?
(användarnivå)
•
•
•
•
Text (IR)
Semistrukturerad data
Datamodeller (DB)
Regler + Fakta (KB)
struktur
precision
7
Innehåll
• Text: filmodell, formell modell,
konceptuella modeller (boolesk, vektor)
• Databaser: Relationsdatabaser,
Objektorienterade databaser
• Semistrukturerad data
• Kunskapsbaser
8
Text - Information Retrieval (IR)
• sökning baseras på ord
• konceptuella modeller:
boolesk, vektor, probabilistisk, …
• filmodell:
flat fil, inverterad fil, ...
9
IR - Filmodell: inverterad fil
inverterad fil
anslagningsfil
WORD
HITS
LINK
…
…
…
adrenergic
32
…
…
cloning
…
receptor
22
…
…
…
DOCUMENTS
…
Doc1
…
Doc2
1
…
53
…
DOC# LINK
dokumentfil
…
…
5
…
1
2
…
5
…
…
10
IR - Filmodell: inverterad fil
• kontollerad vokabulär
(eng. controlled vocabulary)
• stoplista
• stamning (eng. stemming)
11
IR - formell beskrivning
IR modell: (D,Q,F,R)
• D är en mängd av dokumentrepresentationer
• Q är en mängd av frågor
• F är ett ramverk för
dokumentrepresentationer, frågor och deras
samband
• R tilldelar ett dokument-fråga-par ett reellt
tal (rankning)
12
IR - konceptuella modeller
Klassikt IR
• Boolesk
• Vektormodellen
• (Probabilistisk)
13
Booleska modellen
dokumentrepresentation
Doc1
adrenergic
yes
cloning
yes
Doc2
no
yes
receptor
no
--> (1 1 0)
no
--> (0 1 0)
14
Booleska modellen
frågor : boolesk (and, or, not)
Q1: cloning and (adrenergic or receptor)
frågor översätts till disjunktiv normalform (DNF)
DNF: disjunktion av konjunktioner av ord med eller utan ‘not’
Regler: not not A --> A
not(A and B) --> not A or not B
not(A or B) --> not A and not B
(A or B) and C --> (A and C) or (B and C)
A and (B or C) --> (A and B) or (A and C)
(A and B) or C --> (A or C) and (B or C)
A or (B and C) --> (A or B) and (A or C)
15
Booleska modellen
Q1: cloning and (adrenergic or receptor)
--> (cloning and adrenergic) or (cloning and receptor)
DNF kompletteras
+ översätts till samma representation som dokumenten
(cloning and adrenergic) or (cloning and receptor)
--> (cloning and adrenergic and receptor)
or (cloning and adrenergic and not receptor)
or (cloning and receptor and adrenergic)
or (cloning and receptor and not adrenergic)
--> (1 1 1) or (1 1 0) or (1 1 1) or (0 1 1)
--> (1 1 1) or (1 1 0) or (0 1 1)
16
Booleska modellen
Doc1
adrenergic
yes
cloning
yes
Doc2
no
yes
Q1: cloning and (adrenergic or receptor)
--> (1 1 1) or (1 1 0) or (0 1 1)
receptor
no
--> (1 1 0)
no
--> (0 1 0)
Resultat: Doc1
Q2: cloning and not adrenergic
--> (0 1 0) or (0 1 1)
Resultat: Doc2
17
Booleska modellen
Fördelar
• baserad på en intuitiv och enkel formell
modell (mängdteori och boolesk algebra)
Nackdelar
• binära beslut
- ett ord är relevant eller inte
- ett dokument är relevant eller inte,
ingen partiell matchning
18
Booleska modellen
Doc1
adrenergic
yes
cloning
yes
Doc2
no
yes
Q3: adrenergic and receptor
--> (1 0 1) or (1 1 1)
receptor
no
--> (1 1 0)
no
--> (0 1 0)
Resultat: tom
19
Vektormodellen (förenklad)
Doc1 (1,1,0)
Doc2 (0,1,0)
cloning
Q (1,1,1)
adrenergic
sim(d,q) = d . q
|d| x |q|
receptor
20
Vektormodellen
• Introducerar vikter i dokumentvektorerna
(t.ex. Doc3 (0, 0.5, 0))
• Vikterna representerar hur viktigt en term är
för beskrivningen av ett dokuments innehåll
• Vikterna är positiva reella tal
• Om en term inte förekommer i dokumentet,
då är vikten = 0
21
Vektormodellen
Doc1 (1,1,0)
Doc3 (0,0.5,0)
cloning
Q4 (0.5,0.5,0.5)
adrenergic
sim(d,q) = d . q
|d| x |q|
receptor
22
Vektormodellen
• Hur definierar man vikterna? tf-idf
dj (w1,j, …, wt,j)
wi,j = vikten för term ki i dokument dj
= fi,j x idfi
23
Vektormodellen
• Hur definierar man vikterna? tf-idf
term frequency freqi,j: hur ofta förekommer
term ki i dokument dj?
normaliserad term frequency:
fi,j = freqi,j / maxl freql,j
24
Vektormodellen
• Hur definierar man vikterna? tf-idf
document frequency : i hur många dokument
förekommer term ki?
N = totala antalet dokument
ni = antal dokument i vilka ki förekommer
inverse document frequency idfi: log (N / ni)
25
Vektormodellen
• Hur definierar man vikterna för en fråga?
rekommendation:
q= (w1,q, …, wt,j)
wi,q = vikt för term ki i fråga q
= (0.5 + 0.5 fi,q) x idfi
26
Vektormodellen
•
•
-
Fördelar
termviktning förbättrar sökprestanda
partiell matchning
rankning enligt likhet
Nackdel?
antagande att termerna förekommer
oberoende av varandra
27
IR - effektivitetsmått
Precision =
antal hittade dokument som är relevanta
antal hittade dokument
Recall =
antal hittade dokument som är relevanta
antal relevanta dokument
28
IR - mått
Precision =
|RA| / |A|
Relevanta dokument
i svarsmängden
|RA|
Recall =
|RA| / |R|
Relevanta dokument
|R|
Svarsmängden
|A|
29
Databaser
• Relationsdatabaser:
- modell: tabeller + relationsalgebran
- frågespråk (SQL)
• Objektorienterade databaser:
- modell: fortlevande object,
meddelande, inkapsling, ärvning
- frågespråk (t.ex. OQL)
• System: GDB (R), ACEDB (OO)
30
Relationsdatabaser
PROTEIN
REFERENCE
PROTEIN-ID
1
ACCESSION
DEFINITION
SOURCE
PROTEIN-ID
ARTICLE-ID
NM_000684
Homo sapiens
adrenergic,
beta-1-, receptor
human
1
1
1
2
ARTICLE-AUTHOR
ARTICLE-ID
1
1
1
1
1
1
2
2
2
2
ARTICLE-TITLE
AUTHOR
Frielle
Collins
Daniel
Caron
Lefkowitz
Kobilka
Frielle
Kobilka
Lefkowitz
Caron
ARTICLE-ID
TITLE
1
Cloning of the cDNA for the human
beta 1-adrenergic receptor
2
Human beta 1- and beta 2adrenergic receptors: structurally
and functionally related
receptors derived from distinct
genes
31
SQL
select source
from protein
where accession = NM_000684;
PROTEIN
PROTEIN-ID
1
ACCESSION
DEFINITION
SOURCE
NM_000684
Homo sapiens
adrenergic,
beta-1-, receptor
human
32
SQL
select title
from protein, article-title, reference
where protein.accession = NM_000684
and protein.protein-id
= reference.protein-id
and reference.article-id
= article-title.article-id;
PROTEIN
PROTEIN-ID
1
REFERENCE
PROTEIN-ID
ARTICLE-ID
1
1
1
2
ARTICLE-TITLE
ACCESSION
DEFINITION
SOURCE
NM_000684
Homo sapiens
adrenergic,
beta-1-, receptor
human
ARTICLE-ID
TITLE
1
Cloning of the …
2
Human beta 1- …
33
Objekt-Orienterade Databaser
(OODB)
• Världen modelleras m.h.a. objekt.
• Ett objekt har ett tillstånd (värde) och ett
beteende (operationer).
• fortlevande (eng. persistent) objekt permanent lagring
(ibland tillåts även kortvariga (eng.
transient) objekt)
34
Objekt
• Ett objekt har en objektidentifierare (OID)
som inte är synlig för användaren.
• OIDn kan inte ändras
• objekt versus värde
(ett värde har ingen OID)
• objektstrukturen kan vara godtyckligt
komplex (atom, tuple, set, list, bag, array)
35
Exempel - objekttillstånd
• o1(id1, tuple,
<accession: NM_000684,
source : human,
definition: ’Homo sapiens adrenergic …’,
reference: o2>)
• o2(id2, set, {o3,o4})
OBS: Dessa exempel använder inte någon standardsyntax36
Exempel - objekttillstånd
• o3(id3, tuple,
<title: `Cloning of …’, author: o5 >)
• o4(id4, tuple,
<title: `Human beta-1 …’, author: o6 >)
• o5(id5, list, [Frielle, Collins, Daniel, Caron,
Lefkowitz, Kobilka])
• o6(id6, list, [Frielle, Kobilka , Lefkowitz,
Caron])
37
human
NM_000684
”Homo sapiens adrenergic,
beta-1-, receptor”
SOURCE
ACCESSION
DEFINITION
REFERENCE
AUTHOR
set
AUTHOR
TITLE
list
”Cloning of …”
Frielle
TITLE
list
Collins
Frielle
Daniel
Kobilka
Caron
Lefkowitz
Lefkowitz
Kobilka
”Human beta-1 …”
Caron
38
Klasser
define class protein
type tuple (
accession: string;
source : string;
definition: string;
reference: set(article); );
operations
create-protein(string,string,string,set(article)): protein;
get-accession: string;
get-source: string;
get-definition: string;
get-references: set(article);
add-reference(article): void;
end protein;
39
Klasser
define class article
type tuple (
title: string;
author: list(string); );
operations
create-article(string, list(string)): article;
get-title string;
get-authors: list(string);
print-article-info string;
end article;
40
Exempel program
program
variables: article1, article2, protein1;
begin
article1 := create-article(’Cloning….’, list(Frielle, Collins,
Daniel, Caron, Lefkowitz, Kobilka));
protein1 := create-protein(NM_000684, human,’Homo
sapiens adrenergic …’, set(article1));
article2 := create-article(’ Human beta-1….’, list(Frielle,
Kobilka , Lefkowitz, Caron]);
protein1.add-reference(article2);
end;
41
Operationer
• inkapsling: operation = gränssnitt + kropp
- gränssnitt: hur anropar man operationen?
vad ger operationen som resultat?
> synlig för användaren, används i program
- kropp: hur implementeras operationen?
> osynlig för användaren
• program baseras på skickandet av
meddelande (eng. message passing)
42
Ärvning
• journal-article subtype-of article:
journal-name journal-volume page-numbers
journal-article ärver alla attribut och operationer från article
och har även journal-name, journal-volume och pagenumbers som attribut
• human-protein subtype-of protein (source = ’human’)
43
Operator overloading
• Man kan använda samma operatornamn för
olika implementationer
exempel:
print-article-info för article skriver ut information om titel
och författare.
print-article-info för journal-article skriver ut information
om titel och författare men även om tidskriftens namn,
volym och sidonummer.
44
Frågespråk OQL
• select … from … where
select distinct … from … where
• iteratorvariabler
• väguttryck (eng. path expressions)
• struct
45
Frågor
select o.source
from o in protein
where o.accession = NM_000684;
NM_000684
ACCESSION
human
SOURCE
46
Frågor
select struct (accession: o.accession, source: o.source)
from o in protein
where Frielle in
(select a.author
from a in o.reference);
NM_000684
human
ACCESSION
SOURCE
REFERENCE
set
AUTHOR
Frielle
AUTHOR
Frielle
47
Frågespråk OQL
OQL tillåter även:
• vyar
• aggregering
• speciella operationer för list och array
(first, last, nth)
• order-by
• group-by
48
Semistrukturerad data
• data som är ej enbart text, men som inte är
så välstrukturerad som data i databaser
• förekommer ofta i webbdatabanker och när
man vill integrera databanker
• system: Genbank, OMIM
49
Semistrukturerad data egenskaper
•
•
•
•
•
oregelbunden struktur
strukturen är implicit
strukturen är partiell
’data guide’ och schema
stora data guides
50
Semistrukturerad data egenskaper
• man ska kunna ignorera data guide vid
frågor
• data guide utvecklas snabbt
• objekt kan ändra tillhörighet av typ/klass
• skillnaden mellan data och data guide
försvinner
51
Semistrukturerad data - modell
• nätverk av noder
• objektmodell (oid)
• fråga: vägsökning i nätverket (’path search’)
52
”Homo sapiens adrenergic,
beta-1-, receptor”
human
NM_000684
SOURCE
ACCESSION
DEFINITION
REFERENCE
REFERENCE
AUTHOR
TITLE
AUTHOR
AUTHOR
”Cloning of …”
Frielle
Collins
TITLE
AUTHOR
AUTHOR
”Human beta-1 …”
AUTHOR
AUTHOR
Daniel
AUTHOR
Caron
AUTHOR
AUTHOR
Lefkowitz
Kobilka
53
Frågor
NM_000684
select source
where accession = NM_000684;
human
SOURCE
ACCESSION
My-protein
NM_000684
select reference.title
where accession = NM_000684;
ACCESSION My-protein
REFERENCE
TITLE
”Cloning of …”
REFERENCE
TITLE
”Human
beta-1 …”
54
Frågor
NM_000684
select source
where accession = NM_000684;
human
SOURCE
ACCESSION
My-protein
NM_000684
select #p.title
where accession = NM_000684;
ACCESSION My-protein
REFERENCE
TITLE
”Cloning of …”
REFERENCE
TITLE
”Human
beta-1 …”
55
Kunskapsbaser
• ofta baserad på en logik
• svarsmekanism baserad på en
inferensmekanism
• kunskapsbaser kan ofta lagras i
primärminne
• användbar vid ontologier
56
Kunskapsbaser
(F) source(NM_000684, Human)
(R) source(P?,Human) => source(P?,Mammal)
(R) source(P?,Mammal) => source(P?,Vertebrate)
Q: ?- source(NM_000684, Vertebrate)
A: yes
Q: ?- source(x?, Mammal)
A: x? = NM_000684
57
Litteratur
• Information Retrieval
* Baeza-Yates, R., Ribeiro-Neto, B., Modern Information Retrieval, AddisonWesley, 1999.
• Databaser
* Elmasri, R., Navathe, S., Fundamentals of database systems, 4th edition,
Addison Wesley, 2004.
• Semistrukturerad data
* Abiteboul, S., ‘Querying semi-structured data’, Proceedings of the Sixth
International Conference on Database Theory, LNCS 1186, pp 1-18, 1997.
(* = ingår helt eller delvis i kurslitteraturen, se kurshemsidan)
58