Populärvetenskaplig sammanfattning
advertisement

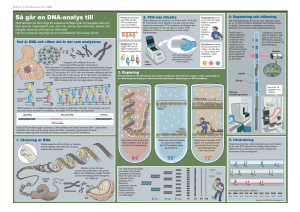
Hänglås och gener Marcus Nystrand Dideoxyribonukleotidsyra, DNA. Denna enkla, spiralformade molekyl – snillrikt uppbyggd av blott fyra byggstenar kallade nukleotider – som utgör ritningen för majoriteten av cellens komponenter. Länge var denna molekyls betydelse förbisedd, men efter det att dess struktur och funktion utretts och accepterats skred vetenskapen snabbt framåt för att kulminera i utrönandet av det mänskliga genomet för ett par år sedan. Vad finns då att finna i vårt genom? Utöver uppskattningsvis 30 000 gener består mer än 90% av vårt genom av DNA-sekvenser utan känd funktion. Med undantag för enäggstvillingar har olika individer olika DNA-sammansättning. Dessa skillnader kan vara betydelsefulla att känna till i exempelvis rättsmedicin, faderskapsmål eller diagnosticering av sjukdomar. En mycket vanlig genetiskt variation mellan individer är vad som kallas enbaspolymorfier eller SNP. En SNP är ett ställe i en DNA-molekyl där en individ har en nukleotid och en annan individ en annan nukleotid. Då de förekommer ofta och överallt i genomet har de visat sig vara värdefulla genetiska vägskyltar, och har därför blivit grundligt studerade. Naturligtvis finns det ett intresse för molekylära redskap att studera SNP med på dagens växande marknad för verktyg för genetisk analys. Ett av dessa är en svensk uppfinning, hänglåssonder. DNA har den egenskapen att det kan binda till sig själv enligt ett specifikt mönster. Hänglåssonder, som består av korta strängar DNA, kan alltså binda till genomiskt DNA. När hänglåssonden bundit kan denna sträng – med hjälp av ett enzym – fogas ihop till en cirkel. Denna DNA-cirkel kan vidare användas för att ge en signal i en genetisk analys. Men om hängslåssonden inte binder till genomprovet på ett korrekt sätt kan inga cirklar bildas och inga signaler kommer att erhållas. Genom att noggrant konstruera olika typer av hänglåssonder kan dessa utnyttjas för att särskilja mellan SNPs. För att snabba upp analyserna vill man gärna tillsätta sonder i ett stort överskott. Detta orsakar emellertid vissa problem då ett överskott av sonder leder till ökat bakgrundsbrus i analysen på grund av ickereagerade sonder. Tidigare har man använt ett enzym för att eliminera ickereagerade sonder, men då detta tar mer tid och kostar mer vill man kringgå denna metod. Mitt examensarbete berörde en ny metod för att undkomma ickereagerade sonder. Vanligtvis används två enzym vid analys med hänglåssonder, ett ligas för att foga ihop sonder till cirklar och ett polymeras för att få signal av cirklarna. Under mina laborationer undersökte jag möjligheten att låta polymeraset, det enzym som ger signal av cirklar, eliminera eventuella ickereagerade sonder. Jag använde då hänglåssonder med en” ögla” i ena änden. Har sonderna en ögla i ena änden kommer nämligen polymeraset att bearbeta även ickereagerade sonder, dock utan att ge någon signal från dessa. Istället kommer alla ickereagerade sonder förvandlas till ”döda” molekyler som inte kan ge upphov till något bakgrundsbrus. Det första jag gjorde var att undersöka hur stora öglorna skulle vara. Denna information fick jag genom att använda mig av ett förminskat modellsystem, jag undersökte alltså bara en bit av hänglåssonden. När jag utrönat en fungerande öglestorlek överförde jag detta till fullstora hänglåssonder för att se om teorin fungerade även i mer komplexa system. Emellertid gjorde den inte det. Visserligen hade några hänglåssonder med öglor bearbetats av polymeraset, men till en sådan liten grad att bakgrundsbruset inte skulle minskas. Mina försök visade alltså att teorin med ögleförsedda hänglåssonder inte skulle hjälpa till att lösa problemet med bakgrundsbrus. Examensarbete i Biologi, 20 p, VT 2003 Institutionen för biologisk grundutbildning och Institutionen för Genetik och Patologi, Uppsala universitet Handledare: Olle Ericsson och Jonas Jarvius