nukleinsyrornas uppbyggnad
advertisement

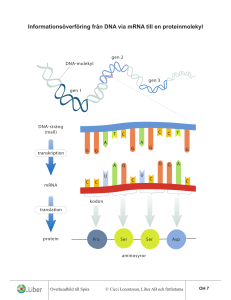
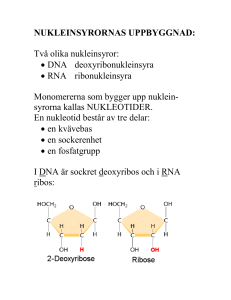
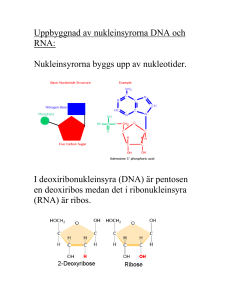
NUKLEINSYRORNAS UPPBYGGNAD: Två olika nukleinsyror: DNA deoxyribonukleinsyra RNA ribonukleinsyra Monomererna som bygger upp nukleinsyrorna kallas NUKLEOTIDER. En nukleotid består av tre delar: en kvävebas en sockerenhet en fosfatgrupp I DNA är sockret deoxyribos och i RNA ribos: I respektive nukleinsyra är det nukleotidernas ordning som står för den genetiska informationen som så småningom kan översättas till aminosyrasekvens. Det finns fem olika kvävebaser som delas in i två grupper efter deras struktur: I DNA finns: C T A G I RNA finns: C U A G Sockret och kvävebasen kopplas samman via en N-glykosidbindning – en nukleosid bildas: När en fosfatgrupp förestrats till C5 erhålls en nukleotid. Nukleinsyror bildas när nukleotider kopplas samman. Två nukleotider förbinds via fosfatgruppen i en FOSFODIESTERBINDNING. Det är C3 i ett socker som förbinds med C5 i nästa. En DNA- eller RNA-sekvens skrivs ALLTID 5’ 3’ så att inte missförstånd ska uppstå (precis som proteinsekvensen alltid skrivs N C). DNA är dubbelsträngat medan RNA är enkelsträngat. I DNA-helixen basparar adenin (A) med tymin (T) cytosin (C) med guanin (G) Notera; En DNA-helix består av två antiparallella nukleotidkedjor, den ena löper 5’ 3’ och basparar med en annan kedja som går 3’ 5’. Fosfatgrupperna och sockerenheterna sitter på utsidan och kvävebaserna inåt. En purin basparar alltid med en pyrimidin så helixens diameter blir hela tiden lika stor. Ett AT-baspar hålls samman av två vätebindningar och ett GC-baspar av tre. DNA-strukturen publicerades av Watson & Crick 1953. Dubbelhelixens geometri: Högervriden helix Eftersom en större pyrimidinbas alltid basparar med en mindre purinbas är innerdiametern på helixen hela tiden 11Å (1Å = 1·10-10 m) och ytterdiametern 20Å. Ett varv i dubbelhelixen är 34Å och innehåller 10 baspar. På ett varv finns en ”MAJOR GROOVE” och en ”MINOR GROOVE”. I dessa fördjupningar kan proteiner fästa. Utsidan av helixen är negativt laddad (fosfatgrupper) vilket gör att positivt laddade joner (katjoner) som Na+ och Mg2+ associeras till DNA:t in vitro. In vivo är DNA:t i cellkärnan uppvirat på positivt laddade proteiner, s k histoner, vilket ger effektiv tätpackning. Prokaryot DNA är cirkulärt medan eukaryot DNA är linjärt. Olika strukturvarianter av DNA: Bilder Stryer 4.14 och 4.16 - A-DNA - B-DNA - Z-DNA Cellulärt DNA: Vanligen B-DNA A-DNA: Kortare och bredare högervriden helix där basparen inte är vinkelräta mot helixaxeln. Ribosenheten har en annan konformation än i B-DNA. Hybridhelixar DNA-RNA som temporärt bildas har en struktur som påminner om A-DNA. Z-DNA: Vänstervriden helix. Uppkommer vid sekvenser med varannan purin och varannan pyrimidin som CGCGCG. Man har funnit proteiner som känner igen just sådana här sekvenser genom deras speciella struktur. Denaturering av DNA: Eftersom dubbelhelixen bara hålls samman av nonkovalenta bindningar (vätebindningar) räcker det att värma ett DNA-prov för att de två kedjorna ska separera. När DNA-kedjorna separeras säger man att DNA:t denatureras eller smälter. Vilken temperatur som krävs beror på andelen GCbaspar relativt AT-baspar. Om temperaturen sänks återbildas dubbelhelixen spontant. Man kan följa denatureringen spektrofotometriskt vid 260 nm. Denaturerat DNA absorberar mer ljus än den nativa formen eftersom kvävebaserna som svarar för absorptionen exponeras i denaturerat DNA. Även enkelsträngat RNA bildar väldefinierade tredimensionella strukturer som stabiliseras av vätebindningar. Vätebindningarna binder ihop olika delar av en och samma RNA-kedja. Ett vanligt motiv är s k hårnålsloopar: Molekylärbiologins centrala dogma: Replikation: Bassekvensen i DNA står för den genetiska informationen. När en cell ska delas måste DNA:t dupliceras – man måste få nytt DNA med exakt samma bassekvens som originalet. Vid proteinsyntes överförs informationen från DNA till aminosyrasekvens i två steg: Transkription: Bassekvensen i DNA översätts till bassekvens i mRNA. mRNA fungerar sedan som mall för proteinsyntesen. Translation: Bassekvens i mRNA översätts till aminosyrasekvens m h a tRNA. Processen sker på ribosomerna. En sekvens av tre baser utgör den GENETISKA KODEN och specificerar en viss aminosyra. REPLIKATIONEN: Först måste de två DNA-kedjorna separeras och sedan ska två nya kedjor bildas med originalen som mallar: Eftersom det i varje ny DNA-molekyl kommer att finnas en ny och en gammal DNA-kedja säger man att DNA-replikationen är SEMIKONSERVATIV. (DNA)n + dNTP (DNA)n+1 + PPi Reaktionen katalyseras av DNA-polymeraser. dNTP = dATP, dCTP, dGTP, dTTP Reaktionsmekanism för bildande av en ny fosfodiesterbindning: Hydroxylgruppen på C3 i ett socker gör en nukleofil attack på den innersta fosforatomen i nästa trifosfatnukleotid. Fosfaten är kopplad till C5 i nästa socker, alltså 3’,5’fosfodiesterbindning. Notera; Ny nukleotid binder alltså först till mallkedjan genom basparning och sedan sätts den nya kedjan ihop kovalent. Energikrävande process DNA-polymeraser kräver en primer för att kunna starta syntes av en ny kedja. Primern utgörs av en kort RNA-sekvens och det viktigaste strukturmotivet är en fri 3’-OHgrupp: Finns flera olika DNA-polymeraser men de viktigaste är: DNA-polymeras III: Främst ansvarig för polymerisationen av den nya kedjan. DNA-polymeras I: Kontrollerar att rätt nukleotid satts in. DNA-polymeras I har EXONUKLEAS-aktivitet vilket innebär att om ett fel upptäcks kan polymeraset klippa bort det och sätta in rätt nukleotid istället. Mycket viktigt eftersom fel vid replikationen kan få stora konsekvenser! Problem: DNA-polymeras katalyserar bildande av ny DNA-kedja 5’ 3’ och rör sig längs den gamla kedjan 3’ 5’. Men en av kedjorna i helixen exponeras ju 5’ 3’…. Lösning: Den ena kedjan syntetiseras diskontinuerligt i s k Okazakifragment. För varje Okazakifragment krävs en primer och för att sedan koppla ihop fragmenten krävs ytterligare ett enzym, DNA-ligas. Andra proteiner som är viktiga vid replikationen: Topoisomeras och helikas: Öppnar upp dubbelhelixen. ”Single strand binding proteins”: Binder till enkelsträngat DNA som temporärt bildas och skyddar det från nukleaser som annars attackerar enkelsträngat DNA och bryter ner det. Primas: Katalyserar bildandet av RNA-primers som behövs för att DNA-polymeraset ska kunna syntetisera ny kedja. Ligas: Kopplar ihop fragment kovalent. Ibland blir det dock fel vid replikationen: PUNKTMUTATIONER Tre typer: SUBSTITUTION: en kvävebas byts ut mot en annan DELETION: en bas tas bort INSERTION: en bas sätts till De två sista är allvarligast eftersom de förskjuter hela läsramen. Substitution är vanligast och finns i två varianter: *TRANSITION purin purin pyrimidin pyrimidin *TRANSVERSION purin pyrimidin pyrimidin purin Felfrekvens: 1/1010 nukleotider Kemiska mutagener: Ämnen som liknar de normala baserna och därför kan inkorporeras i deras ställe. Ex. 5-bromouracil. Ämnen som modifierar befintliga baser vilket leder till mutationer vid nästa replikation. Ex. HNO2. Ämnen som interkalerar, binder in mellan baserna, i DNA-strukturen och kan uppfattas som en bas vid nästa replikation. Ex. etidiumbromid. UV-ljus: UV-ljus kan leda till ihopkoppling av närliggande tyminer. Skydd: Reparationsmaskineri i tre steg: Felet upptäcks och felaktig bas tas bort – olika enzymer för olika fel. Ny korrekt bas sätts in av DNApolymeras I. Kedjan kopplas ihop av DNA-ligas. Flera cancersjukdomar orsakas av defekt DNA-reparationsmaskineri: Exempel: I sjukdomen Xeroderma pigmentosum saknas enzymet som tar bort tymindimererna som bildas vid exponering för UV-ljus. Patienterna får hudcancer och dör ofta innan de fyllt 30 år. TRANSKRIPTION OCH TRANSLATION = Översättning av bassekvensen till aminosyrasekvens OBS! I grova drag för prokaryota system - mycket mer komplicerat i eukaryota system! RNA: Tre huvudtyper: tRNA transfer RNA rRNA ribosomalt RNA mRNA messenger RNA Alla tre är involverade vid proteinsyntesen: DNA mRNA Protein Transkription Translation rRNA tRNA I eukaryoter har man även funnit ytterligare en variant, snRNA (small nuclear): Involverade vid ’splicing’. Funktioner hos RNA: rRNA: Bygger tillsammans med proteiner upp RIBOSOMER. På ribosomerna sker proteinsyntesen. tRNA: Transporterar aminosyror från cytosolen till ribosomen. Ser till att rätt aminosyra hamnar på rätt plats i den växande polypeptidkedjan. mRNA: Utgör mall för proteinsyntesen. mRNA bildas genom avskrivning av DNA-sekvensen. Strukturen hos RNA: tRNA: Minsta typen av RNA, ca 80 nukleotider. Intramolekylära vätebindningar gör att tRNA-molekylen blir klöverformad (2D). o Aminosyran som hämtas i cytosolen och transporteras till ribosomen binds till 3’änden av tRNA-molekylen. o I antikodon-loopen finns tre kvävebaser som basparar med mRNA och svarar för att aminosyrorna kommer i rätt ordning i proteinet. o Innehåller flera ovanliga, modifierade baser. Tredimensionellt viker sig sedan tRNA så den blir L-formad. I den tredimensionella formen hamnar bindningsstället för aminosyran så långt som möjligt ifrån antikodonet. Olika tRNA-molekyler binder olika aminosyror – aminosyran måste ”stämma” med antikodonet. rRNA: Stora molekyler som tillsammans med proteiner bygger upp ribosomer (1/3 av massan är RNA, 2/3 proteiner). Ribosomerna består av en mindre och en större subenhet (30S och 50S). mRNA: Bassekvensen i mRNA specificerar aminosyrasekvensen i ett protein. mRNA bildas när protein ska syntetiseras och bryts sedan snabbt ner för att nukleotiderna ska kunna återanvändas och för att cellen ska kunna kontrollera när ett visst protein syntetiseras. Bassekvensen i mRNA erhålls genom transkription (avskrivning) av bassekvensen i DNA. TRANSKRIPTIONEN: Transkriptionen katalyseras av RNApolymeras II, ett enzym som kopplar samman ribosnukleotider i riktning 5’ 3’. E.coli-enzymet består av fyra olika subenheter: 2’ -enhetens funktion är att hitta startstället, sedan dissocierar den och 2’ katalyserar själva polymerisationen. Mekanismen liknar DNA-polymerasets mekanism: Syntetiserar ny kedja 5’ 3’. 3’-OH gör nukleofil attack på innersta fosfaten i trifosfatnukleotiden varvid pyrofosfat lämnar. Tar instruktioner från en DNA-mall. Skillnader i funktion mellan DNA- resp. RNA-polymeras: RNA-polymeraset behöver ingen primer. RNA-polymeraset saknar exonukleasaktivitet. Gör inte lika mycket om det blir fel!!! Transkriptionen startar vid s k promoterregioner. Dessa regioner är AT-rika: -35-regionen Pribnow-boxen (TATA-boxen) För att RNA-polymeraset ska hitta rätt startställe krävs ett antal transkriptionsfaktorer; TFIIA, B, D, E, F, H. Bild 29.25 Stryer 1. TFIID binder till TATA-boxen via en av sina subenheter, TBP (’TATA-box binding protein’). Detta leder till konformationsförändringar. 2. TFIIA och TFIIB binder in till TBP:s yta. 3. Efter att TFIIB bundit byggs komplexet på med TFIIF, E och H och RNA-polymeras II varvid transkriptionen startar. Specifika funktioner: o TFIID hjälper polymeraset att finna promotorn. o TFIIF fungerar som ett helikas. Transkriptionen fortgår till en termineringssignal nås. Terminering: När en viss sekvens skrivits av viks RNA:t till en hårnålsloop som följs av flera U. TRANSKRIPTIONSBUBBLA: DNA-helixen öppnas och stängs med samma hastighet så hela tiden är ca 17 baser oparade. Ihopvikningen baseras på basparning. RNApolymeraset känner av veckningen och aktiviteten avstannar. När det gäller vissa gener krävs dock ytterligare en faktor för att avsluta transkriptionen, ett protein som kallas rho. Rho känner igen ytterligare sekvenser i det nysyntetiserade transkriptet, binder till RNA:t och detta i sin tur leder till att RNA-polymeraset lossnar. Notera; I båda fallen ovan så är det signaler i det nysyntetiserade RNA:t som känns igen och inte signaler i DNA:t! I prokaryoter translateras mRNA innan hela genen transkriberats medan det i eukaryoter bildas ett primärt transkript som processas innan färdigt mRNA lämnar kärnan för transport till ribosomerna i cytosolen. Bild 29.21 Stryer ’SPLICING’: De primära transkripten i eukaryoter består av EXONER och INTRONER. o Exoner: Kodande genmaterial o Introner: Icke-kodande genmaterial Processen då introner avlägsnas och exoner sammanfogas kallas ’splicing’. Varför introner? Experimentverkstad för evolution? Mekanism: En intron startar alltid med GU och slutar med AG. De har också alltid en intern förgreningssite med ett A. Bild 29.34 Stryer Stegvis: Bild 29.35 Stryer 1. 2’-hydroxylgruppen på A i förgreningssitet gör en nukleofil attack på fosforn i fosfodiesterbindningen som förbinder exon 1 och intronen. Bindningen bryts. 2. 3’-hydroxylgruppen på exon 1 attackerar fosfodiesterbindningen mellan intronen och exon 2 varvid en ny fosfodiesterbindning bildas mellan exon 1 och exon 2. Katalyseras av ett komplex, SPLICEOSOMEN, som består av proteiner och snRNA. OBS! Även RNA kan ha katalyserande roll! Alternativ splicing: Viktigt för ett begränsat genmaterial kan ge upphov till många olika proteiner. Typexempel: Antikroppsproduktion Notera; En stor andel av alla genetiska sjukdomar orsakas av mutationer som påverkar var gener ’splicas’. TRANSLATIONEN: Bassekvensen i mRNA överförs till aminosyrasekvens. mRNA-sekvensen kan översättas till aminosyrasekvens via den s k GENETISKA KODEN. Varje aminosyra motsvaras av en eller flera bastripletter. De tre baserna kallas med ett ord för ett kodon. 4 baser kan kombineras och ge 43=64 olika tripletter. 61 av dessa kodar för aminosyror medan 3 koder är STOPP-koder: UAG, UGA, UAA. 20 aminosyror – 61 koder: Flera aminosyror har flera koder genetiska koden är degenererad. Uppgifter: Ange vad följande kodoner motsvarar för aminosyror: AAG CCU GAG Ange alla koder för: asparagin serin Notera; - De olika koderna för en aminosyra liknar varandra vilket minskar risken för allvarliga effekter av mutationer. - Antalet koder är korrelerat till hur vanlig respektive aminosyra är i proteiner. Varför är genetiska koden degenererad? Om alla aminosyror bara hade en kod: 20 kodon 44 stoppsignaler Risken är mycket hög att ett protein blir inaktivt om translationen avbryts för tidigt. Ett protein med en utbytt aminosyra har oftast viss aktivitet om det inte är någon av de katalytiska aminosyrorna som ersatts. Genetiska koden är nästan universell: Vissa variationer har dock upptäckts i mitokondrie-DNA. Mitokondrierna har bakteriellt ursprung men upptogs av en eukaryot värd mycket tidigt i evolutionen. Kodon – antikodon: tRNA interagerar antiparallellt med mRNA: Uppgifter: 1. Kodon: GCA Antikodon? 2. Ange antikoden för en tRNA som bär metionin. Translationen stegvis: 1. Aktivering av aminosyran – koppling till tRNA via aminoacyl-tRNA-syntetas. Energi krävs. Aminoacyl-tRNA-syntetaser = Enzymer som kopplar aminosyran till rätt tRNA. Aminosyra + ATP aminoacyl-AMP + 2 Pi Aminoacyl-AMP + tRNA aminoacyl-tRNA + AMP Varje aminoacyl-tRNA-syntetas är specifikt för en viss aminosyra. Garanti för specificitet; o Aktiva ytan utformad för att passa en viss aminosyra. o Finns ett editeringssite – undersöks ytterligare en gång att det är rätt aminosyra som kopplats på. Enzymet måste även välja rätt tRNA. Vissa känner igen sina tRNA-partners enbart på deras antikodon medan andra även kräver närvaro av vissa sekvenser nära vinkeln i ”L-et”. 2. Initiering av polypeptidsyntesen Syntesriktning N C mRNA:t läses 5’ 3’ OBS! Beskrivning av prokaryot system – mer komplicerat i eukaryoter! Peptidkedjan börjar alltid med formylmetionin, fMet: Metionin har koden AUG vilket alltså också fungerar som START-kod. Start-AUG märks ut genom att det finns en purinrik sekvens (A+G) i mRNA:t, SHINEDALGARNO-sekvensen, som föregår ett startAUG och skiljer det från övriga AUG. Sekvensen finns på 5’-sidan, ca 10 nukleotider från start-AUG. När fMet bundit till mRNA binder komplexet till den mindre ribosomsubenheten som då associeras med den större. För detta krävs energi samt flera initieringsfaktorer. Bild 30.20 Stryer 1. I vilande läge bildar den lilla ribosomenheten ett komplex med IF1 och IF3. Syftet är att förhindra interaktion med den stora ribosomenheten i frånvaro av mRNA. 2. IF2 bildar ett komplex med formylmetioninladdad tRNA och mRNA och binder sedan in till den lilla ribosomenheten varvid IF1 och IF3 lossnar. 3. GTP bunden till IF2 hydrolyseras vilket leder till konformationsförändringar och association med den stora ribosomenheten. IF2 dissocierar. Translationen kan starta…. Shine-Dalgarnosekvensen (A+G) basparar med pyrimidinrik (C+U) sekvens i rRNA och positionerar på det sättet mRNA:t rätt för translationsstart. Start-AUG ska exponeras i P-sitet. Tre bindningssite för tRNA på ribosomen: A – aminoacylsitet P – peptidylsitet E – exit 3. Polypeptidkedjans förlängning Stegvis: Bild 30.23 Stryer En elongeringsfaktor och GTP (energi) krävs för förlängningsreaktionerna. 1. En tRNA-molekyl som bär den växande polypeptidkedjan (eller i första varvet formylmetionin) sitter bunden i P-sitet. 2. En ny tRNA laddad med aminosyra binder in till A-sitet. Nu kollas att kodonet som exponeras i A-sitet stämmer överens med antikodonet på tRNA:t. 3. Den växande polypeptidkedjan på tRNA:t som sitter i P-sitet överförs till aminosyran i A-sitet varvid en ny peptidbindning bildas vid det s k peptidyltransferascentret, som är en del av den stora ribosomenheten. 4. Elongeringsfaktorn EF-G med bunden GTP påverkar ribosomen så när GTP hydrolyseras till GDP ändras formen på EF-G vilket leder till en TRANSLOKERING. tRNA:t med den växande polypeptidkedjan flyttas från A-sitet till P-sitet och det fria tRNA:t flyttas till E-sitet. Samtidigt flyttas mRNA:t framåt motsvarande 3 baser så att ett nytt kodon exponeras i A-sitet. 5. Det fria tRNA:t lämnar E-sitet för att hämta ny aminosyra i cytosolen. Nu kan processen börja om och en ny tRNA med aminosyra binda in i A-sitet. Detta upprepas till ett STOPPKODON visas i A-sitet. Mekanismen för bildande av en ny polypeptidbindning: Bild 30.22 Stryer Kvävet i aminogruppen på aminosyran bunden till tRNA i A-sitet gör en nukleofil attack på karbonylkolet hos aminosyran bunden till tRNA i P-sitet. 4. Terminering: Speciella ”RELEASE”-faktorer känner igen stoppkoderna UAA, UGA och UAG och binder till dem. - Blockerar för ny aminoacyl-tRNA i Asitet. - Binder en vattenmolekyl och när syret i vattenmolekylen gör en nukleofil attack på karbonylkolet som fäster polypeptidkedjan till tRNA så bryts bindningen mellan den C-terminala aminosyran och tRNA:t. Hela ribosomkomplexet dissocieras och polypeptidkedjan veckas till sin tredimensionella form. Viktiga skillnader vid eukaryot translation: o Eukaryota ribosomer är större; 40S + 60S o Den första aminosyran är metionin, inte formylmetionin. Dock finns speciell tRNA som binder den första metioninen. o Finns ingen Shine-Dalgarno-sekvens utan det AUG som är närmast mRNA:ts 5’ände fungerar som start. Notera; mRNA i prokaryoter kodar för mer än ett protein, i eukaryoter motsvarar en mRNAmolekyl = ett protein. o Eukaryoter använder flera initieringsfaktorer. o Termineringen sköts av en ”release”faktor jämfört med två hos prokaryoter. ANGREPPSPUNKTER FÖR ANTIBIOTIKA!!! -Streptomycin interfererar med inbindning av formylmetionin-laddad tRNA -Erytromycin binder till stora ribosomenheten och förhindrar translokering Resistensutveckling – nya strategier?????