Biochemistry 6/e
advertisement

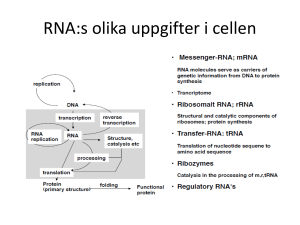
Nukleinsyror: DNA och RNA Kurs MNXA10/12 Hans-Erik Åkerlund (Mod från Henrik Stålbrand) DNA: ”deoxy-ribonucleic acid”, deoxi-ribonukleinsyra Bärare av cellens arvsanlag RNA: ”ribonucleic acid”, ribonukleinsyra Tillfällig bärare av arvsanlag (mRNA) Struktur för proteinsyntesmaskineriet, ribosomen (rRNA) Bärare av aktiverade aminosyror (tRNA) Övrigt: Bärare av arvsanlag, katalys, RNA-världen DNA struktur Dubbelhelix Stor fåra (major groove) Liten fåra (minor groove) Nukleinsyror är Polymerer med Nukleotider som byggstenar Nukleotid: Fosfat Kvävebas Socker Flera nukeotider bildar en polymer, en polynukleotid, dvs en sträng av RNA eller DNA Polymerens stomme: ....fosfat-socker-fosfat-socker.... Fosfat: grund till ”syra” (acid) i nukleinsyra, dvs A i DNA och RNA Nukleotid Sockret R i RNA D i DNA Syre saknas Deoxy Kvävebaser: RNA Adenin Guanin Cytosin Uracil DNA Adenin Guanin Cytosin Thymin Kvävebaser Plan struktur Plan struktur Endast i RNA, H byts ut mot deoxyribos i DNA = och mot ribos i RNA Endast i DNA En av fyra nukleotider - ATP Utgångsmaterial vid syntes av RNA Fosfat Adenin Ribos Fosfat Kvävebas Socker Polynukleotider (nukleinsyror) Riktning för syntes 5’ till 3’ Nukleotidenehet syra binder till alkohol = esterbindning här fosfodiesesterbindning DNA struktur. 1) DNA sträng: polynukleotid, kvävebaser A,G,C,T 2) DNA sträng: har polaritet: 5´ o 3´ ändar 3) Dubbel strängat (ds) DNA: basparning mellan två antiparella strängar 4) H-bindingar, 2 st mellan A o T, 3 st mellan G o C 3´ 5´ 5´ 5´ A G C T C T C G A G 3´ 3´ DNA dubbelhelix struktur Basparning: Plana strukturer G o C (3 vätebindningar) A o T (2vätebindningar) socker Vätebindning socker Dubbelsträngad DNA (dsDNA) bildar dubbel helix (spiral) motriktade kedjor 3´ 5´ A G C T C T C G A G 3´ 5´ 5´ 3´ 3´ 5´ DNA dubbelhelix 5´ 3´ 10 nukleotider Stor fåra (major groove) eller Liten fåra (minor groove) Baspar (bp) Mörk färg: fosfodiester o socker (huvudkedja) Ljus färg: basparade kvävebaser 3´ 5´ DNA dubbelhelix socker baser fosfodiester (-) Stabilisering av dubbel helix. Kovalenta bindningar Vätebindningar mellan baser Hydrofob effekt Van der Waals/dispersionskrafter Fosfatgrupperna Negativt laddade ökar avstånd och minskar risk för hydrolys Avsaknad av 2’-syret på ribos minskar risk för hydrolys RNA Ribos istället för deoxyribos G-C och A-U i stället för G-C och A-T Normalt enkelsträngat men kan vecka sig tillbaks på sig själv och bildar då dubbelhelix där basparningen stämmer mRNA, rRNA, tRNA, snRNA, siRNA tRNA rRNA Hair pin loop Cellulära processer Replikation DNA Transkription Translation RNA PROTEIN Process Huvudsaklig funktionell enhet/ enzym Replikation DNA polymeras. (DNA templat, mall) Transkription RNA polymeras. (DNA templat eller ) Translation Ribosom, består av rRNA, protein. (mRNA templat) Replikation: kopiering av DNA Enzym: DNA-polymeras 3´ 5´ 5´ DNA templat 3´ Ny DNA sträng DNA replikation 5 3 5 3 3 5 5´ 5´ 3´ 3´ Transkription DNA avläses till mRNA med enzymet RNA-polymeras Syntes 5´ till 3´ ”Sense strand” Promotor = RNA-polymeras inbindning. Translation mRNA AUG CCG XXX XXX XXX UGA 3´ 5´ protein N Translation Met Pro C Gentiska koden 5´ 3´ Translation Ribosom inbindning Ribosom= organell för proteinsyntes (rRNA, protein) Stop kodon Initierings kodon mRNA AUG CCG XXX XXX XXX UGA 3´ 5´ polypetid Met Pro N Translation C Translation Aminosyra 1 Met Ribosom= organell för proteinsyntes (rRNA, protein) tRNA 3´ Antikodon 3´UAC 5´ mRNA AUG CCG XXX XXX XXX UGA 5´ 3´ Kodon Translation Aminosyra 2 Aminosyra 1 Met Pro N-Met-Pro... tRNA Syntes av Peptidbindn. 3´GGC 5´ 3´UAC 5´ mRNA AUG CCG XXX XXX XXX UGA 5´ 3´ Gen expression (uttryck) DNA 5´ 3´ ATG CCG TTT GTA ... ... ... TGA 3´ 5´ Transkription mRNA AUG CCG UUU GUA ... ... ... UGA 3´ 5´ Translation polypetid Met Pro Phe Val ... .... ... N C Veckning protein FUNKTION! C N