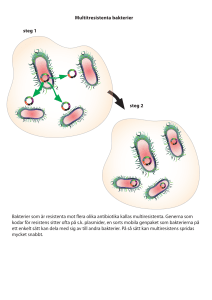
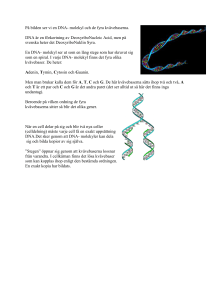
Laborationer i Cellbiologi med biokemi Lab 6-10
advertisement

Laborationer i Cellbiologi med biokemi Lab 6-10 Biomedicinprogrammet, termin 2 VT 2013 2 Innehållsförteckning Ordnings- och säkerhetsföreskrifter 3 6: Odling av bakterier 4 7: Polymerase Chain Reaction (PCR) 9 8: Kloning av DNA 24 9: Sekvensering och bioinformatik 37 10: Kvantitativ (q) omvänd (R) transkription (T) PCR (qRT PCR) 46 3 Ordnings- och säkerhetsföreskrifter 1. På laboratoriet skall skyddsrock användas. Använd skyddsglasögon vid risk för stänk. 2. Rökning, ätande eller dylikt får inte förekomma på laboratoriet. Gäller även snusning. 3. Handtvättning skall ske innan laboratoriet lämnas. 4. Före och efter arbetet torkas bänken av med fuktat papper samt spritas. 5. Betrakta använt material som potentiell smittorisk. Behandla laboratoriebakterierna som om de vore patogena. Vid spill av bakterier torka torrt med papper och tvätta sedan med sprit. 6. Smittförande avfall (dvs. bakterier och eukaryota celler) kastas i där för avsedda riskkartonger (GUL märkning). Trasigt glas samt stickande avfall läggs i lämplig burk och kastas i riskkartonger (BLÅ märkning). Kemiskt eller radioaktivt avfall placeras i där för avsedda behållare. Annat avfall kastas i papperskorgarna. 7. Tag bort märkning från använt glasmaterial och skölj i vatten. 8. Använd endast brännare för att bränna av bakterieöglor. Inte för värmning av lösningar och buffertar. 9. Gör dig bekant med brand-­‐‑ och räddningsutrustning som finns vid/i laboratoriet. 10. Informera dig om användandet av ev. hälsovådliga ämnen i laborationerna. 11. Innan Du går hem för dagen, kontrollera att vattenbad, skakar och gaslågor är avstängda. 12. Varje person i gruppen skall föra journal över laborationsarbetet. Arbetet på laboratorium samt arbetet med gemodifierade organismer (GMO) är reglerat enligt svensk och europeisk lagstiftning. Vi hänvisar bl.a. till Arbetarskyddsstyrelsens författningssamling AFS1997:10 ”Laboratoriearbete med kemikalier” samt AFS 2000:5 ”Innesluten användning av genetiskt modifierade mikroorganismer”. Riskbedömning av arbetet samt skadeförebyggande åtgärder skall genomföras innan arbetet påbörjas. Vid frågor eller oklarheter kring arbetets risker skall du tala med labammanuenserna. 4 6: Odling av bakterier Dag 1: Dag 2: • • • • Gjutning av agarplattor Göra spädningsserie av en bakteriekoloni Sprida bakterier på platta (renstryck och rackling) Räkna bakteriekolonier på plattor Teori Odling och hantering av bakterier är grundläggande för all molekylärbiologisk metodologi. En av de vanligaste bakterierna som används på labb är Escherichia coli (E. coli). E. coli växer bra i flytande kultur (rör eller sk. batch-­‐‑kultur) eller på agarplattor. Flytande kulturer bör odlas under skakning. Bakterierna kan växas i olika typer av näringslösning eller sk. medium. De flesta medier innehåller extrakt av döda jäst-­‐‑ och/eller bakterie-­‐‑celler (trypton/peptone). Olika tillsatser som exempelvis antibiotika (ampicillin eller kanamycin) är vanligt förekommande. Bakterier som växer på platta bildar kolonier (kolonier ser ut som små prickar). En koloni härstammar från en enskild bakterie som delat sig och gett upphov till ”kloner” av sig själv. Ensklida kolonier, ”kloner”, kan sedan plockas för vidare bruk. Det finns olika sätt att få enskilda kolonier. Två metoder att åstadkomma separata kolonier exemplifieras i laborationen: Renstrykning av bakterie på agarplatta samt spridning med glaskulor /rackling på agarplatta. Särskilda instruktioner Momenten utförs individuellt av samtliga gruppmedlemmar. Betrakta bakterier som potentiellt patogena. Börja därför med att studera de allmänna instruktionerna för arbete med bakterier och smittfarligt material. Handskar behöver inte användas, men handtvätt efter labben är obligatorisk. Laborationsrapport -­‐‑ -­‐‑ -­‐‑ -­‐‑ -­‐‑ -­‐‑ Rapporten lämnas in gruppvis men de enskilda resultaten redovisas, i tabellform där det är möjligt. Förklara enkelt teorin bakom bakterieodling: mediuminnehåll och spridning av bakterier. Beskriv kortfattat resultatet av renstrykningen i en figur. Ange hur många kolonier det fanns på respektive spädning/platta. Ange hur många bakterier det fanns i den plockade kolonin och beskriv hur beräkningen av antalet bakterier i kolonin gått till. Glöm inte skriva namn och gruppnummer på rapporten! 5 Dag 1: Gjutning av plattor, spridning av bakterier Gjutning av agarplattor Laborationen börjar med preparation av TYE-­‐‑bakteriemedium samt TYE-­‐‑agarplattor. Alla grupper skall tillverka TYE-­‐‑agar, men endast en grupp per labb gör flytande TYE-­‐‑medium till resten av kursen. Denna grupp utses av labhandledare. Beräkna volymen bakteriemedium samt antalet agarplattor som behövs under hela laborationsblocket (Lab 6 och 8). Notera om plattorna skall innehålla ev. tillsatser som antibiotika. Lägg till en liten säkerhetsmarginal om en eller annan platta skulle förolyckas. Räkna med 15 ml agarmedium per 9 cm-­‐‑platta. Gör medium/ agar i autoklaverbara Schott-­‐‑flaskor med skruvlock. Vid tillverkning av agar är det viktigt att inte fylla mycket mer än halva flaskan (~0,6 l) inför autoklaveringen. Använd flera flaskor om det är nödvändigt. Material -­‐‑ Plastpetriskålar: färglösa samt röda -­‐‑ Ampicillin (stocklösning 100 mg/ml); slutkoncentration i agar: 100 µμg/ml -­‐‑ Autoklaverbara 1l Schott-­‐‑flaskor -­‐‑ dH20 (destillerat H2O) TYE-­‐‑agar TYE-­‐‑medium (en grupp gör detta till hela kursen) NaCl 8 g/l NaCl 8 g/l Peptone 10 g/l Peptone 10 g/l Jäst extrakt 5 g/l Jäst extrakt 5 g/l Agar 15 g/l Väg upp kemikalier och fyll upp med destillerat vatten (ur ljusgrön vattenkran på lab) till rätt volym (använd mätkolv). Agarn kommer inte att lösa sig på egen hand utan måste smältas. Därför skall lösningarna autoklaveras, vilket även steriliserar lösningen (dvs. dödar ev. bakteriekontamination). Drag inte åt locken under autoklaveringen pga. det ökade trycket. Autoklav finns i på labbet och lösningarna autoklaveras gemensamt av handledare. -­‐‑ Under tiden agarn autoklaveras utförs första momenet på lab 7. Låt agarn svalna något innan plattorna ska tillverkas. Häll agar till markeringen i färglösa petriskålar för agar utan tillsatser, samt i röda skålar för agar innehållandes ampicillin. Ev. bubblor tas bort med med hjälp av glödgad platinaögla eller punkteras med en pipettespets. Gjut först plattor utan ampicillin, tillsätt därefter ampicillin (slutkoncentration 100 µμg/ml) till resten av innehållet i flaskan (uppskatta volymen). OBS! Tillsätt ampicillinet först då agarn svalnat så att det går att ta i flaskan (ca 50°C) eftersom det är värmekänsligt. Jobba relativt fort och håll agarflaskan i rörelse så att inte agarn svalnar och börjar stelna utmed kanterna (sker vid 38°C). Lägg locket på glänt medan agarn svalnar för att undvika rinnande kondens. Var noga med att sedan lägga på locket och lämna plattorna inte öppna i nödan då de lätt kan kontamineras av bakterier i luften. Förvara de stelnade plattorna upp och ner för 6 att undvika att de torkar ut och för att samla eventuell kondens i locket och inte på agarytan. Beräkning av antalet bakterier i en koloni Material (per person): -­‐‑ 1 st platinaögla samt brännare -­‐‑ 5 st sterila 1,5 ml Eppendorfrör -­‐‑ Skruvlocksrör med sterilt TYE-­‐‑medium -­‐‑ Rör med sterila 3mm glaskulor -­‐‑ 4 st TYE-­‐‑agarplattor utan ampicillin (ofärgad petriskål) -­‐‑ Agarplatta med bakteriekolonier Hantering och inkubering av sterila agarplattor: plattor skall alltid hanteras upp och ned när locket är på. Ev. kondens kommer då att hamna i locket och inte på agarytan och kondensen kan då hällas av. Från amanuenserna får ni en platta med bakteriekolonier. En stor koloni plockas med platinaögla eller pipettespets och förs till ett Eppendorfrör som gjorts iordning i förväg med 1 ml TYE medium. Se till att hela kolonin kommer med. Märk röret ”0”, förslut röret och blanda noggrant under någon minut. En spädningsserie görs på innehållet i röret: 1:100, 1:1000, 1:10 000 samt 1:100 000 enligt figuren nedan. Byt spetsar på pipetten mellan varje rör! Pipettera upp 990µμl TYE-­‐‑ medium till ett Eppendorfrör märkt ”1”. Pipettera upp 900µμl TYE-­‐‑ medium till 3 stycken Eppendorfrör märkta ”2 -­‐‑ 4”. Pipettera sedan 10µμl från rör ”0” till rör ”1”, förslut röret, skaka och pipettera 100µμl från rör ”1” till ”2”. Fortsätt spädningsserien med att pipettera 100µμl från rör ”2” till rör ”3” osv. Var noga med att blanda om rören efter varje steg. Blanda rören igen och pipettera 100µμl av resp. spädning (rör ”1-­‐‑4”) till märkta agarplattor som försetts med 5-­‐‑10 sterila glaskulor. Locket sättes på och plattorna med respektive spädning snurras runt (racklas) så att bakterierna fördelas jämt över agarytan i plattan m.h.a. glaskulorna. Glaskulorna hälles sedan ut i uppsamlings-­‐‑ bägare innehållande desinfektionsmedel. Plattorna inkuberas upp och ner i 37°C över natt. 100µl 1 koloni 100µl 100µl 10µl 0 1 2 100µl 7 3 4 Renstrykningsövning Material: -­‐‑ 1 st platinaögla och brännare -­‐‑ TYE-­‐‑ agarplattor utan tillsatser (ofärgad petriskål, 1 st per person) -­‐‑ 1 st rör med bakterier (från amanuenserna) Märk upp en vit TYE-­‐‑agarplatta med namn, gruppnummer och laborationsnummer i botten längs kanten. Agarplattor skall alltid märkas på botten eller på sidan och inte på locket för undvika att skålarna förväxlas. I just denna lab är det viktigt att ni märker skålarna på sidan av botten-­‐‑skålen, för att ni senare ska kunna se och räkna kolonierna genom att titta på undersidan av plattan. Placera den märkta plattan upp och ned på bänken. Sterilisera platinaöglan genom att bränna av den i öppen låga, tills den glöder fullständigt. Lyft upp plattan utan lock (låt locket ligga på bordet) och kyl av öglan i agarn i kanten på plattan. Kyler man inte av öglan på detta sätt innan man doppar den i bakteriekulturen kommer bakterierna att dö. Lägg tillbaka plattan i locket. Doppa öglan i bakteriekulturen, sätt tillbaka locket och ställ ifrån dig röret. Fatta plattan igen och gör ett utstryk på ytan av agarn (enligt figuren nedan ”område 1”). Ytan skall ej repas. Öglan bränns av på nytt tills den glöder och kyls på liknande sätt. Ett andra stryk utan att doppa öglan i kulturen görs vinkelrätt mot det första (se figuren ”område 2”). Öglan bränns av och kyls och ett tredje stryk görs vinkelrätt mot det andra på liknande sätt (”område 3”). Varje person i gruppen skall göra en renstrykning som placeras i 37°C inkubator över natt. 1 2 3 8 Dag 2: Räkna bakteriekolonier Beräkning av antalet bakterier i en koloni, fortsättning Material: -­‐‑ Dina agarplattor med spädningsserien och renstrykningen. -­‐‑ En vattenfast tuschpenna med fin spets. Kontrollera dina agarplattor med bakterier. Räkna antalet bakterier på respektive platta i spädningsserien. Använd en tuschpenna och sätt en punkt för varje räknad koloni på undersidan av plattan. Dela in plattan i sektorer vid stort antal kolonier. Notera om antalet kolonier (bakterier) stämmer överens med spädningarna (faktor 10 mellan plattorna). Om det är omöjligt att räkna någon platta, försök uppskatta antalet bakteriekolonier. Varje person i gruppen ska beräkna antalet bakterier i sin ursprungskoloni. Utgå ifrån den spädning som gav 100 -­‐‑ 500 kolonier på en platta. Beakta spädningsfaktor och volym som tillsattes på plattan. Studera även plattan med renstrykningen. Finns det enskilda kolonier som man skulle kunna plocka? Var bakteriekulturen ren eller kan bakterier med olika koloniutseende urskiljas? Ser din platta likadan ut som dina bänkgrannars? 9 7: Polymerase Chain Reaction (PCR) Dag 1: Dag 2: Dag 3: Själva: • • • • • • • Lysera levervävnad Preparera genomiskt DNA Mäta DNA-koncentration (spektrofotometri) Blanda PCR-reagenser, köra PCR Gjuta agarosgel Köra ut PCR-reaktionen på gel Primer design Teori PCR, "ʺpolymerase chain reaction"ʺ, är kanske den enskilt viktigaste molekylär-­‐‑ biologiska metoden och används inom ett stort antal applikationer från isolering av nya gener till klinisk diagnostik. Styrkan i metoden är dess oerhörda känslighet och att det är möjligt att amplifiera (mångfaldiga) DNA med utgångspunkt t.o.m. från enstaka DNA-­‐‑molekyler. I denna laboration kommer ni designa PCR-­‐‑primers för att sedan amplifiera ett specifikt DNA-­‐‑fragment från hela kycklinggenomet med hjälp av PCR. Ni kommer att bekräfta att ni har amplifierat ett DNA-­‐‑fragment av rätt storlek med hjälp av agarosgelelektrofores. Särskilda instruktioner Momenten utförs i grupper om 2-­‐‑3 personer. Etiduimbromid (EtBr) är mutagen och måste hanteras med försiktighet i dragskåp och med handskar. Handskar måste även användas när ni jobbar med PCRen för att inte kontaminera proverna eller reagensen. Taq-­‐‑polymeraset får inte värmas upp och måste hela tiden förvaras i frysblocket. Laborationsrapport Rapporten lämnas in gruppvis. Besvara de specifika frågorna som står i slutet och beskriv även i korta ordalag vilka resultat ni fick. Om ni fick avvikande och/eller felaktiga resultat ska ni göra en felanalys där ni anger troliga förklaringar till varför ni fick dessa. 10 Dag 1: Lysering av kycklinglever Lysering av kycklinglever Denna del av lab 7 utförs samtidigt med lab 6. Frusen lever tinas i lyseringsbuffert innehållande Proteinas K. Proven inkuberas vid 55°C och vävnaden bearbetas mekaniskt varvid vävnaden bryts ner och det genomiska DNAt frisätts. Material: -­‐‑ -­‐‑ -­‐‑ -­‐‑ -­‐‑ Eppendorfrör Rör med frusen levervävnad från kycklingembryo Lyseringsbuffert1 Proteinas K, 10 mg/ml Värmeblock, 55°C Utförande: 1. 500µμl lyseringsbuffert pipetteras till ett Eppendorfrör med en bit frusen vävnad från kycklingembryo. 2. 2,5µμl Proteinas K pipetteras till röret och lösningen blandas. 3. Röret inkuberas i värmeblock vid 55°C över natt. (Proverna blandas ytterligare några gånger av labhandledare). 1 Lyseringsbufferten innehåller: -­‐‑ 100 mM Tris-­‐‑HCL, pH 8.5 -­‐‑ 5 mM EDTA -­‐‑ 0,2% SodiumDodecylSulphate (SDS) -­‐‑ 200 mM NaCl 11 Dag 2: Preparation av DNA och PCR-körning Preparation av genomiskt DNA DNAt ifrån den lyserade kycklinglevern fälls ut till ett salt med etanol och samt centrifugeras ned med hjälp av en mikrocentrifug. DNAt löses upp i vatten och en del används för att mäta DNA-­‐‑koncentrationen. Material: -­‐‑ Eppendorfrör -­‐‑ Värmeblock, 55°C -­‐‑ Etanol 99,5%, kall, förvaras i –20°C frys. -­‐‑ Etanol 70%, kall, förvaras i –20°C frys. -­‐‑ MQ H2O Om vävnaden inte är helt upplöst: 1. Använd en 1000µμl-­‐‑pipett och pipettera innehållet i röret långsamt upp och ned tills vävnadsbiten är uppluckrad. Inkubera sedan i ytterligare 10 minuter vid 55°C. Lösningen bör vara något viskös och börja klarna. Annars, fortsätt här: 2. Centrifugera röret i mikrofug i 5 min. 10 000 rpm. (10K) för att bli av med olösligt material. 3. För över 400µμl av supernatanten till nytt ett rör (undvik att få med något av pelleten) 4. Tillsätt försiktigt 800µμl kall 99.5% etanol och vänd röret långsamt. En liten tuss av vitt DNA skall falla ut i interfasen mellan etanol och vattenfas. Vänd röret helt upprepade gånger. 5. Centrifugera ner DNAt i mikrocentrifug 5 minuter, 10K (Med gångjärnet uppåt så att du vet var pelletten hamnar -­‐‑ Eppendorfrör skall alltid sättas i rotorn på detta sätt). Sug försiktigt bort supernatanten med en pipett. 6. Tillsätt 1ml 70% etanol för att tvätta pelleten och vortexa (löser inte alltid upp sig). Centrifugera 30 sek. 10K (med samma orientering på röret). Sug försiktigt bort lösningen. 7. Låt provet lufttorka (röret kan ställas upp och ned) i 10-­‐‑15 minuter. Lös upp pelleten i 100µl MQ H2O. 8. För att lösa DNAt sätt det i 55°C värmeblock ca 20 minuter. Blanda om DNAt med jämna mellanrum genom att slå försiktigt på röret med fingret. 9. Centrifugera i 5 minuter 10K för att spinna ner olöst material och för över supernatanten i ett nytt Eppendorfrör som märks tydligt med gruppnummer och innehåll. 12 Spektroskopisk kvantifiering av genomiskt DNA Ett spektrum tas på DNA-­‐‑lösningen och koncentrationen beräknas med utgångspunkt från absorbansen vid 260nm. DNAt används sedan som templat i PCR-­‐‑reaktionen. Material: -­‐‑ Spektrofotometer (Nano-­‐‑Drop) -­‐‑ MilliQ H20 -­‐‑ Eppendorfrör -­‐‑ Prov med genomiskt DNA från punkt 9 ovan. Utförande: 1. Späd en del av DNA-­‐‑provet i ett Eppendorfrör (1:10). Tag 5µμl DNA till 45µμl MQ H20. 2. Mät både ert spädda prov och ursprungsprovet i Nano-­‐‑Drop:en och notera absorbansen vid 260 och 280 nm. Vid behov kan ni späda provet ytterligare. 3. Beräkna DNA-­‐‑koncentrationen med hjälp av Lambert-­‐‑Beer'ʹs Lag2. Notera DNA-­‐‑koncentrationen och beräkna vilken volym som behövs för att ta 1µμg DNA. Använd det utspädda DNAt som templat för PCR-­‐‑reaktionen. 4. Beräkna den totala mängden DNA ni fått vid preparationen. Absorbans vid 260nm (spädning): Absorbans vid 280nm (spädning): Ratio 260/280: Beräknad DNA koncentration (spädning): Beräknad DNA koncentration (prov): Volym som krävs för 1µμg DNA från spädning: Lambert-­‐‑Beer lag: A=ε x l x c A: absorbans ε: extinktionskoefficient (0,02 ml µμg-­‐‑1cm-­‐‑1) l: kyvettlängd (cm) = 1 (obs. att kyvettlängen är omräknad jmf m Nanodrop) c: koncentration (µμg/ml). 2 13 PCR-amplifiering från genomiskt DNA Genomiskt DNA från tidigare moment används som templat för att amplifiera ett DNA-­‐‑fragment m.h.a. av två specifika primers. PCR-­‐‑reaktionen blandas ihop och PCR-­‐‑maskinen körs övernatt. För enkelhetens skull har en PCR-­‐‑mix gjorts iordning innehållande buffert, dNTP, MgCl2 samt primers. En negativ kontroll skall köras parallellt med provet. Den negativa kontrollen innehåller allt utom DNA-­‐‑templaten (det genomiska DNAt). Material: -­‐‑ PCR-­‐‑rör -­‐‑ MQ H20 -­‐‑ Templat: genomiskt DNA från tidigare moment -­‐‑ Taq DNA polymerase (AmpliTaqGold 2,5units/µμl) -­‐‑ PCR mix3 (2.5x stocklösning) OBS! När ni sätter upp PCR-­‐‑reaktionen ska ni ha handskar för att undvika att kontaminera proverna; arbeta noggrant och försiktigt! Pipettera ihop PCR-­‐‑reaktionerna i två märkta PCR-­‐‑rör enligt nedan: Prov och kontroll. X= volymen av ert genomiska DNA som ni räknat fram (29,5-­‐‑X)µμl H20 MQ 20µμl PCR mix 1µμg (Xµμl) genomiskt DNA-­‐‑templat eller Xµμl MQ H20 för kontrollreaktionen 0,5µμl enzym AmpliTaq Gold 50µμl totalvolym Förslut rören noggrant och blanda röret lätt med handen. Skaka eller centrifugera ner innehållet i spetsen på rören. Amanuenserna startar sedan PCRen som körs över natt. Följande PCR-­‐‑program med ”hot start” körs: 1. 95°C 10 min ("ʺHot start"ʺ aktivering av Taq DNA pol.) 2. 95°C 1 min (denaturering) 3. 55.5°C 1 min (annealing) 4. 72°C 1 min (elongering) Steg 2 till 4 repeteras 36 gånger (cykler) 3 PCR mixen innehåller vid slutkoncentrationen (1x): -­‐‑ -­‐‑ -­‐‑ -­‐‑ -­‐‑ Taq DNA polymeras buffer: o 100mM NaCl o 10mM Tris-­‐‑HCl, pH: 7.5 1,5 mM MgCl2 0,2 mM dNTP 0,5 pmol/µμl uppströmsprimer 18-­‐‑mer (5´-­‐‑CAG AGT ACG GGG TTT CTC-­‐‑3´) 0,5 pmol/µμl nedströmsprimer 16-­‐‑mer (5´-­‐‑CCG ATT TCC TGC TGA G-­‐‑3´) 14 Dag 3: Verifiera PCR-produktens storlek på agarosgel Gjutning av agarosgel Resultatet av PCR-­‐‑reaktionen skall separeras på en 1,2% agarosgel. Gelen gjutes genom att agaros först smälts i TAE-­‐‑buffert i mikrovågsugn och sedan hälles i ett geltråg med gelkam. Etidiumbromid (EtBr) inkluderas i gelen för att visualisera DNAt. Etidiumbromid är ett mutagen så använd handskar vid detta steg och arbeta i dragskåp! Material: -­‐‑ Agaros -­‐‑ 200 ml Erlenmeyerkolv (E-­‐‑kolv) -­‐‑ 1x TAE-­‐‑buffert4 -­‐‑ Etidiumbromid, EtBr (10mg/ml) -­‐‑ Handskar Utförande: 1. Väg upp 0,72 g agaros i en 200 ml E-­‐‑kolv. 2. Tillsätt 60ml 1xTAE till E-­‐‑kolven med agaros och koka upp agarosen i mikrovågsugn. Se upp, den kokar över mycket lätt! Agarosen skall vara helt smält och inga korn/sliror får synas om lösningen hålls upp mot ljuset. 3. Preparera ett geltråg med en gelkam (5-­‐‑tandad, 9 mm breda kammar) och tejpa trågets kortsidor. 4. Låt agarosen svalna till ca 60°C (knappt att man kan hålla i den). Blanda om försiktigt med jämna mellanrum medan den svalnar så att den inte stelnar utmed kanterna. Blanda i 8µμl EtBr (i dragskåp). 5. Häll den avsvalnande agaroslösningen i tråget i dragskåp. Låt svalna till rumstemperatur. Se till att brunnarna inte blir ”sammankopplade” p.g.a. för mycket agaros, se figuren nedan. Undvik att det bildas bubblor. Gräns för agaros Max gräns för agaros Gelkam Geltråg 4 1x TAE innehåller: -­‐‑ -­‐‑ 40 mM Tris-­‐‑acetat 1mM EDTA 15 Agarosgelelektrofores av PCR produkt Gelen förs över till elektroforesapparaten och TAE-­‐‑buffert hälls på så att gelen täcks helt. Gelkammen dras ur försiktigt och proven laddas i de brunnar som bildas. En storleksmarkör (en blandning av DNA-­‐‑fragment med kända längder) pipetteras i en brunn på sidan för att kunna uppskatta storleken på PCR-­‐‑produkten. DNA är negativt laddat och kommer att vandra mot anoden (den positiva polen) när spänningen läggs på. DNAt visualiseras med hjälp EtBr och UV-­‐‑ljus på ett UV-­‐‑bord. Gelen fotograferas och storleken på PCR-­‐‑produkten bestäms. Tänk på att gelen innehåller EtBr och att EtBr kommer att föras över i TAE-­‐‑bufferten under tiden gelen körs. Använd handskar då ni hanterar gel, geltråg och buffert. Torka upp spill och släng allt kontaminerat av EtBr i riskavfallet. Material: -­‐‑ PCR-­‐‑reaktion från tidigare moment -­‐‑ Storleksmarkör (”Generuler”, laddas direkt på gel, ladda 4µμl på gelen) -­‐‑ Gelladdningsbuffert5 -­‐‑ 1x TAE-­‐‑buffert Utförande: 1. Ta bort tejpen från kanterna och ställ ner hela tråget med gelen i elektroforesapparaten och häll på 1x TAE-­‐‑buffert så att gelen täcks av buffert. Tänk på att sätta gelen åt rätt håll (− och + )! 2. Pipettera 7µμl gelladdningsbuffert direkt till PCR-­‐‑rören med prov och kontroll. Blanda och centrifugera kort eller slå ner proven i botten på röret. 3. Avlägsna försiktigt kammen på den stelnade agarosgelen så att brunnar bildas. 4. Så här skall brunnarna laddas (från vänster till höger): • Storleksmarkör ”Generuler” (4µμl) • 15µμl av provet • Lämnas tom • 15µμl kontroll • Lämnas tom 5. Ladda proverna på gelen med fin pipettspets. Undvik bubblor i pipettspetsen och att pipettera luft då bubblor som stiger ur brunnarna stör provet. Gelladdningsbufferten innehåller bl.a. glycerol som gör att proverna är tyngre än bufferten och sjunker ner i brunnarna. 6. Kör gelen vid 100V tills den blå markören har vandrat ca 3 cm (ca 60 min). 5 Gelladdningsbufferten innehåller: -­‐‑ 1% xylencyanol -­‐‑ 1% bromfenol-­‐‑blått -­‐‑ 40% glycerol 16 7. Lyft ur tråget med gelen och placera gelen på UV bordet. Slå på UV-­‐‑ljuset och dokumentera sedan gelen för storleksbestämning av PCR-­‐‑produkten. Detta moment gör ni tillsammans med labammanuens. Tänk på att bufferten i apparaten innehåller EtBr efter avslutat körning. Ha därför ett papper under så att det inte droppar buffert i labbet. Detta är storleksmarkören som används. 1µμg (10µμl) av GeneRuler DNA Ladder Mix innehåller: -­‐‑ 120ng av 500 bp fragmentet -­‐‑ 120ng av 1000bp fragmentet -­‐‑ 32ng av 2000bp fragmentet -­‐‑ 120 ng av 3000bp fragmentet. Rita av resultatet av er agarosgelelektrofores nedan: 17 Egenuppgift: Primerdesign Denna uppgift kan göras hemma eller i en datasal.. Primerdesign in silico (=i datorn) För att köra en PCR, vare sig det är en ”vanlig PCR” (Lab 7) eller en kvantitativ RT-­‐‑ PCR (Lab 10) måste man ha sk. primers, dvs. korta bitar esDNA (16-­‐‑25bp) som designats för att känna igen en specifik DNA-­‐‑sekvens. Primerns uppgift är att erbjuda DNA-­‐‑polymeraset en startpunkt (dvs. en fri 3’-­‐‑ände) från vilken nysyntes av DNA kan ske. Man ”ramar in” den sekvens man vill amplifiera med PCR genom att designa två stycken olika primers som passar ovanför och nedanför sekvensen man är intresserad av (sk. uppströms eller 5’-­‐‑primer och nedströms eller 3’-­‐‑primer, tillsammans kallas de för ett primerpar). Innan man kan designa sina primers behöver man ha information om den sekvens man vill amplifiera. Det finns flera olika databaser på internet som har sådan sekvensinformation för olika arter. I denna del av lab. 7 ska ni först hämta hem information om sekvensen för kycklingens beta-­‐‑ NGF/NGFB (beta-­‐‑nerve growth factor) gen från Ensamble-­‐‑databasen6 och sedan designa ett primerpar som kan amplifiera en bit av NGF genen med hjälp av Primer3plus7. Material: Ensembl: http://www.ensembl.org/index.html Primer3Plus: http://www.bioinformatics.nl/cgi-­‐‑bin/primer3plus/primer3plus.cgi Utförande: Ni ska söka efter NGF-­‐‑genen i kycklingens genom. Välj ”Chicken” i sökfältet, skriv in ”NGF” och klicka sedan på ”Go”. Ensembl är ett sökverktyg som innehåller bla. sekvensinformation från ett stort antal organismers genom sekvenser, bland annat kycklingens. 7 Primer3plus är ett online-verktyg för att designa primers. 6 18 Gallus gallus (=latinska namnet för kyckling). Ett antal sökresultat dyker upp. Klicka på ”chicken” och sedan ”gene”. Tryck på ”Gene ID”. En ny sida laddas upp med all möjlig info om NGF-genen. Titta igenom sidan och ta reda på nedan uppgifter. Bläddra mellan de olika flikarna längst upp på sidan. När det gäller sista frågan angående genens funktion får ni själva söka efter informationen. Det är viktigt att ni använder en pålitlig källa samt att ni anger vilken källa ni använde er av för att besvara frågan. På vilken av kycklingens kromosomer finns NGF? Ligger NGF på kromosomens forward eller reverse strand? Hur många exoner består NGF genen av? Hur många aminosyror består NGF-proteinet av? Vad för funktion har NGF genen? 19 Ni vill ha cDNA-­‐‑sekvensen för NGF. Klicka fliken ”Transcript NGF_CHICK” högst upp (bild 1) och därefter ”Exons” längst ut till vänster (bild 2). Ni får då fram sekvensen i en grå ruta. Markera sekvensen med musen och kopiera den (CTRL+C). Klistra sedan in den (CTRL+V) i ett nytt txt-­‐‑ eller word dokument för att ha den tillgänglig senare. Ni har nu hämtat sekvensen för kyckling NGF. Dags att designa primers. Följ länken till Primer3plus. Klistra in sekvensen för kyckling NGF i det stora vita fältet och välj sedan ”General settings” (bild 3). I General settings väljer ni följande parametrar: Product size ranges: 400-­‐‑410 Önskad storlek på vår PCR produkt (ca 400bp är bra). Primer size: Min: 18, Opt: 20, Max: 25 Föredragen storlek på våra primers (mellan 18-­‐‑22bp) Det finns även en del andra parametrar man kan ställa in, bl.a. smältpunkt för primrarna (Tm) och önskat GC innehåll osv. Men de lämnar vi åt sidan så länge. Tryck sedan på den gröna knappen ”Pick primers” (bild 4). 20 Ni får nu upp ett nytt fönster med olika förslag på primerpar. Skriv ner datorns första förslag på primrar här nedan: Primer 1/left: Primer 2/right: Produktens längd: Kolla igenom resten av listan notera följande till labrapporten: • Hur många olika förslag på primerpar fick ni upp? • Jämför förslag nr1 på primerpar med sekvensen ni klistrade in (blå och gul markerat). En av primrarna är inte identisk med sekvensen ni klistrade in. Varför? • Vilken primer är 5’ resp. 3’ primer? I nästa steg är det viktigt att kontrollera att primrarna är specifika och verkligen bara känner igen den region/gen man vill amplifiera med PCR. Detta kontrolleras genom att söka med primersekvenserna mot kyckling-­‐‑genomet exempelvis NCBI-­‐‑Entrez-­‐‑ databasen för att se om det eventuellt finns andra regioner i kyckling-­‐‑genomet som primrarna kan binda till. Om primrarna känner igen fler platser än en, är de inte specifika nog och man måste välja/designa nya primers. Denna typ av sökning eller sekvensjämföresle kallas för BLAST (Basic Local Alignment Search Tool) och ni ska öva mer på BLAST-­‐‑sökningar i lab 9. Gå till NCBI-­‐‑Entrez (http://www.ncbi.nlm.nih.gov/sites/gquery). Välj funktionen BLAST i övre högra hörnet (bild 5). En ny sida kommer upp (bild 6). Välj programmet Nucleotide blast (pil) och ytterligare ett fönster (bild 7) visas i vilket ni skall skriva/klistra in era primer sekvenser. 21 Skriv in sekvensen på Primer 1 och 2 på två rader i den översta rutan. Välj sedan Database: Others/Nucleotide collection (nr/nt) Organism: ”Gallus gallus” (bild 7) Starta Blast med BLAST knappen! 22 Ett nytt fönster öppnas medan databasen arbetar. Det kommer förmodligen att dröja ett tag (ca 1 minut) innan sökningen är färdig eftersom databasen söker med primersekvenserna mot hela kycklingens genom för att försöka hitta träffar. Det är färdigt när ett nytt fönster dyker upp. Ni får nu se en ny sida som visar vilka era sekvenser i databasen som era primers känner igen. Nedanför kommer även en lista att visas med eventuella träffar. Varje träff ges en ”score” och ju högre desto bättre. ”Query coverage” säger hur många procent av söksekvensen som matchar i databasen. Under listan visar resultatet av jämförelsena. Besvara följande till labrapporten: • Vad känner era primers igen? • Vad kan ni säga om dessa primers specificitet utifrån sökresultatet? Binder de på flera ställen med hög % av ”Query coverage”? • Skulle ni vilja använda dessa för att köra PCR? Primer-­‐‑design delen av lab 7 klar! Observera dock att det primerpar som ni just har letat fram inte är samma primerpar som används i PCR-­‐‑labben. För varje bit DNA man vill amplifiera med PCR finns det oftast flera olika primers man kan använda! 23 Mall för labrapport med specifika frågor till laboration 7: Lysering och spektrofotometri: 1. Vilken funktion har Proteinas K och SDS i lyseringsbufferten? 2. Vad är SDS för typ av reagens? 3. Visa era DNA-­‐‑koncentrationsberäkningar. 4. Hur mycket DNA (µμg) fick ni totalt ut från kycklinglevern? 5. Vad blev kvoten A260/A280?Vad visar denna kvot? Inom vilka värden bör den ligga? Redovisa även mätvärdena ni fick. PCR och gelelektrofores: 1. Beskriv funktionen hos de komponenter som ingår i en PCR-­‐‑reaktion. 2. Vad menas med "ʺhot start"ʺ och varför använder man denna metod vid PCR? 3. Varför utförs en negativ kontroll till en PCR-­‐‑reaktion? 4. Varför kör man ut PCR-­‐‑reaktionen på agarosgel? 5. Vilken funktion har laddningsbufferten och Generuler? 6. Bilägg gelbild från agaroselektroforesen av PCR-­‐‑produkten (märk ut tydligt på gelen vad som är vad och relevanta storlekar i stegen). 7. Vilka fragment kan ni se? Notera storlekar på banden i storleksmarkören samt för PCR-­‐‑produkten. 8. Syns några band i den negativa kontrollen? Varför/ varför inte? Förklara. 9. Uppskatta mängden DNA som bildats i PCR-­‐‑reaktionen (uttryckt i µμg) genom att jämföra PCR-­‐‑bandets intensitet med intensiteten i ett av banden i storleksmarkören (som har känd DNAmängd/µμl, se figur ovan). Räkna sedan baklänges för att få reda på hur mycket DNA som måste finnas i PCR-­‐‑röret. 10. Varför syns DNAt i UV-­‐‑ljus? 11. Om du råkar göra en 2%-­‐‑ig agarosgel i stället för 1%-­‐‑ig, hur påverkar detta din elektrofores? Primer design: 1. Besvara frågorna ang. NGF sökträffarna. 2. Hur såg datorns första primer-­‐‑förslag ut? Hur lång produkt blir det? 3. Besvara frågorna ang. primersökningen. 4. Besvara frågorna ang. BLAST-­‐‑sökningen. 24 8: Kloning av DNA Dag 1: Dag 2: Dag 3: • • • • • • • • • Ligera PCR-produkt och vektor Transformera bakterier Sprida bakterier på plattor Räkna blå/vita kolonier Plocka kolonier till övernattskultur Preparera plasmid-DNA Restriktionsenzymsklyvning av plasmid-DNA Gjuta agarosgel Köra ut klyvda och oklyvda plasmider på gel Teori Kloning är en av de absolut vanligaste metoderna på lab och olika varianter av kloningsarbete används rutinmässigt överallt för att isolera och arbeta med DNA och gener. Varianter på tillvägagångssättet används i all forskning där man har behov av ett specifikt DNA-­‐‑fragment eller sekvens, men att klona olika typer av fragment i plasmid-­‐‑vektorer (som i denna lab) är antagligen det enskilt vanligaste molekylärbiologiska experimentet. Några andra exempel där kloning används: isolering av nya gener, screening av bibliotek, sekvensning, riktad mutagenes, in vitro transkription, mm. All kloning bygger dock på att med hjälp av enzymer klippa och klistra i DNA för att bilda det konstrukt man vill ha, och sedan utnyttja exempelvis bakterier eller virus för att bära och mångfaldiga konstruktet. Särskilda instruktioner Momenten utförs i grupper om 2-­‐‑3 personer. Etiduimbromid (EtBr) är mutagen och måste hanteras i dragskåp och med handskar. X-­‐‑Gal är giftigt och måste hanteras med handskar. Handskar måste även användas vid ligeringen och restriktionsklyvning för att inte kontaminera reagensen. Enzymerna (T4 DNA ligas & restriktionsenzymer) får inte bli rumstempererade och måste hela tiden förvaras i frysblocket. Laborationsrapport Rapporten lämnas in gruppvis. Besvara de specifika frågorna som står i slutet. Om ni fick avvikande och/eller felaktiga resultat ska ni göra en felanalys där ni anger troliga förklaringar till varför ni fick dessa. 25 Bakgrund I denna lab ska ni först ligera samman ett DNA-­‐‑fragment (samma som ni ampliferade med PCR) med en vektor som heter pMOS-­‐‑Blue och sedan transformera in detta konstrukt i bakterier. pMOS-­‐‑Blue är en vektor som möjliggör sk. blå-­‐‑vit selektion och efter transformationen kommer ni att plocka både blåa och vita kloner som ni odlar vidare. Sedan preparerar ni fram plasmid-­‐‑konstruktet ur dessa och klyver upp med restriktionsenzym och kör ut på agarosgel. På så sätt får ni bekräftat om ert kloningsexperiment lyckades och någon av bakterierna innehåller det konstrukt ni ville skapa. Det är enklast att ligera två DNA-­‐‑molekyler som har komplementära och överlappande ändar (sk. ”sticky-­‐‑ends”) men det går också att ligera "ʺblunt ends"ʺ eller sk. trubbiga ändar. För att en sticky-­‐‑end ligering skall lyckas är det absolut nödvändigt att ändarna är komplementära (dvs. kan baspara) till varandra. Därför måste man ha samma sorts ändar på fragmentet som på vektorn. Om man inte har det måste man först skapa sk. blunt-­‐‑ends för att man ska kunna ligera samman fragment och vektor. I denna lab använder vi blunt-­‐‑end kloning. pMOS Blue-­‐‑vektorn som ni använder i denna lab för ligeringen har redan klyvts med restriktionsenzymet EcoRV som ger trubbiga ändar. Den EcoRV kluvna plasmiden har dessutom behandlats med enzymet fosfatas som tar bort 5'ʹ-­‐‑fosfatgrupperna. Fosfatasbehandlingen görs för att pMOS inte skall kunna ligeras ihop med sig själv (ringslutas). PCR-­‐‑produkten som skall ligeras in har även den i förväg behandlats för att få trubbiga ändar, men dessa ändar har inte fosfatasbehandlats utan har sina 5’-­‐‑ fosfatgrupper kvar. Efter ligeringen förs plasmid-­‐‑DNAt in i bakterier. Detta kan ske genom transformation eller elektroporering. Vid transformation behövs sk. kompetenta bakterier som kan ta upp plasmid-­‐‑DNA. Kompetenta bakterier kan köpas eller framställas genom att behandlas med bl.a. CaCl2. Normalt tar bara ett fåtal av bakterierna upp DNA. Endast de bakterier som tagit upp plasmid och därmed en antibiotika-­‐‑resistensgen kommer att vara resistenta mot antibiotika (vanligtvis ampicillin) och kommer att överleva selektionen när de sprids ut på amp-­‐‑plattor. En plasmidvektor som har ett DNA-­‐‑fragment (ett sk. insert) kallas en rekombinant plasmid. Vid ligeringen bildas normalt både rekombinanta och icke-­‐‑rekombinanta plasmider. De selekterade bakteriekolonierna screenas ytterligare för om den upptagna plasmiden är rekombinant eller ej. Denna andra typ av screening kallas blå/vit-­‐‑screening eftersom den görs utifrån bakteriekoloniernas färg. pMOS har ett sk. "ʺmultiple cloning site"ʺ (MCS) mitt i en gen, LacZ, som kodar för enzymet ß-­‐‑galaktosidas. ß-­‐‑galaktosidas kan spjälka substratet X-­‐‑gal (se figurer nedan) och ger då en blå produkt. Om ett DNA fragment ligeras in i MCS så förstörs enzymets aktivitet. Bakterier som bär på en icke-­‐‑rekombinant plasmid (endast vektor) kommer alltså att innehålla ett aktivt enzym och kommer bilda blåa kolonier på X-­‐‑gal plattor. Bakterier som tagit upp rekombinant plasmid kommer vara färglösa 26 (vita). Genom att undvika att plocka blåa kolonier kan man öka chansen att få en rekombinant plasmid. Överkurs: För att LacZ genen skall vara aktiv behövs vanligtvis isopropyl-­‐‑ß-­‐‑ thiogalaktosid (IPTG). IPTG är en galaktosanalog och aktiverar ß-­‐‑galaktosidasgenen. Vi kommer dock att använda en stam av E.Coli-­‐‑bakterier som kallas TOP10. Denna stam kräver ej IPTG för att aktivera LacZ och ni behöver därför inte tillsätta det. 27 Dag 1: Ligering och transformation Ligering av PCR-produkt och pMOS-Blue vektor Ni ska sätta upp två ligeringsreaktioner. En som innehåller PCR-­‐‑produkten (kloningsligering) och en utan PCR-­‐‑produkt (kontroll-­‐‑ligering). Material: -­‐‑ Renad PCR-­‐‑produkt (med trubbiga ändar och 5’-­‐‑fosfat) -­‐‑ pMOS-­‐‑Blue vektor (klyvd med EcoRV (trubbiga ändar), fosfatas-­‐‑behandlad) -­‐‑ Eppendorfrör -­‐‑ T4 DNA ligas -­‐‑ MQ H2O -­‐‑ 10x ligasbuffert8 Utförande: Märk 2 Eppendorfrör med gruppnummer samt ”Ligering” och ”Kontroll-­‐‑ligering” och pipettera upp följande: Ligering: Kontroll-­‐‑ligering: 6 µμl MQ vatten 7 µμl MQ vatten 1 µμl PCR produkt Ingenting 1 µμl vektor 1 µμl vektor 1 µμl 10x ligasbuffert 1 µμl 10x ligasbuffert 1 µμl T4 DNA ligas 4 U (4 U/µμl) 1 µμl T4 DNA ligas 4 U (4 U/µμl) tot volym: 10 µμl tot volym: 10 µμl Inkubera rören vid rumstemperatur i 15 minuter. 8 10x Ligasbuffert innehåller: -­‐‑ 100 mM Tris-­‐‑HCl pH7,5 -­‐‑ 50mM NaCl -­‐‑ 10mM ATP 28 Transformation av kompetenta bakterier Ligeringarna blandas med de kompetenta bakterierna och inkuberas i isbad. Förutom ligeringarna så transformeras en kontroll-­‐‑plasmidDNA (TK). Denna transformation fungerar som positiv kontroll för transformationen och talar om hur bra transformationen fungerat. Bakterierna utsätts sedan för en värmechock och sprids på plattor med ampicillin. Bara bakterier som tagit upp plasmid och är resistenta mot ampicillin kommer att överleva på plattorna (selektion). Plattorna ska även förses med X-­‐‑gal som används som substrat för blå/vit-­‐‑screeningen. Material: -­‐‑ 6 st TYE-­‐‑agarplattor med ampicillin 100µμg/ml (röd petriskål) från lab 6 -­‐‑ Glaskulor för bakteriespridning -­‐‑ X-­‐‑gal (5-­‐‑bromo-­‐‑4-­‐‑chloro-­‐‑3-­‐‑indolyl-­‐‑ß-­‐‑galactoside) 20mg/ml -­‐‑ De två rören med ligeringar -­‐‑ Kontroll-­‐‑plasmidDNA, 0,01ng/µμl (för positiv transformationskontroll=TK) -­‐‑ Kompetenta bakterier: E.coli TOP10 (3 frusna rör, 20µμl/rör) -­‐‑ 1 rör med sterilt TYE-­‐‑medium -­‐‑ Isbad, värmeblock 42°C och vattenbad 37°C. Utförande: 1. Fyll en islåda med is. Hämta sedan 3 rör med 20µμl kompetenta bakterier av stammen TOP10 från kursledare. OBS! De kompetenta bakterierna är mycket känsliga och måste hållas på is hela tiden! 2. Pipettera direkt till var sitt rör med bakterier (endast en ”ligering” per rör): -­‐‑ 4µμl ligering (markera röret med +) -­‐‑ 4µμl kontroll-­‐‑ligering (markera röret med −) -­‐‑ 2µμl kontroll-­‐‑plasmidDNA (markera röret med TK) Blanda INTE med pipettering upp och ner utan försiktigt genom att knäppa med fingret mot röret. Undvik att värma upp proven genom att hålla långt ner på rören, håll i stället uppe vid locket! 3. Inkubera rören på is 15 minuter. Se till att värmeblocket håller 42°C. Under tiden: Märk upp era amp-­‐‑plattor. Märk era plattor med gruppnummer samt: +, − eller TK (två plattor till varje). Märk sedan +, − eller TK-­‐‑plattorna med ”50µμl” resp. ”150µμl” (totalt sex plattor). 4. Efter det att bakterierna stått på is 15 minuter, är det dags att värmechocka bakterierna för att transformera dem. Inkubera rören med bakterier på värmeplattan (42°C) i EXAKT 30 sekunder. Sätt genast rören på is igen och inkubera i ytterligare 2 minuter. 5. Tillsätt 250µμl TYE-­‐‑medium till rören med transformerade bakterier och inkubera rören vid 37°C i 40 minuter under skakning. 6. Preparera amp-­‐‑plattor med X-­‐‑gal. Häll ut ett tiotal glaskulor på plattorna märkta med + eller – (totalt fyra plattor) och pipettera 40µμl X-­‐‑gal till varje platta och sprid. Fördela lösningen direkt på plattan genom att skaka runt kulorna. 29 Var noga så att X-­‐‑gal kommer på hela plattan (brist på X-­‐‑gal kan ge falska resultat vid er blå/vit-­‐‑screening). Behåll glaskulorna i plattan i väntan på spridning av bakterierna. X-­‐‑Gal är giftigt och måste hanteras med handskar. Spridning av transformerade bakterier Utförande: 1. 50µμl av transformations-­‐‑lösningarna skall sättas på plattor märkta ”50µμl” och 150µμl skall sättas till plattor märkta ”150µμl”. Fördela bakterierna med hjälp av glaskulorna. 2. Låt agarn suga upp lösningen några minuter innan glaskulorna hälles av i angivet uppsamlingskärl. 3. Inkubera plattorna upp och ner vid 37°C över natt. 30 Dag 2: Beräkna transformationseffektivitet och plocka kloner Beräkna transformationseffektiviteten De bakterier som växer på plattorna måste ha tagit upp plasmid eftersom de uppvisar ampicillin-­‐‑resistens. Olika bakterier har dock tagit upp olika plasmider. Bakterier som tagit upp rekombinanta plasmider blir färglösa (vita) och de som tagit upp icke-­‐‑rekombinanta plasmider (endast vektor) blir blå (se bild). Räkna och notera hur många kolonier ni fått på plattorna samt andelen blå och vita kolonier. Alla gruppers resultat noteras på tavlan och resultaten diskuteras gemensamt i labbet tillsammans med handledare. Plocka blå och vita kloner Två vita och en blå koloni plockas till rör med TYE-­‐‑medium och odlas upp i 37°C till nästa dag. Dessa kulturer kommer sedan att används för minipreparation av plasmidDNA. Material: -­‐‑ Agarplattor med kolonier från igår -­‐‑ Pipettspetsar -­‐‑ 3 st skruvlocksrör med 2ml TYE-­‐‑medium (Innehåller Amp) Utförande: 1. Märk tre skruvlocksrör: ”VIT A”, ”VIT B” (vita kolonier) och ”BLÅ” (blå koloni) samt gruppnummer. Två vita kolonier (med förhoppningsvis ligerad PCR-­‐‑produkt, tas från +-­‐‑plattan) och en blå koloni (förmodligen utan PCR-­‐‑produkt, tas från −-­‐‑plattan) skall plockas. 2. Ta plattan med den spädning som gett enskilda, lätt urskiljbara, kolonier och välj ut två vita och en blå koloni. Plocka kolonierna till var sitt skruvlocksrör med hjälp av en pipettspets. Spetsen släpps ner i röret och lämnas där. 3. Skruva fast locken på rören. Rören inkuberas i 37°C med skak över natt (tas om hand av labhandledare). 31 Dag 3: Plasmidpreparation, restriktionsenzymsklyvning, gel Plasmidpreparation (TENS-metoden/”mini-prep”) Bakterierna som odlats i skruvlocksrör förs över till Eppendorfrör, centrifugeras ner och slammas upp i en liten volym för att sedan brytas ner med TENS-­‐‑buffert. Lösningen neutraliseras med NaAc varvid bakteriecellvägg samt genomiskt DNA faller ut och centrifugeras ner. PlasmidDNAt och RNA finns i supernatanten och precipiteras (fälls ut) med etanol. Plasmid DNAt löses sedan upp i vatten och analyseras med agarosgelelektrofores. Proven behandlas med RNase för att selektivt bryta ner bakteriellt RNA. Material: -­‐‑ 6 st Eppendorfrör -­‐‑ TENS-­‐‑buffert9, pH >12 -­‐‑ 3M NaAc, pH 5,2 -­‐‑ RNase A lösning -­‐‑ Etanol, kall, 99,5% samt 70% -­‐‑ MQ H20 Utförande: 1. Kontrollera att de plockade klonerna har vuxit i rörkulturerna. TYE-­‐‑mediet skall vara grumligt som ”uppslammat mjöl”. Från varje rör skall 1500µμl bakterielösning pipetteras över i Eppendorfrör och centrifugeras med högsta hastighet (10K) under 10 sekunder. Sätt i rören med gångjärnet utåt för att få bakteriepelleten under gångjärnet. 2. Efter centrifugering skall det bli en klart synlig bakteriepellet. Häll av den klara supernatanten försiktigt utan att få med pelleten (ca 100µμl medium blir kvar i röret om man försiktigt häller av lösningen utan att knacka ut det sista). Vortexa kraftigt tills pelleten är helt uppslammad i de resterande 100µμl medium. 3. Tillsätt 1µμl RNase A lösning till varje rör. Inkubera 5 min i rumstemperatur. 4. Tillsätt 300µμl TENS buffert per rör och vänd försiktigt rören upp och ner 5-­‐‑7 gånger för att blanda (vortexa inte). Lösningen skall bli mer eller mindre klar och slemmigt tjockflytande (viskös) av denaturerat genomiskt DNA och cellvägg (kan vara svårt att se). 5. Tillsätt 150µμl 3M NaAc lösning till rören och vänd rören upp och ner 5 gånger för att blanda och skaka sedan om rören kraftigt med handen (vortexa inte). 6. Centrifugera rören 10 minuter, 10K. Gångjärnet utåt! 7. Ta rören direkt efter det att centrifugen stannat och för över supernatanterna till nya märkta Eppendorfrör utan att få med något av pelleten. 9 TENS innehåller: -­‐‑ 10mM Tris-­‐‑HCL pH: 7,5 -­‐‑ 1mM EDTA -­‐‑ 0,1M NaOH -­‐‑ 0,5% SDS 32 8. 9. 10. 11. 12. 13. 14. Volymen av den överförda supernatanten bör vara ca 450µμl. PlasmidDNAt finns löst i supernatanten. Fäll ut (=precipitera) plasmidDNAt genom att tillsätta 900µμl kall 99,5% etanol till rören och blanda sedan om genom att vända rören under några sekunder. Inkubera rören 2-­‐‑5 minuter i rumstemperatur på bänken. Centrifugera sedan rören 10 minuter 10K i mikrofug. Gångjärnet utåt! Det skall bli en synlig liten vit pellet (pelleten är inte alltid synlig) som består av plasmidDNA samt eventuellt RNA. Supernatanten suges försiktigt av utan att vidröra pelleten. Tillsätt 1ml 70% etanol för att tvätta pelletten och vortexa lätt. Centrifugera 30 sek. 10K (med samma orientering på röret). Sug försiktigt bort lösningen utan att få med pelletten. Sug av så mycket som möjligt av etanolen och ställ röret upp och ned på en pappershandduk under några minuter. Knacka försiktigt ut den sista etanolen på pappret och kontrollera att pelleten sitter kvar. Det går även att torka ut den sista etanolen genom att ställa röret med öppet lock i värmeblocket en kort stund. Tillsätt 50µμl MQ H20 till rören och vortexa tills pelleten gått i lösning. Märk rören och tejpa på märkningen med genomskinlig tejp så texten inte försvinner. Gjuta agarosgel Gör gelen på samma sätt som i lab 7, men använd 0,60 g agaros (1,0 % agarosgel). Använd en kam med 8 tänder istället för 5. Material: -­‐‑ Agaros -­‐‑ 200ml Erlenmeyerkolv (E-­‐‑kolv) -­‐‑ 1x TAE-­‐‑buffert -­‐‑ Etidiumbromid (10mg/ml) Medans gelen stelnar utförs restriktionsklyvningen. 33 Restriktionsenzymklyvning av plasmidDNA Restriktionsenzymsklyvning i kombination med storleksanalys av DNA-­‐‑fragment används för att enkelt analysera identiteten på DNA och för att klyva ut DNA-­‐‑ fragment från större DNA-­‐‑bitar eller för att kontrollera resultatet efter en ligering. För att analysera storleken av ett DNA-­‐‑insert i en rekombinant plasmid används restriktionsenzym som klyver ut fragmentet. Klyvningen analyseras sedan på agarosgel. Man kan förutom att studera fragmentets storlek även kontrollera i vilken riktning det har satt sig inuti plasmiden genom att använda ett restriktionsenzym som man vet klipper osymmetriskt inuti fragmentet. I detta moment ska ni dock bara klyva upp plasmiden för att bekräfta och kontrollera att ett fragment av rätt storlek hamnat i MCS. Att klyva ut fragmentet: Studera pMOS-­‐‑Blue multiple cloning site igen. Som ni kommer ihåg ligerades fragmentet in med hjälp av trubbiga ändar i EcoRV sitet. Denna process förstörde därmed EcoRV igenkänningssekvensen och EcoRV kan inte användas för att på nytt klyva ut fragmentet. För att göra det måste vi använda andra restriktionsenzym som ligger på varsin sida om fragmentet. Vi väljer att använda EcoRI och HindIII. Överkurs: Eftersom vi ligerade med trubbiga ändar kommer fragmentet att slumpmässigt hamna i antingen rätt eller fel riktning. För att kontrollera i vilken riktning fragmentet har hamnat kan man antingen sekvensera sin plasmid eller använda restriktionsenzym som: a) klipper endast en gång i vektorn, b) klipper endast en gång i fragmentet, samt c) klyver osymmetriskt inuti fragmentet så att två olika långa ”fragmentbitar” bildas. I denna laboration kommer vi dock inte att utföra en ”riktnings-­‐‑klyvning”. Material: -­‐‑ Eppendorfrör -­‐‑ Fällt DNA från prov VIT A och VIT B -­‐‑ Fällt DNA från BLÅ -­‐‑ MQ H20 -­‐‑ Restriktionsenzym EcoRI (FastDigest enzym) -­‐‑ Restriktionsenzym HindIII (FastDigest enzym) -­‐‑ Restriktionsenzymbuffert, 10x FastDigest buffer10 (Slutkoncentration: 1x) 34 Utförande: Plasmiderna (VIT A, VIT B och BLÅ) klyvs med restriktionsenzymen EcoRI och HindIII och analyseras på agarosgel. Storleken på de utkluvna fragmenten i den rekombinanta plasmiden och storleken på vektorn uppskattas genom jämförelse med storleksmarkör. Även prov med oklyvda plasmider skall analyseras på gelen. Sex Eppendorfrör märks, tejpas och klyvningarna sätts upp som nedan. OBS! endast ett prov plasmidDNA/ rör (dvs. VIT A, VIT B eller BLÅ). Fragment-­‐‑klyvning (3st): 7µμl MQ H20 2µμl 10x FastDigest buffer 9µμl plasmidDNA (VIT A/B, BLÅ) 1µμl enzym EcoRI 1µμl enzym HindIII 20µμl total volym Oklyvd kontroll (3st): 9µμl MQ H2O 2µμl 10x FastDigest buffer 9µμl plasmidDNA (VIT A/B/ BLÅ) 0µμl enzym 0µμl enzym 20µμl total volym Rören blandas försiktigt genom att slå med fingret på rörets spets och lösningen samlas upp i rörets botten genom att kort centrifugera rören. Klyvningarna och kontrollerna inkuberas i 5 minuter i 37°C (ställ rören i värmeskåpet där ni hade era bakterieplattor). 35 Agarosgelelektrofores av plasmidDNA De olika klyvningarna samt storleksmarkören laddas på gelen. Material: -­‐‑ Rör med klyvningarna från ovan -­‐‑ Gelladdningsbuffert -­‐‑ Storleksmarkör (Stege; Generuler) -­‐‑ Handskar Utförande: 1. Pipettera 3µμl gelladdningsbuffert direkt till varje rör med klyvningar och kontroller. Blanda och centrifugera samtliga prov kort för att spinna ner proven i botten på röret. 2. Avlägsna försiktigt kammen på den stelnade agarosgelen så att brunnar bildas. 3. Ladda 20µμl av proven på gelen med pipettspets. Från vänster till höger: • Storleksmarkör (4µμl) • BLÅ [Oklyvd] • VIT A [Oklyvd] • VIT B [Oklyvd] • Lämnas tom • BLÅ [EcoRI+HindIII] • VIT A [EcoRI+HindIII] • VIT B [EcoRI+HindIII] 4. Kör gelen på 100V tills den första blå markören har vandrat ca 3cm (ca 30 min). 5. Lyft ur tråget med gelen och placera gelen på UV-­‐‑bordet. Slå på UV-­‐‑ljuset och beskåda och fotografera gelen. Kör eventuellt gelen ytterligare för att få fullständig upplösning av banden och dokumentera sedan gelen. Identifiera proverna och storleksbestäm de olika banden med hjälp av storleksmarkören. Jämför de kluvna proverna med respektive okluvna. Innehåller plasmiderna VIT A eller VIT B PCR-­‐‑produkten? I så fall: Grattis! Ni har lyckats med att "ʺklona"ʺ ett DNA-­‐‑fragment. Om detta skulle varit ett "ʺskarpt"ʺ experiment så skulle man sparat rören med levande bakterier för att starta upp en större plasmidDNA preparation för att få mer DNA som sedan skulle kunna användas för de fortsatta forskningsändamål man har. 36 Mall för labrapport med specifika frågor till laboration 8: Ligering av PCR-­‐‑produkt och vektor: 1. Vad menas med ”blunt-­‐‑” och ”sticky ends”? Vilken typ använde ni? 2. Vad är fördelen, respektive nackdelen, med att använda blunt eller sticky ends vid kloning? 3. I lab 7 använde ni dATP och i denna lab ATP. Vad är skillnaden? I vilka syften använder man de olika nukleotiderna? Transformation: 1. Varför inkuberar man de transformerade bakterierna i TYE-­‐‑medium 30 minuter vid 37°C innan spridning på ampicillinplattor? 2. Vilka är de olika kontrollerna ni utför i detta moment och vad syftar de till? 3. Visa resultatet av transformationen i tabellform (antalet kolonier per platta per transformation, blå/vita kolonier, etc). 4. Varför blir kolonierna vita eller blå? 5. Transformationseffektiviteten uttrycks som ’antal bakterier per µμg DNA’ och beskriver hur bra bakterierna är på att ta upp plasmid. Beräkna transformationseffektiviteten med utgångspunkt från hur många kolonier som växte på transformationskontroll-­‐‑plattorna (den positiva kontrollen). 6. Varför blir frekvensen av återligering (ringslutning) av vektorn låg? 7. Hur kan det uppstå falska positiva kolonier? Analys av transformerade bakterier: 1. Efter att valda kloner odlats i flytande medium över natt preparerar ni fram plasmidDNA. I denna process, vilka funktioner har TENS och RNase? 2. Vilka restriktionsenzymer användes för analys av plasmidpreparationen? Varför utförs en kontroll utan restriktionsenzym? 3. Baserat på de kloner ni plockade för analys, vilka band förväntar ni er att se? 4. Bilägg gelbild och identifiera banden i storleksmarkören och proven (dvs. märk upp utskriften eller en kopia med svaren på fråga 5-­‐‑6 nedan). 5. Vilka band såg ni? Identifiera och beräkna storleken på de olika banden på gelen. Stämmer storleken på fragment respektive vektor med vad ni förväntat er? Vilka andra eventuella band ser ni? 6. I vilka former förekommer banden ni ser på gelen? Vilka band är ”supercoiled”, relaxerad cirkulär plasmid och/eller linjäriserad plasmid. Kan ni se RNA och bakteriellt genomiskt DNA i på gelen och i så fall var? 7. Uppskatta mängden plasmidDNA i klyvningen av prov BLÅ (uppskatta grovt) genom att jämföra bandets intensitet med intensiteten av banden i storleksmarkören (som har känd DNAmängd/µμl, se figur). 8. Beräkna hur mycket plasmidDNA (µμg) från prov VIT A som ni fick från i er minipreparation (dvs. TENS-­‐‑steget). Börja med att uppskatta intensiteten av banden liksom ovan. Beakta sedan eventuella fragments storlek i förhållande till hela plasmiden, hur mycket av provet ni laddade på gelen samt den totala volymen som ni spädde upp ert plasmidDNA prov i. 37 9: Sekvensering och bioinformatik Dag 1: • • Analys av sekvenseringsdata Sekvensjämförelse med hjälp av BLAST alignment Teori Majoriteten av DNA-­‐‑sekvensering sker med Sanger-­‐‑metoden. Metoden (också kallad dideoxymetoden eller ”chain termination method”) bygger på DNA-­‐‑polymerisering från en specifik primer och att polymeriseringen avbryts vid en av de fyra baserna. Detta sker genom att en låg andel av dideoxy-­‐‑nukleotider (ddNTP) blandas in i polymeriseringsreaktionen vilken stoppas då en dideoxynukleotid inkorporeras. Stoppet sker eftersom 3’–OH saknas på dideoxy-­‐‑nukleotiderna och då kan fortsatt DNA-­‐‑syntes inte fortgå. Om en reaktion innehåller ddATP, dGTP, dATP (lägre konc.), dTTP, dCTP, så kommer alla fragment som bildas ha ett A i slutet. Om fyra reaktioner körs med respektive ddGTP, ddTTP och ddCTP kommer alla baser i ett DNA-­‐‑fragment att representeras (se närmare Lodish et al). För att detektera DNA-­‐‑ fragmenten används fluorescens-­‐‑märkta nukleotider/primers. Storleken på DNA-­‐‑ fragmenten bestäms sedan genom någon form av elektrofores. All sekvensering är idag automatiserad och sköts ofta av lab-­‐‑robotar. Sekvenseringen använder sig av laserbaserad fluorescens-­‐‑detektion och i stället för en reguljär gelelektrofores används kapillärelektrofores. Sekvenseringsteknologin utvecklas snabbt och andra tekniker som kan sekvensera ett stort antal DNA fragment samtidigt har börjat användas mer och mer: 454 och Illumina är de viktigaste och bygger inte Chain termination/Sanger utan på Pyro-­‐‑sekvensering. Denna laboration är teoretisk och ni kommer att analysera resultat från en automatiserad Sanger-­‐‑sekvensering. Resultatet är från sekvensering baserad på fluorescens-­‐‑ och kapillärelektrofores av s k ”big dye termination type”. Vid denna typ av reaktioner är de olika dideoxynukleotiderna inmärkta med olika fluorokromer. Detta ger den stora fördelen att alla fyra baserna kan analyseras i en och samma reaktion. Sekvensreaktionen sker direkt på denaturerat dubbelsträngat plasmid-­‐‑DNA med hjälp av Taq DNA-­‐‑polymeras och en PCR-­‐‑maskin. DNA-­‐‑ fragmentet som skall sekvenseras kan vara klonat i en plasmid med sekvenseringsprimer-­‐‑sekvenser på vardera sidan av kloningsstället (multipel cloning site, MCS). Exempel på sekvensprimers är T3, T7, M13 eller M13rev. Särskilda instruktioner Labben utförs i respektive laborationsgrupp. Laborationsrapport Rapporten lämnas in gruppvis. Besvara de specifika frågorna som står i slutet. 38 Analys av sekvensinformation Bakgrunden till denna lab är att ni, precis som i lab 8, klonat in en gen från kyckling som heter Morf4l1 i pMOS-­‐‑Blue vektorn. Men istället för att använda PCR för att fiska upp Morf4l1-­‐‑genen ur genomiskt DNA (som med NGF), preparerade ni fram mRNA från kyckling som ni sedan översatte till cDNA med hjälp av reverse transcriptase. Sedan ampliferades Morf4l1 cDNAt med hjälp av PCR och klonades in i vektorn. Genom denna process klonade ni cDNA-­‐‑sekvensen för Morf4l1 (=sekvensen på det splicade mRNAt = bara exonerna) istället för den genomiska sekvensen som innehåller både exoner och introner. pMOS-­‐‑Blue vektorn innehåller ett T7-­‐‑site i början av MCS (se kartan i lab 8) som man kan använda för att sekvensera det som sitter i MCS. När ni börjar denna lab har ni precis fått er sekvensdata som erhållits genom sekvensering med T7 och kapillärelektrofores. Nu vill ni jämföra sekvensen på det Morf4l1 ni har klonat med den ”officiella sekvensen” för Morf4l1 som finns i databaserna. Syftet är att dels bekräfta att ni klonat fram rätt gen (dvs. Morf4l1) samt att kontrollera kvalitén på den Morf4l1-­‐‑sekvens ni har klonat jämfört med den officiella sekvensen. Material: -­‐‑ Filen: ”Morf4l1 sequenced with T7” -­‐‑ Programet “Chromas Lite” -­‐‑ Websidan “Entrez”: http://www.ncbi.nlm.nih.gov/sites/gquery Utförande: Hämta hem Morf4l1-­‐‑sekvensen från studentportalen (ligger i mappen ”Laborationer och seminarier” under filarean tillhörandes CMB kursen). Spara den på datorn. Öppna Chromas och öppna sedan filen “morf4l1seq”. Titta på sekvenseringsdiagrammet och notera var det finns regioner av bra respektive dålig sekvensinformation. Bra sekvensinformation är när topparna är höga, väl skilda åt och inte överlappar varandra för mycket. Sekvensen (ATCG) kan utläsas ovanför topparna (om ”N” skulle finnas ovanför topparna betyder detta obestämd nukleotid och många N är ytterligare ett tecken på ”dålig sekvens”). Notera följande till labrapporten: S-­‐‑1: Hur lång är hela sekvensen (i bp)? S-­‐‑2: Mellan vilka bp (ungefär) finns det bra sekvensinformation? S-­‐‑3: Vilka regioner innehåller riktigt dålig sekvensinformation? För att ni ska kunna analysera sekvensen mot databaser på Internet måste sekvensinformationen först exporteras i sk. FASTA-­‐‑format, som är ett format som databaserna förstår och kan använda. I Chromas, välj: File -­‐‑> Export -­‐‑> Döp filen: ”Morf4l1 sequence grupp nr.txt”. Var noga med att ni exporterar filen i FASTA-­‐‑format, samt att ni anger ”.txt” som suffix i filnamnet. Öppna sedan er .txt-­‐‑fil och kopiera den till datorns minne (markera allt, CTRL+C). 39 Följ länken till Entrez sidan. Gör denna sida till ett bokmärke i webläsaren (Spara som ”Favorit”), eftersom ni kommer att behöva återvända hit ofta under labben. Entrez är en mycket användbar plats som samlar en mängd databaser genom vilka man kan utföra i princip all sorts biologisk och medicinsk bioinformatik. Välj BLAST-­‐‑ funktionen i översta högra hörnet (bild). En ny sida öppnas där man kan välja mellan olika sorters för BLAST-­‐‑sökningar (bild 2). Titta på sidan för att bekanta er med de olika alternativen. Bland annat kan man BLAST:a nukleotid-­‐‑sekvenser, aminosyra-­‐‑sekvenser men även låta datorn translatera exemplevis en DNA-­‐‑sekvens till aminosyror för att sedan söka efter liknande aminosyrasekvenser. Vi är intresserade av att jämföra vår DNA-­‐‑sekvens med annan DNA-­‐‑sekvens och väljer därför ”Nucleotide blast” (bild 2). En ny sida öppnas där vi ska fylla i vad vi vill undersöka och i vilken art (bild 3) 40 Börja med att klistra in Morf4l1-­‐‑sekvensen från datorns minne i ruta A (CTRL+V). Sedan måste vi välja vilken databas vi vill söka i (B). Titta på alternativen i drop-­‐‑ down menyn. Här kan man välja att söka i flera olika databaser, bl.a. i musens eller människans genom. Men vi är ju ute efter att undersöka kvalitén på vår ”hemma-­‐‑ klonade” kyckling Morf4l1-­‐‑sekvens genom att jämföra den med den redan publicerade kyckling-­‐‑sekvensen. Kycklingen har ingen egen genomisk databas i denna meny, så vi väljer istället ”nucleotide collection (nr/nt)” i menyn (B) och sedan organismen kyckling (=Gallus gallus) i ruta C. I ruta C, börja att skriva ”Gallus gallus” (med stort G i början) och så erbjuds ni flera olika alternativ under tiden in skriver. Välj ”Gallus gallus (taxid 9031)” från menyn. Om “taxid 9031” inte dyker upp automatiskt går det bra att söka utan att skriva det. I ruta D, välj ”Highly similar sequences”. Tryck sedan på BLAST-­‐‑knappen och vänta på att sökningen blir färdig. En ny sida öppnas när sökningen är klar. Titta noggrant på sidan och besvara följande till labrapporten: S-­‐‑4: Hur många träffar fick ni upp? Vilken var bäst? Vad heter den? S-­‐‑5: Hur många procent av sekvensen ni sökte med (Query) träffade någonting i databasen (lägg fokus på den bästa träffen)? S-­‐‑6: Vid vilka bp i er Query börjar respektive slutar träffen? S-­‐‑7: Vad är likheten (=identity) mellan Query och Sbjct (Sbjct = subject) i procent? S-­‐‑8: Hur många gaps (dvs. positioner ”utan par”) finns det? I vilken sekvens finns det gaps – i Query, Sbjct eller båda? S-­‐‑9: Vilket ”Accession number” har Sbjct-­‐‑sekvensen? Gå nu tillbaka till sidan som visas i bild 3 och välj istället ”Somewhat similar sequences” i fältet D. Svara sedan på följande frågor: S-­‐‑10: Hur många träffar får ni upp nu? Vilken träff var den bästa? S-­‐‑11: Spelar det någon roll vilket sökalternativ i D ni använder? S-­‐‑12: Vilket sökalternativ skulle ni föredra att använda själva? Motivera. Nu är denna del av labben klar och vi fortsätter med nästa moment. 41 Sekvensjämförelser med BLAST alignment Syftet är att jämföra hur lik eller olik kycklingens Morf4l1-­‐‑gen (mRNA/cDNA-­‐‑ sekvens) är jämfört med några andra organismers Morf4l1-­‐‑gener (mRNA/cDNA). Er uppgift är att jämföra kyckling Morf4l1 med fisk, mus och människa på både nukleotid-­‐‑nivå och aminosyra-­‐‑nivå. Utförande: Först vill vi hämta nukleotidsekvensen (cDNA) för Morf4l1 i kyckling. Börja med att gå tillbaka till Entrez-­‐‑ startsidan och sök på ”morf4l1”. Notera att ”morf4l1” ger en mängd träffar i flera olika databaser, allt från vetenskapliga artiklar (PubMed), till nukleotid-­‐‑ sekvenser (Nucleotide, EST, SNP, osv), till proteininformation (Protein, Structure, 3D-­‐‑domain, osv) till genomsekvenser (Genome, Gene, osv). Alla dessa träffar är för morf4l1 generellt och inkluderar därför information från en mängd olika arter. Vi vill söka specifikt för kycklingens Morf4l1, så därför begränsar vi vår sökning genom att lägga till ”gallus” som sökord och söka igen. Nu blev det betydligt färre träffar. Klicka på ” Nucleotide” för att gå till nukleotid-­‐‑sökresultaten (bild). Jämför de träffar ni fick upp genom att klicka på länkarna och se vilken sorts information de olika träffarna innehåller. Kolla framförallt på datumet då de lades in i databasen. Ta reda på vilken av träffarna som är inlagd i databasen mest nyligen och som samtidigt innehåller information om nukleotid-­‐‑sekvensen för Morf4l1. Det är det sökresultatet som ni vill använda. Ni upptäcker här att det finns två varianter av Morf4l1 och dessa är inlagda i databasen samma dag. Att det finns två transkriptionsvarianter beror på alternativ splicing som innebär att vissa exon i mRNA-­‐‑transkriptet klipps bort medan andra är intakta. Använd den långa varianten (variant 1) för de fortsatta övningarna i denna laboration. Notera ”Accession number” för den träffen: (tips: Accession number ska inledas med prefixet ”NM_” som står för ”mRNA transkript”; se vidare på webben: http://www.ncbi.nlm.nih.gov/RefSeq/key.html). Denna träff är den officiella kodande sekvensen för kycklingens Morf4l1. Sekvensen i detta datablad är alltså lika med sekvensen av de sammanfogade exonerna i det splicade Morf4l1-­‐‑mRNAt. Dessutom får ni via denna sida tillgång till den 42 translaterade aminosyra-­‐‑sekvensen för Morf4l1 samt flera användbara länkar och korsreferenser till andra databaser. Studera sökresultatet noga och svara på följande frågor: B-­‐‑1: Hur många bp består Morf4l1 mRNA (cDNA) av? B-­‐‑2: På vilken av kycklingens kromosomer ligger Morf4l1? B-­‐‑3: Vilken funktion har Morf4l1? Nu ska vi använda kycklingens Morf4l1 sekvens för att jämföra den mot andra arter. Först vill vi därför ”översätta” sekvensen till FASTA-­‐‑format så att vi kan använda den när vi ska BLAST:a senare. För att få fram FASTA sekvensen gör så här: när ni har fått era sökresultat och valt den långa versionen av kycklingens Morf4l1 via nucleotide (1) och mRNA fliken (2) klickar ni på FASTA knappen i nästa fönster (3). Sekvensen visas nu i FASTA-­‐‑format. Markera sekvensen, kopiera den och klistra sedan in den i ett nytt .txt-­‐‑dokument för att ha den tillgänglig senare. Nu ska vi börja jämföra den här sekvensen med musens Morf4l1. Gå tillbaka till Entrez-­‐‑ startsidan och sök efter ”Morf4l1 mus musculus”. Följ länken via ”Nucleotide” till sökresultatet och upprepa vad ni gjorde för att konvertera sekvensen till FASTA-­‐‑ format. När ni har både kycklingens och musens Morf4l1 sekvenser i FASTA-­‐‑format i var sitt .txt-­‐‑dokument är ni redo att jämföra hur lika/olika de är. B-­‐‑4: Notera ”Accession number” för musens Morf4l1: B-­‐‑5: Hur många bp består musens Morf4l1 mRNA (cDNA) av? Gå nu tillbaka till Entrez-­‐‑startsidan och välj BLAST funktionen i övre högra hörnet (som förut). En ny sida laddas upp och nu ska ni välja ”Align 2 sequences using BLAST” (bild). 43 En ny sida laddas upp (se bild nedan). Klistra in den ena FASTA-­‐‑sekvensen i ruta A och den andra FASTA-­‐‑sekvensen i ruta B och tryck sedan på ”BLAST” för att jämföra de båda sekvenserna. När den nya sidan laddas upp med resultatet ska ni svara på följande frågor: B-­‐‑6: Hur många % identitet uppvisar de båda sekvenserna mot varandra? B-­‐‑7: Hur lång sekvens (bp) är det som överlappar total sett? B-­‐‑8: Vid vilka baspar börjar respektive slutar överlappet mellan kycklingens sekvens och musens sekvens? Backa nu tillbaka i web-­‐‑läsaren så att ni kommer till fönstret med BLAST alternativ och klicka återigen på ”Align 2 sequences using BLAST” långt ner på den sidan. Testa nu att istället för FASTA skriva in de ”Accession number” ni har antecknat för kycklingens respektive musens Morf4l1-­‐‑sekvens i fält A respektive B. Tryck på ”Align”. Ni får upp samma resultat! Att använda Accession number istället för FASTA-­‐‑ formatet är betydligt enklare när man vill jämföra sekvenser som redan finns lagrade i olika databaser. Nu är dags att jämföra kycklingens Morf4l1-­‐‑sekvens med andra arters Morf4l1 sekvens för att ta reda på hur många procents likhet det är. Sök upp sekvenserna för de olika arterna genom att söka på ”morf4l1” och artnamnet på latin. Leta upp och anteckna Accession number för alla sekvenser och jämför dem sedan med 44 kycklingens Morf4l1 sekvens genom att använda ”Align 2 sequences using BLAST” funktionen precis som nyss . Nu vet ni hur ni gör, så det är bara att fylla i tabellen nedan (skall redovisas i labrapporten): ART Latinskt namn % likhet Antal bp Accession number (med kyckling) Kyckling Gallus gallus Zebrafisk Danio rerio Mus Mus musculus Människa Homo sapiens När ni är klara med tabellen ovan ska ni göra samma sak fast för Morf4l1 aminosyra-­‐‑ sekvensen! Den enda egentliga skillnaden är att ni istället för att gå in på ”Nucleotide” går ni in på ”Protein” när ni får upp era sökresultat i Entrez (bild). När ni gör alignment ska ni välja ”blastp” uppe till vänster. Fyll i tabellen över likheterna mellan aminosyra-­‐‑sekvensen för de olika arterna (skall redovisas i labrapporten). Tänk på att Accession number ska inledas med prefixet ”NP_”. ART Latinskt namn % likhet Antal aa Accession number (med kyckling) Kyckling Zebrafisk Mus Människa Gallus gallus Danio rerio Mus musculus Homo sapiens 45 Mall för labrapport med specifika frågor till laboration 9: Analys av sekvenseringsdata: 1. Redovisa svaren på de frågor som står i labhandledningen. 2. Hur stor del av er söksekvens (query) träffade något i databasen? Vad tror ni att den övriga sekvensen (som inte träffade något) består av? 3. Förklara varför gaps:en bara finns i en av sekvenserna? Vad säger det om kvalitén på sekvensinformationen tror ni – bra, OK eller dålig? Sekvensjämförelse mellan olika arter: 1. Redovisa svaren på de frågor som står i labhandledningen. 2. Redovisa tabellerna med nukleotid-­‐‑ och aminosyrasekvenser. 3. Vilken art/ vilka arter är mest lika/olika kycklingen med avseende på nukleotidsekvens? 4. Vilken art/ vilka arter är mest lika/ olika kycklingen med avseende på aminosyrasekvens? 5. Finns det en korrelation mellan likheten på nukleotidnivå och aminosyra-­‐‑ nivå (utgå ifrån era egna resultat)? 6. Varför skiljer sig likheterna mellan nukleotidsekvens och aminosyrasekvens åt? 46 10: Kvantitativ (q) Omvänd (R) Transkription(T) PCR (qRT PCR) Dag 1: • • • • Demonstration av qRT PCR Beräkning av mRNAuttryck Jämförelse och test av resultaten Databassökning med primerpar Teori Kvantitativ PCR (qPCR) är en vidareutveckling av PCR-­‐‑tekniken med skillnaden att man kan mäta mängden av den amplifierade produkten i reaktionen efter varje cykel. En vanlig användning av qPCR är att mäta mRNA nivåerna för gener och på så sätt få reda på hur mycket de uttrycks. Som med all PCR bygger tekniken på att man har primers som är specifika för det sekvensavsnitt man vill amplifiera. Man måste utgå från DNA som templat när man amplifierar sin PCR-­‐‑produkt. Eftersom vi har mRNA som vi vill mäta måste detta översättas till cDNA (complementary DNA), vilket sker med hjälp av enzymet reverse transcriptase. Man amplifierar sedan upp sin PCR-­‐‑produkt från cDNAt. Antalet cDNA-­‐‑kopior står i proportion till ursprungsnivån av mRNA-­‐‑kopior och därmed kommer även PCR-­‐‑produkten att göra det. I en vanlig PCR med genomiskt DNA som templat kan man använda primers som känner igen sekvens i både introner, exoner, promotorer eller egentligen var som helst i genomet. I qPCR måste man designa primers som sitter i exonerna på genen man vill studera. Ofta vill man se förändringar och man mäter skillnader mellan olika prover och inte den absoluta uttrycksnivån med qPCR. Man måste alltså ha minst två prover som man jämför med varandra. Vid en qPCR-­‐‑körning avläser maskinen hur mycket PCR-­‐‑ produkt som finns efter varje PCR-­‐‑cykel genom att mäta mängden fluorescens som finns i provet. Denna fluorescens kommer från SYBR-­‐‑green, en molekyl som fluorecserar då den binder till dubbelsträngat DNA. Ju mer PCR-­‐‑produkt som bildats i reaktionen, desto mer fluorecsens kan detekteras. Beroende på hur mycket cDNA som fanns från början kommer det ta ett visst antal cykler för reaktionen att uppnå tillräckligt mycket PCR-­‐‑produkt för att fluorescensen ska vara detekterbar. Ju mer en gen är uttryckt desto mer mRNA finns det, vilket i sin tur ger fler cDNA som nya PCR-­‐‑produkter kan amplifieras ifrån. Detta gör att vi fortare (med färre cykler) når en mätbar flourescens om det finns mycket mRNA. Ofta sätter man ett tröskelvärde som är tio gånger högre än bakgrundsfluorescensen, och mäter vid vilken cykel ett prov passerar denna nivå (Ct-­‐‑värde). Ct står för ”cycle threshold”. Genom att jämföra antalet cykler det tar för olika prover att nå Ct, får man fram relativa mRNA nivåer. Man jämför ex. skadad mot oskadad vävnad, olika tidpunkter under utvecklingen av ett embryo eller tumörvävnad mot frisk vävnad. 47 För att kompensera så att alla prover har lika mycket total RNA från början samt att cDNA-­‐‑syntesen kan fungera lite olika bra mäter man även en eller flera refensgener. Som referensgen används vanligtvis sk. ”house-­‐‑keeping”-­‐‑gener. En ”house-­‐‑ keeping”-­‐‑gen är en gen som finns i alla celler och som är nödvändig för alla cellers normala funktion. Dess mRNA nivå varierar lite eller inte alls mellan olika celler eller i tiden. Referensgenerna gör att man på ett säkert sätt kan jämföra sina prover. Särskilda instruktioner • • • Demostration av qRT PCR sker på forskningslaboratorium i A2:2. Uppgifter utförs i respektive laborationsgrupp i datasal efter demonstrationen. Till laborationen finns en animation som kan laddas ned på Studentportalen under länkar; under laborationer och seminarier finns en användbar excelfil som kan underlätta datalaborationen (filen heter lab10_kursen.xls) Laborationsrapport Resultaten skall presenteras muntligt på genregleringsseminariet och labrapporten skall lämnas in gruppvis. Bakgrund till laborationen I denna lab ska ni på genreglerings-­‐‑nivå studera vad som händer i näthinnan efter en skada som påminner om vad som sker vid ögonsjukdomen grön starr (glaukom). Försöket som gav dessa data såg ut på följade sätt: 48 Skadan gjordes på ena ögat hos vuxna råttor, därefter avlivades djuren och näthinnorna togs ut för analys. Som kontroll till de skadade djuren användes näthinnor från normala (ej skadade) djur. Syftet med försöket var att genom att jämföra de skadade djuren med de normala djuren förstå vilka gener och cellulära processer som ändras i näthinnan vid grön starr. RNA preparerades från näthinnorna och cDNA syntetiserades för att användas vid qPCR. I försöket hade man fyra olika primerpar som var specifika för de gener man ville studera. För att kompensera för att proverna kan ha innehållit olika mängd RNA användes en referensgen för att ”normalisera” respektive prov. I det här försöket användes en gen som kodar för ett ribosomalt protein som referensgen. Uppgiften Ni ska börja med att ta reda på vilka fyra testgener (A, B, C, D) som undersöktes i experimentet. Därefter ska ni göra beräkning av mRNA-­‐‑uttryck. Här nedan finns de rådata (Ct-­‐‑värden) som man fått fram vid en qPCR-­‐‑körning. Man mätte nivåerna av fyra stycken testgener (A, B, C, D) i prover från en normalgrupp (Prov 1-­‐‑4) och i prover från en skadegrupp (Prov 5-­‐‑8). Dessutom mättes nivåerna av en referensgen i dessa prover. Ni ska med hjälp av Ct-­‐‑värdena i tabellen beräkna om var och en av de fyra generna ändrat sig i de skadade proverna jämfört med de normala proverna. För att ta reda på om de två grupperna verkligen skiljer sig åt ska ni utföra ett statistiskt test av resultaten (Student’s t-­‐‑test). Era uppgifter är att: 1) Gör en databassökning i BLAST med primersekvenserna för att ta reda på vilka de fyra generna (A, B, C, D) är. Primrarna är designade mot råtta (Rattus norvegicus). 2) Beräkna det relativa genuttrycket av gen A, B, C, respektive D i proverna i normalgruppen och skadegruppen. 3) Beräkna gruppernas medelvärden och standadavvikelser samt gör ett t-­‐‑test för att undersöka om de två grupperna skiljer sig åt statistiskt sett. 4) Gör ett stapeldiagram som visar ert resultat. Databassökning med primersekvenser Ni ska börja med ta reda på i vilka transkript primerparen i tabellen binder in i. Gör på samma sätt som i Egenuppgiften med primerdesign (i Lab 7). Gå till: http://www.ncbi.nlm.nih.gov/sites/gquery. Välj BLAST uppe till höger. Välj sedan nucleotide blast. Skriv in primersekvenserna på varsin rad i den första rutan. Under ”Choose Search Set” Välj: Others Eftersom vi mäter mängden mRNA (omvandlat till cDNA) väljer ni: Reference mRNA sequences (refseq_rna) Vävnaden kommer från råtta; välj: Rattus norvegicus (taxid: 10116) (Optimize for: Highly similar sequences) Klicka sedan på BLAST längst ner till vänster för att starta sökningen. 49 När sökningen är klar får ni upp en sida med träffar i databasen. Högst upp finns träffar med mest likhet till de primers ni matat in. Om ni scrollar ner på sidan ser ni hur många av baserna i varje primer som överensstämmer med sekvensen i databasen och om det finns något glapp (gaps). Ni kan ta reda på mer om varje gen genom att klicka på Gene som finns på höger sida under Related information. På den nya sidan finner ni information om vad proteinet som genen kodar för gör och ev. vilka andra proteiner det binder till (Gene Ontology) samt om genen har flera namn (Also know as). Ta reda på informationen som ni behöver till labrapporten och genregleringsseminariet! Primersekvenser för testgener Gen A B C D Forward primer GCCACCAGTAACATGCAAGAAA AGCCCCGAATCCTGTAGAGAG CAGTACAGTGGCGGATATGGTG CTTCAACACCCCAGCCATG Reverse primer CGGGAAGCAACGTCTGTGA CGTGGTGCAGTATGAGTTCCA TGCCGCGGGTCACTACA GTGGTACGACCAGAGGCATACA Gennamn Gen A = _________________________ Alternativt namn __________________________________ Gen B = _________________________ __________________________________ 50 Gen C = _________________________ __________________________________ Gen D = _________________________ __________________________________ Att normalisera och beräkna mRNA-uttrycksnivåer från en qPCR För att få fram det relativa genuttrycket måste vi först normalisera testgenens Ct-­‐‑ värde mot Ct-­‐‑värdet för referensgenen för samma prov. Därefter måste man ta hänsyn till att man optimalt får en fördubbling av PCR-­‐‑produkten vid varje PCR-­‐‑ cykel. Det dubbelsträngade DNAt separeras och på varje enkelsträngad DNA-­‐‑ molekyl syntetiseras en ny sträng. Detta gör att man måste beräkna sina värden i 2-­‐‑ logaritm (en cykel=21=2, (2 upphöjt till x skrivs som 2^x), två cykler=2^2=4 o.s.v.). I vårt fall får vi i stället färre cykler om vi har mer av ursprungs cDNAt och vi har därför ett omvänt proportionerligt förhållande (d.v.s. 1/2^Ct eller som man också kan skriva det: 2^-­‐‑Ct). Skillnaden mellan testgenen och referensgenen är Ct-­‐‑testgen – Ct-­‐‑ referensgen vilket ger oss vårt normaliserade värde =2^-­‐‑(Ct-­‐‑gen – Ct-­‐‑referensgen). Ex. Normaliserat värde för Prov 1 i gen A: Ct-­‐‑gen – Ct-­‐‑referensgen: 28,84 -­‐‑18,42 = 10,42 2^-­‐‑(Ct-­‐‑gen – Ct-­‐‑referensgen): 2^-­‐‑10,42 = 7,299 x 10^-­‐‑4 Beräkna separat för varje gen (A, B, C, D): Beräkna med denna formel; 2^-­‐‑(Ct-­‐‑gen – Ct-­‐‑referensgen); fram ett normaliserat värde för varje prov. Ni beräknar det enklast i datorn med beräkningsprogrammet MS Excel. För att kunna tolka era data ska ni sedan relatera alla normaliserade värden för gruppen med skada till normalgruppens medelvärde. Ni får då relativa värden som visar hur mycket den skadade gruppens prov skiljer sig från normalt. Den beräkningen görs genom att dividera det normaliserade värdet för varje prov med medelvärdet av normalgruppen. Beräknar ni sedan medelvärden av grupperna kommer normalgruppen att ha medelvärdet 1 och skadegruppen ett medelvärde som visar förhållandet till normal dvs till 1 (ex. 5,2 säger att genen uttrycks 5,2 ggr mer i skadeproverna än i normalproverna eller 520%). Ct-värden för de olika generna Prov Referensgen A B C D Normala 1 2 3 4 18,42 18,33 18,26 18,33 28,84 27,31 27,78 28,3 26,63 26,31 25,99 25,81 27,83 27,31 27,99 27,81 18,98 19,31 19,19 19,2 Skadade 5 6 18,67 18,62 24,93 25,29 24,35 24,68 27,35 27,68 19,35 18,88 51 7 8 18,58 18,7 24,92 24,33 25,22 25,53 28,22 27,83 19,22 19,95 Skiljer sig skadegruppen åt från normalgruppen? Gör ett statistiskt test! Om skade- och normalgruppens medelvärden ligger nära varandra eller om de olika proverna inom varje grupp varierar mycket, kan det vara svårt att bedöma om grupperna verkligen skiljer sig åt. Man kan då göra ett enkelt statistiskt test, tex ett “Student’s, t-test”. Genom att beräkna medelvärdet och standardavvikelsen och genom att man vet hur många prover det finns i varje grupp så kan man relativt enkelt testa om medelvärdena skiljer sig statistiskt åt. Man kan beräkna standardavvikelsen med hjälp miniräknare, resurser på nätet eller med hjälp ett beräkningsprogram. Samma sak gäller t-testet. Beräkna standardavvikelsen i Excel: Skriv in de relativa värdena för respektive gen i två kolumner i Excel. Markera en tom cell och skriv: =STDEV(markera ena kolumnen på arbetsbladet) Tryck på Enter. I rutan finns sedan standardavvikelsen inom kolumnen/gruppen. standardavvikelse=√((1/(n-1))∑(Xj-Xmedel)2) OBS! Om ni arbetar med en svensk version av excel skriver man =STDAV istället för =STDEV Gör t-­‐‑test i Excel: Genom att göra ett t-­‐‑test får man får fram ett p-­‐‑värde. p-­‐‑värdet är ett mått på sannolikheten att två grupper skiljer sig åt och beror av medelvärdet, standardavvikelsen (SD; standard devation) och antalet prov i respektive grupp. Man brukar säga att man tillåter en risk att man bedömt grupperna fel som är mindre än 5 %, dvs 0,05. Om p vid testet <0.05 brukar man anse att två grupper är statistiskt sett skiljda åt. Välj Formulas, sedan Insert Function. Under Insert Function: sök efter ”ttest” och välj detta genom att klicka OK. Markera värdena för normalgruppen i Array 1 och markera värdena för skadegruppen i Array 2. Skriv i rutan Tails: 2, och Type: 2. Klicka på OK. I rutan finns sedan p-­‐‑värdet som säger sannolikheten för att de två grupperna är olika. Mall för labrapport med specifika frågor till laboration 10: 1. Vilka var generna som studerades? Har dessa gener något ytterligare namn? 2. Visa ett exempel för hur ni räknade fram de normaliserade värdena och de relativa nivåerna mellan normal-­‐‑ och skade-­‐‑gruppen. Ange de relativa värdena i tabellform för varje gen (A, B, C, D). 3. Ange medelvärdet och standardavvikelsen för de relativa nivåerna i mellan provgrupperna. Ange för varje gen (A, B, C, D). 4. Visa ett stapeldiagram över genuttrycket i grupperna, rita även ut standardavvikelsen som ni räknat ut. Gör ett diagram för varje gen (A, B, C, D). 52 5. Skiljer sig normal-­‐‑ och skade-­‐‑grupperna åt statistiskt sett? Vilket p-­‐‑värde fick ni i t-­‐‑testet? Ange för varje gen (A, B, C, D). 6. Vid en qPCR körning med ett primerpar för genen TATA binding protein kom Ct-­‐‑värden upp för såväl de positiva proverna (där cDNA tillsatts) som för de negativa kontrollerna dvs där inget cDNA tillsatts. Ct-­‐‑värdet låg på mellan 36,65 och 37,14 för negativa kontrollerna medan de övriga Ct-­‐‑värdena (för de prover där cDNA tillsatts) låg på mellan 22,63 och 22,78 (se tabell nedan). Vad tyder Ct-­‐‑värdena för dessa negativa kontroller på? Var resultatet från denna qPCR körning pålitligt? 7. Vid en annan qPCR körning med ett primerpar för genen Retinoblastoma blev Ct värderna på de positiva proverna väldigt höga. De postiva proverna (med cDNA) hade nästan samma värde som de negativa. Ct-­‐‑värderna för de positiva var mellan 35.17 och 37.65 medan Ct-­‐‑värderna för de negativa låg mellan 36.74 pch 39.55 (se tabell nedan). Vad kan anledning-­‐‑en/arna vara till sådana resultat? Hur ska du gå vidare för att verifiera qPCR körningen? Positiv kontroll, cDNA tillsatt Positiv kontroll, cDNA tillsatt Positiv kontroll, cDNA tillsatt Negativ kontroll, inget cDNA tillsatt Negativ kontroll, inget cDNA tillsatt Negativ kontroll, inget cDNA tillsatt Prov 1 2 3 4 5 6 Referensgen 17.53 17.68 18.24 N/A N/A N/A Retinoblastoma 35.17 37.11 37.65 36.74 37.49 39.55 Förbered er för att (kortfattat, ca 5 min) presentera era resultat på genreglerings-­‐‑ seminariet. Gör bl.a. over-­‐‑head kopior på era grafer. Positiv kontroll, cDNA tillsatt Positiv kontroll, cDNA tillsatt Positiv kontroll, cDNA tillsatt Negativ kontroll, inget cDNA tillsatt Negativ kontroll, inget cDNA tillsatt Negativ kontroll, inget cDNA tillsatt Prov 1 2 3 4 5 6 53 Referensgen TATA bindning protein 18,42 22,63 18,33 22,78 18,26 22,69 N/A 36,65 N/A 37,05 N/A 37,14